서 론
식품안전사고의 배경으로는 생물학적, 화학적, 물리적 요 인이 있으나, 식중독균에 의한 발생률이 가장 큰 부분을 차 지하고 있다. 따라서, 식품산업현장에서는 식중독균으로부 터의 안전성을 확보하기 위해서 HACCP (hazard analysis and critical control point)과 같은 제도적 장치를 마련하여 정부차원에서 노력을 하고 있다. 이러한 제도적 장치의 마 련과 위생에 대한 관심의 증가에도 불구하고 최근 국내 식 중독 사고의 발생은 증가하고 있으며, 이는 비단 여름철에 국한되지 않고 점차 사계절로 확대되고 있다. 특히, 근래에 발병하는 식중독은 주로 집단 감염 형태로 나타나며 단체 급식을 시행하는 초·중·고등학교에서 높은 발생률을 보이 고 있다(Jo et al., 2009).
이러한 경향들로 인해, 식품산업분야에서는 미생물 검출 에 높은 관심을 보이고 있으며, 검출 대상 미생물로는 Salmonella species, Escherichia coli, Listeria species가 가 장 활발히 연구되어지고 있다(Ha, 2007; Liébana et al., 2016). 그 결과 식중독균 검출기술의 수준이 최근 눈부시 게 발전하고 있음에도 불구하고 실제 식품 시료로부터 미 생물을 검출하는 방법은 아직까지 미생물 배양 및 콜로니 카운팅 방법에 의존하고 있으며 이는 신속검출에 있어서 주요 걸림돌로 작용하고 있다(Kim et al., 2010). 따라서, 가공된 식품으로부터 미생물을 신속하고 정확하게 검출하 기 위해서는 식품 시료에서 물리적 또는 화학적 방법으로 미생물을 효과적으로 분리·농축하는 단계가 선행되어야 한 다(Kim & Ha, 2006). 분리된 미생물을 특이적으로 검출하 기 위해서는 선택도가 높은 핵산증폭기술(PCR) 기반의 기 술들이 활용되고 있다(Lim et al., 2016).
식품 시료로부터 분리·농축하는 과정을 거친 미생물로부 터 핵산을 추출하는 과정이 뒤이어 수행되어야 핵산증폭기 술 기반의 검정법이 효과적으로 수행될 수 있다(Lim et al., 2017). 따라서, 핵산증폭기술을 이용한 미생물 검출방 법의 시행에 있어서 미생물로부터 핵산을 정제·농축하는 단계 역시 중요하다. 특히, 실험실 기반의 미생물 검출이 아닌 현장진단형 검출을 위해서는 분리된 미생물로부터 핵 산을 분리하여 고농도로 농축하는 단계가 필요하며 이를 통해 보다 감도 높은 검출이 이뤄질 수 있다. 핵산을 정제 하고 농축하는 기술은 액상을 기반으로 한 기술에서 발전 하였으며, 최근에는 고체상을 기반으로 한 기술에 대해서 연구가 활발히 진행되고 있다(Reedy et al., 2011).
따라서 본고에서는 시료로부터 미생물을 분리·농축하는 기술과 분리된 미생물로부터 핵산을 정제·농축하는 기술의 최신 동향에 대해서 알아보고, 핵산 증폭 기술 기반의 식 중독균 검출 기술에 대한 사례를 식품산업에의 응용 관점 에서 살펴보고자 한다.
본 론
대부분의 신속 검출 기술에 있어서, 시료로부터 미생물 을 분리(separation), 농축(concentration), 정제(purification) 할 수 있다면 검출 기술의 응용 분야는 더욱 확대될 수 있을 것이다. 특히, 시료에 존재하는 성분들에 의해서 검출 민감도가 저해되는 현상이 주로 발생하기 때문에 시료로부 터 미생물을 분리해내는 과정은 필수적이라 할 수 있다. 더불어 시료에 저농도로 존재하는 미생물을 분리하고 농축 하는 과정을 거친다면, 저농도의 타겟 미생물을 다양한 검 출 기술을 이용해 검출할 수 있게 된다. 미생물을 시료로 부터 분리하는 방법은 크게 immunomagnetic separation (IMS) 등과 같은 선택적 분리방법과 centrifugation 등과 같 은 비선택적 분리방법이 있다(Stevens & Jaykus, 2004).
균일한 크기의 마이크로 입자와 초상자성(superparamagnetic) 나노입자의 접목으로 자기장(magnetic field)에 의해서 마이크로 입자를 제어할 수 있는 기술이 개발되었 으며 이를 응용한 연구들도 활발히 진행되었다(Berensmeier, 2006). 타겟 미생물과 특이적으로 결합하는 항체를 초상자성 입자의 표면에 기능화하여 제작한 면역친화성 (immunoaffinity) 마이크로입자를 이용하여 미생물을 시료 로부터 컬럼 혹은 원심분리 없이 분리하는 방법이 고안되 었다. 최근에는 이런 면역친화성 기반의 미생물 분리 기술 을 immunomagnetic separation (IMS) 기술이라고 지칭하 고 있다(Skjerve et al., 1990).
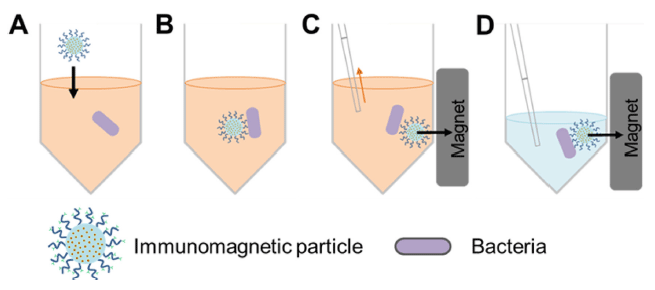
이 시스템의 작동 기작은 다음과 같다. 먼저, 자성 마이 크로 입자의 표면에 타겟 미생물과 결합하는 항체들을 고 정화시켜 immunomagnetic particle을 준비한다. 그리고 타 겟 미생물이 포함된 시료 용액에 immunomagnetic particle 을 첨가하여, 항원-항체 반응을 통해 immunomagnetic particle과 타겟 미생물간의 결합을 유도한다. 그 후, 자석 을 이용해 타겟 미생물과 결합한 마이크로 입자를 포집하 고, 시료 용액을 제거한 후 적절한 용액을 첨가하여 분리 하게 되는 것이다(Fig. 1). 이런 원리를 적용하여 식품으로 부터 살모넬라균을 분리한 결과가 보고되었으며(Cudjoe et al., 1995; Hyeon & Deng, 2017) 또한 탄저균(Bacillus anthracis)의 포자를 분리한 연구도 보고 되었다(Gatto- Menking et al., 1995; Waller et al., 2016). 더불어, 식품으 로부터 증균배양한 시료에서 E. coli O157:H7을 분리한 연 구 결과도 보고되었다(Lim et al., 2017).
고체상의 표면이 가지는 화학적 특성을 조절하여 용액 내에 존재하는 물질들을 비특이적 혹은 가역적으로 결합시 켜 분리하는 연구들이 진행되었다. 용액 내에 존재하는 물 질과 고체상 표면과의 결합은 반데르발스힘(Van der Waal’s force), 정전기적 상호작용(electrostatic interactions), 소수성 상호작용(hydrophobic interaction)과 수소 결합 (hydrogen bonding) 등이 있다(Glantz et al., 1999). 식품에 존재하는 미생물은 식품내의 성분들과 결합을 한 상태로 존재하게 되는데, 이 때 식품내 성분과의 결합을 주도하는 성분은 미생물의 세포벽 성분인 테코산(teichoic acids), 단 백질, 그리고 탄수화물 성분들이다(Meylheuc et al., 2002). 따라서, 식품으로부터 미생물을 분리하기 위해서는 식품성 분들과의 결합력을 낮춰 식품으로부터 분리가 용이하도록 해야 한다.
대부분의 미생물들은 pH 5.0 이상의 조건에서 순전하 (net charge)가 음을 나타낸다. 음전하를 띄는 미생물은 양 전하를 띄는 식품 내 성분들과 결합할 수 있다. 이런 현상 을 이용하여 양의 표면전하를 가진 이온 교환 수지(ion exchange resin) 등을 이용하여 미생물을 시료로부터 분리할 수 있다(Fig. 2) (Payne & Kroll, 1991). 분리된 미생물을 이온 교환 수지로부터 분리하여 농축시키기 위해서는 간단 히 용액의 산도를 증가시켜주면 되고, 미생물의 생존이 중 요하지 않은 경우는 산도를 더욱 높여 회수율을 증가시킬 수 있다(Lindahl & Bakken, 1995).
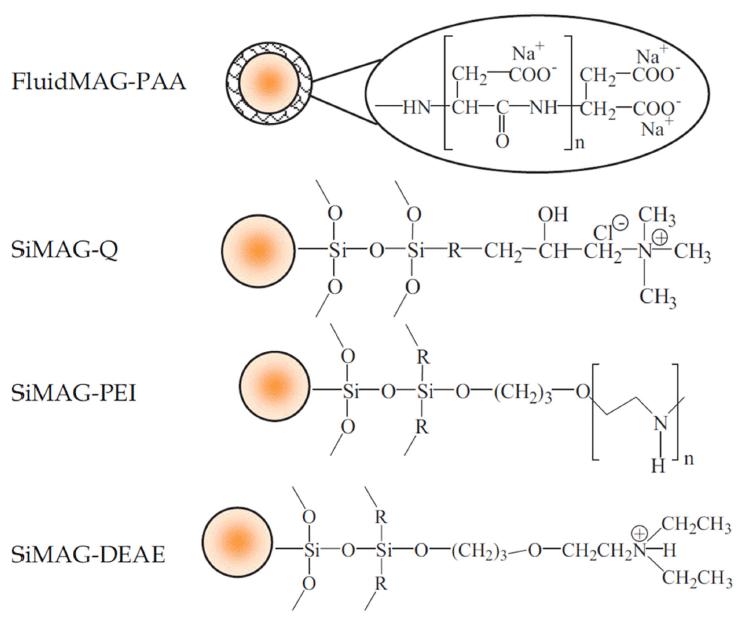
이온 교환 수지와 더불어 미생물 분리·농축 기술에 사용 중인 물질로는 금속 수산화물(metal hydroxides)이 있다. 금속 수산화물의 표면에 존재하는 수산화기(hydroxyl group)와 미생물 세포벽에 존재하는 아미노산과의 결합을 유도하고 추가적인 원심분리를 통해 시료로부터 미생물을 분리할 수 있게 된다(Dwivedi & Jaykus, 2011). 탈지분유 에 존재하는 Listeria monocytogenes와 Salmonella enterica 균을 금속 수산화물을 이용하여 분리·농축한 연구가 발표 되었다(Lucore et al., 2000).
액상에 존재하는 미생물은 물리적인 방법으로 분리할 수 있다. 그 방법에는 원심분리법(centrifugation)과 여과법 (filtration) 등이 있다. 그 중에서 원심분리법은 원심력을 이 용하여 액상에 부유하는 물질들을 침전시키는 방법을 말하 며, 미생물 분리에 사용되는 원심분리법의 종류로는 고속 원심분리법(high-speed centrifugation), 분별 원심분리법 (differential centrifugation)과 밀도구배 원심분리법(density gradient centrifugation)이 있다(Hopkins et al., 1991). 배지 와 같은 단순한 성상의 액상 시료에서 미생물을 분리하기 위해서는 단순히 원심력만을 인가하였다. 이를 적용하여 배지 혹은 액상의 시료에서 Clostriduim welchii를 분리한 연구가 보고되었다(Darby et al., 1970). 분별 원심분리법은 액상에 존재하는 물질의 크기와 밀도에 따른 침강률에 의 해 물질을 분리하는 방법으로, 원심분리 속도를 점차 증가 시켜 세포를 크기에 따라 분리하는 것을 기본으로 한다 (Catsimpoolas, 1977). 치즈로부터 E. coli O157:H7을 분리 함에 있어, 분별 원심분리법에 의해 지방을 먼저 분리하고 미생물을 분리한 연구가 보고되었으며(Meyer et al., 1991), 우유에서 Campylobacter를 지방과 물로부터 분리한 연구가 보고되었다(Wegmüller et al., 1993). 밀도구배 원심분리법 은 원심분리관의 기저부에서 상부로 갈수록 농도가 낮아지 는 밀도 구배를 형성하고, 시료를 주입하여 원심분리를 시 행하였을 때 미생물 혹은 용액내 존재하는 기타 물질들이 가지는 밀도와 동일한 밀도 구배층으로 이동하여 밴드를 형성하는 원리를 이용한 방법이다(Catsimpoolas, 2012). 이 방법으로, 치즈와 우유로부터 Shigella flexneri를 분리한 연 구가 보고되었으며(Lindqvist et al., 1997), 간 쇠고기로부 터 E. coli O157:H7을 분리한 연구가 보고되었다(Uyttendaele et al., 1999).
유전영동(DEP, dielectrophoresis)은 일정하지 않은 전기 장내에서 입자의 움직임을 제어하는 것을 말한다(Gascoyne et al., 1992). 유전영동은 고주파 전기장에 의해서 용액 내 에 존재하는 미생물을 전극으로 당길 수 있다. 특히, 용액 에 다른 종류의 미생물이 혼재하는 경우 각각의 미생물이 가지는 물리적, 화학적 특성에 따라서 분리되어질 수 있다 (Coakley, 1997; d'Amico et al., 2017). Markx 등에 의하면, 액체 배지에서 배양된 Bacillus subtilus와 Micrococcus lutes 를 유전영동에 의해서 분리한 연구결과가 보고되었다(Markx et al., 1996). 하지만, 실제 식품시료로부터 유전영동에 의 해서 미생물을 분리하기 위해서는 전도도가 낮은 액상의 시료가 준비되어야 하는 단점이 있다.
식품으로부터 분리·농축된 미생물을 검출하기 위해서는 항원-항체 반응 기반의 면역적 검사법이 이용될 수 있다. 하지만, 항원-항체 기반의 면역적 검사법은 반응용액 조성 에 따라 반응이 제한을 받을 수 있기 때문에 검출 민감도 및 선택도가 상대적으로 높은 핵산 기반의 검출법이 이용 된다. 핵산 기반의 검출법을 이용하기 위해서는 분리·농축 된 미생물로부터 핵산을 추출해야 하며 추출된 핵산을 정 제·농축하는 과정이 필요하다. 핵산의 정제·농축은 액상을 기반으로 하는 기술에서 시작하여 고체상을 기반으로 한 기 술로 발전하였다(Cady et al., 2003). 고체상 기반의 핵산 분 리·농축 기술이 발전하였지만, 핵산증폭에 있어서 제한요소 로 알려진 카오트로픽 염(chaotropic salt)을 핵산증폭 과정 이전에 제거해야 하는 과정이 필수적이다(Min et al., 2011).
1900년 초기부터 세포로부터 핵산을 분리하기 위한 방법 들이 다양하게 연구되어져 왔으며, 이들 중 페놀-클로로포 름(phenol-chloroform) 침전법이 가장 일반적으로 사용되어 왔다. 페놀과 클로로포름을 이용하여 핵산을 세포로부터 분리하고 에탄올을 처리하여 핵산을 침전시킴으로써 핵산 을 농축할 수 있다(Kado & Liu, 1981). 하지만, 페놀-클로 로포름 기반의 핵산 정제·농축 방법은 시료의 부피가 커 야하며, 사용자의 정교한 기술 습득이 요구되고 시간이 많 이 걸린다는 단점이 있다. 또한, 유기용매 사용에 의한 오 염 사고의 우려가 있다(Leff et al., 1995).
핵산 정제·농축기술은 Table 1에서 정리된 기술들로 나 뉘어 설명될 수 있다. 먼저, 방향족 그룹(aromatic group)으 로 표면을 기능화시킨 고체상 소재를 사용하여 액상에 존 재하는 핵산을 정제 및 농축하는 방법에 대한 연구가 선행 되었다(McCormick, 1989). 하지만, 고체상에서 핵산을 정 제·농축하는 기술의 원천 핵심기술로 인식되고 있는 것은 카오트로픽 염(chaotropic salt)를 이용한 핵산 정제 방법이 다. 이 방법은 Boom 등에 의해 고안되었으며, 카오트로픽 염에 의해 물분자간의 네트워크가 붕괴되는 현상을 이용한 기술이다(Boom et al., 1990). 물분자 네트워크 붕괴로 인 해 고체상의 표면에 존재하는 물은 제거되고, 실리카의 하 이드록실 그룹과 용액내의 양이온 분자가 치환됨으로써 고 체상은 양극화된다. 결과론적으로, 이를 통해 음극을 띄는 핵산이 양극화된 고체상의 표면에 흡착이 가능하게 된다 (Min et al., 2011).
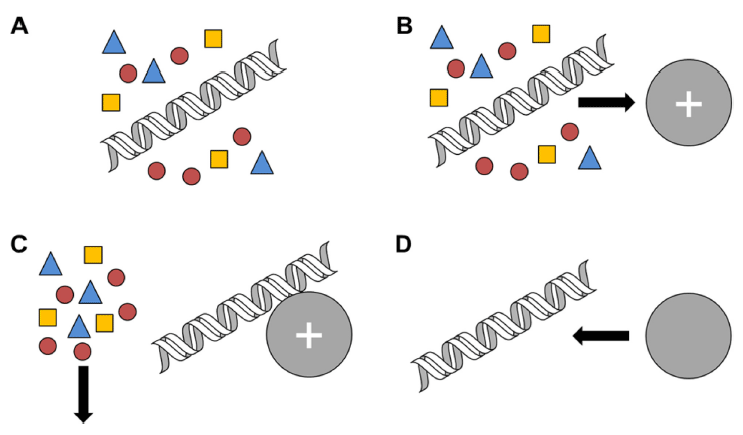
Charge Switch Technolohy (CST)는 유기용매나 세척을 필요로 하는 PEG (polyethylene glycol) 및 염을 사용하지 않으면서 핵산의 흡착 효율을 증대시킨 기술로, 고체상 표 면의 극성을 용액의 pH를 이용하여 개질한 마이크로 입자 를 사용하는 방식이다(Sohni et al., 2008). 이 기술은 다른 여타 물질을 사용하지 않으며, 용액의 pH 조절에 의한 고 체상 표면의 극성 조절에 의해서만 핵산을 흡탈착 시킨다 (Fig. 3) (Lee et al., 2008). 마이크로 입자의 표면에 코팅 되는 물질은 주로 pyrimidine 혹은 imidazole과 같은 방향 족 아로마틱 화합물이 이용된다.
Solid Phase Reversible Immobilization (SPRI)은 고체상 표면에 핵산을 흡탈착 시키기 위해서 카오트로픽 염 대신 일반염과 PEG을 사용한다(de Angelis et al., 1995). SPRI 에서는 카복실기로 표면이 개질되어 있는 고체상 소재를 사용하기 때문에 낮은 pH에서는 표면이 중성의 성질을 갖 고 높은 pH에서는 음극화 되는 현상을 가지게 된다. 이러 한 고체상 소재의 표면전하를 조절하여 핵산을 고체상 표 면에 흡착시키거나 탈리시킬 수 있다(Xu et al., 2003).
Cation Complexation Technology (CCT)는 사질 점토 (sand-clay)를 적용한 필터를 이용하며 흡착된 핵산을 방출 할 때 EDTA를 사용하는 특징이 있다. 핵산을 흡착하는 방식은 극성 작용이 아니라 Mg2+과 같은 2가 양이온을 가 교로 하여 음전하를 띠는 핵산간의 복합체를 유도하는 방 식이다(Hoopes & McClure, 1981). 고체상 표면에 흡착된 핵산의 방출은 핵산과 고체상 표면 간 존재하는 Mg2+과 같은 2가 양이온결합을 제거할 수 있는 킬레이트 물질인 EDTA를 이용한다.
핵산 증폭 기술은 분자생물학과 재조합 DNA 기술에 있 어서 가장 유용하게 사용되는 기술로써, PCR (polymerase chain reaction) 기술의 보급으로 그 응용분야가 확대되고 있다(Gill & Ghaemi, 2008). PCR 기술을 이용한 DNA 증 폭은 감염형 질병의 확인, 유전적 결합과 같은 목적으로 활용되고 있다(Foglietta et al., 1996). 더불어, 핵산 증폭 기술은 박테리아 및 바이러스의 검출에 있어서 높은 특이 성과 선택성을 제공하기 때문에 병원성균 검출에 있어서 강력한 기술로 이용되고 있다(Belgrader et al., 1999; Mao et al., 2016). 따라서, 핵산 증폭 기술로 개발되고 상업화되 어 있는 PCR, RCA (rolling circle amplification), LCR (ligase chain reaction), NASBA (nucleic acid sequence based amplification)와 LAMP (loop-mediated isothermal amplification) 기술들에 대해서 알아보도록 하겠다(Table 2).
Mullis 등에 의해 1980년대에 개발된 중합효소 연쇄 반 응(PCR, polymerase chain reaction)은 유전물질을 조작하 여 유전학적 연구와 생물학 분야 연구에서 다양하게 응용 되고 있다(Saiki et al., 1988). 중합효소 연쇄 반응으로 DNA의 특정 부위만 증폭할 수 있다. 이를 위해서 특정 염기서열을 인식하는 두 개의 짧은 oligonucleotide가 DNA 의 이중나선과 각각 혼성화 되어야 한다. 그리고 DNA 중 합효소의 작용으로 주형 DNA에 상보적인 염기서열을 갖는 단일가닥 DNA를 합성하여 두 가닥의 DNA로 합성하게 된 다. PCR은 열변성 과정, 결합과정, 중합 반응 과정의 3단 계를 반복적으로 수행함으로써 주형 DNA의 특정 부위를 2n 배로 증폭하게 된다. 또한, PCR은 DNA 주형의 증폭에 만 제한되는 것이 아니라 RNA를 주형으로 하여 역전사효 소를 이용한 역전사 반응 시행으로 cDNA (Complementary DNA)를 합성하는 역전사중합효소 연쇄 반응(RT-PCR, reverse-transcriptase polymerase chain reaction)으로도 활용 된다(Lanciotti et al., 1992).
실시간 중합효소 연쇄 반응(real-time polymerase chain reaction)은 PCR 기반의 실험방법으로, 실시간으로 표적 DNA의 증폭 및 증폭산물의 양을 측정할 수 있다(Heid et al., 1996; Vuong et al., 2016). 증폭된 DNA 산물을 정량 적으로 확인하기 위해서는 형광물질이 일반적으로 사용된 다. 증폭된 핵산의 정량을 위해 사용되는 형광물질은 크게 두 가지로 나눠질 수 있다. 먼저, 증폭된 DNA의 이중나선 구조에 결합하여 발광하는 SYBR Green이 이용될 수 있고 특정 DNA의 염기서열을 인식하는 탐침에 발광체를 표지 하여 이용할 수 있다. 증폭된 핵산 산물을 특이적으로 정 량하기 위해서는 발광체를 표지한 탐침을 이용하는 것이 일반적인 방법이다. 또한, real-time PCR은 역전사 중합효 소 연쇄반응과 결합하여 mRNA를 정량적으로 분석하는 방법으로 이용된다(Pfaffl, 2001).
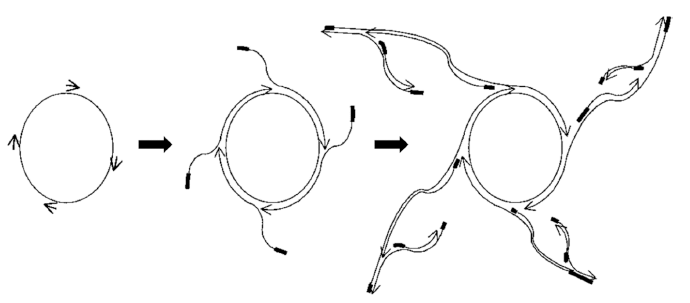
RCA (rolling circle amplification) 방법은 PCR법과 달리 항온에서 증폭이 이루어지는 단일 온도 핵산 증폭 기술이 다(Lizardi et al., 1998; Jiang et al., 2017). RCA는 한 개 또는 여러개의 primer를 사용하여 환형(circular)의 DNA를 효과적으로 증폭하는 기술이다. 타겟 유전자의 특정부위에 결합한 primer로부터 타겟유전자와 상보적인 DNA 가닥이 중합 되고, 주형 DNA가 환형이기 때문에 중합 되는 DNA 가닥의 최전방은 초기 primer가 혼성화 되었던 위치에 이 르게 된다. DNA 중합효소는 기존에 존재하는 DNA 가닥 을 주형 DNA로부터 분리(displacement)를 시키면서 계속 해서 중합반응을 이어 나간다. RCA 방법은 항온에서 증폭 이 이루어 지고 이중가닥의 DNA를 단일가닥의 DNA로 만드는 특징을 가지고 있는 중합효소를 일반적으로 이용된 다. 그 중에서 phi29 중합효소는 displacement 활성이 좋기 때문에 RCA 방법에 널리 이용되고 있다(Dean et al., 2001). RCA 방법에 의한 핵산의 증폭은 PCR법과 달리 다 중반응에 의해 일어나기 때문에 표적 핵산의 양이 작을 때 유용한 핵산 증폭 방법이다(Fig. 4).
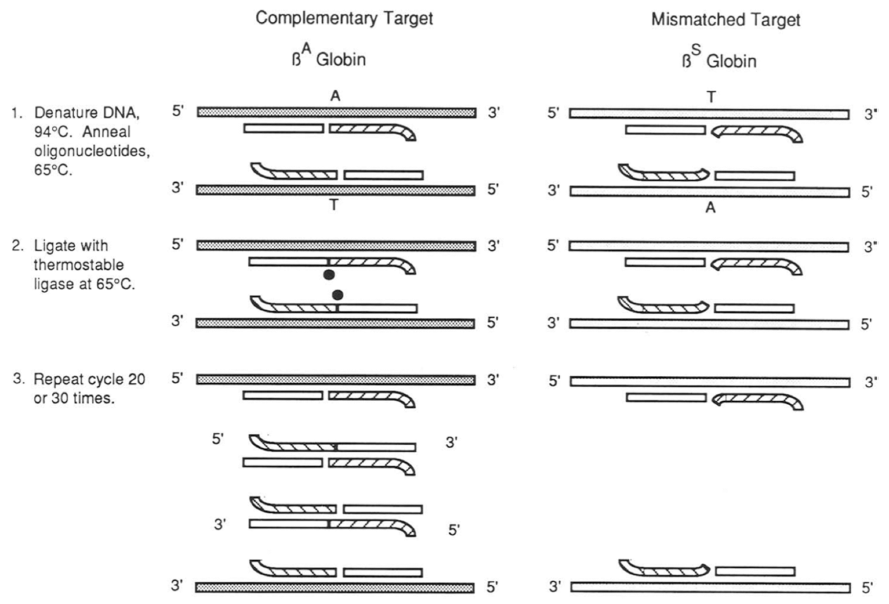
LCR (ligase chain reaction) 방법은 PCR법과 달리 primer 증폭 기술로 분류되고 있는 핵산 증폭 기술이다(Barany, 1991). 이 기술의 핵심은 열에 안정한 ligase를 사용하는 것으로 주형 DNA를 증폭하고 단일 염기서열 변이(single base mutation)을 확인할 수 있다(Fig. 5). PCR은 주형 DNA 와 혼성화된 primer를 기점으로 중합효소에 의해서 핵산이 증폭되는 방식이지만, LCR은 각 primer 사이 단일 염기서 열의 특성에 의하여 primer ligation이 진행되기 때문에 단 일 염기서열의 차이를 확인하고자 할 때 일반적으로 사용 되는 기술이다(Wiedmann et al., 1994; Gibriel & Adel, 2017). 하지만 ligase에 의존하기 때문에 핵산 증폭 속도가 느리고, 증폭된 핵산의 길이가 사용된 시발체와 차이가 크 지 않기 때문에 증폭 산물을 확인하는 단계가 반드시 필요 하다.
NASBA (nucleic acid sequence based amplification)는 등온에서 RNA를 증폭할 수 있는 기술로 바이러스 및 살 아있는 미생물 검출에 유용한 방법이다(Kievits et al., 1991; Clancy et al., 2016). 이 시스템의 특징은 사용되는 primer의 염기서열 중에 T7 RNA 중합효소가 결합하여 반 응할 수 있는 T7 promotor 염기서열을 가지고 있다(Leone et al., 1998). NASBA는 두 개의 primer를 사용하며, 하나 의 primer는 표적 RNA와 혼성화되고, 역전사효소에 의해 서 DNA가 합성된다. 만들어진 DNA에 추가적으로 두 번 째 primer가 혼성화되며, 혼성화된 DNA에는 T7 RNA 중 합효소가 반응을 하여 다량의 anti-sense RNA가 중합되게 된다. 이 반응이 반복적으로 수행되면, 다량의 RNA가 중 합되므로 바이러스 및 살아있는 미생물의 검출이 가능하게 된다. 미생물의 세포질에 존재하는 다량의 RNA는 미생물 의 사멸과 함께 빠른 시간 내에 분해가 됨으로 살아있는 미생물의 주요 마커(marker)로 활용할 수 있다. 하지만, NASBA 방법은 다수의 효소(RT-DNA 중합효소, RNase H, RNA 중합효소)를 사용하기 때문에 반응조건이 까다롭 다는 단점이 있다(Niemz et al., 2011).
LAMP (loop-mediated isothermal amplification)는 Notomi 에 의해서 2000년대 초반에 개발된 방법으로 등온에서 핵 산을 증폭시킬 수 있는 기술이다(Notomi et al., 2000; Oh et al., 2016). LAMP는 기존의 PCR법과 유사하지만 Taq DNA 중합효소 대신 Bst DNA 중합효소를 사용한다. 이는 LAMP가 등온에서 핵산의 증폭반응이 진행되므로 phi29 DNA 중합효소와 유사하게 DNA 사슬 displacement 활성 이 큰 DNA 중합효소가 요구되기 때문이다(Tomita et al., 2008). LAMP는 일반적으로 4개의 primer를 사용하며, primer의 신장 및 displacement에 의해 각각의 말단에 loop 구조가 형성되는 특징이 있다. 더불어, 2개의 primer를 사 용하는 PCR법과 달리 4개의 primer를 사용함으로써 표적 DNA에 대한 선택도를 향상시킬 수 있다는 장점을 가진다.
결 론
식품산업에서 위생 및 안전에 대한 인식이 높아짐에 따 라 여러 제도적 장치가 마련되었지만 여전히 식품안전사고 발생률은 증가하고 있다. 이는 식품의 교역량이 증가하고 식품산업이 대형화되고 단체급식이 증가된 것에 원인이 있 다고 할 수 있다. 따라서, 식품 원재료의 생산에서부터 식 품의 재조 및 유통, 그리고 최종 소비자가 섭취할 때까지 식품의 안전을 모니터링 할 수 있는 기술이 필요하게 되었 다. 그로 인해 다양한 기술을 접목시킨 바이오센서들이 개 발되었고, 현재까지도 많은 연구들이 바이오 혹은 식품산 업체, 국가기관 및 대학 연구소에서 진행 중이다. 하지만, 현재까지의 연구결과들은 대부분 물 혹은 배지와 같은 단 순한 매트릭스를 가진 시료에 존재하는 타겟 미생물 및 바 이러스를 검출하는 단계에 머물러 있다. 따라서, 실제 식품 과 같이 복잡한 매트릭스를 갖는 시료에 존재하는 미생물 을 검출하기 위해서는 적절한 시료 전처리 과정이 반드시 선행되어야 한다. 식품시료 전처리 과정을 통해 분리된 미 생물 및 바이러스를 고감도로 검출하는데 있어서 아직까지 는 핵산 기반의 검출방법이 가장 효율적이고 정확하다. 식 품안전사고로 인한 식품산업의 경제적 손실을 줄이고 국민 의 건강을 증진시키기 위해서는 식품안전을 모니터링 할 수 있는 기술이 필요하고, 그를 위해서는 산업현장에서 활 용될 수 있는 식품 시료 전처리 기술과 핵산 기반의 검출 이 유기적으로 연동된 식중독균 검출법의 개발이 반드시 필요하다.







