서 론
해양미생물은 높은 수압과 염분, 낮은 수온 등의 환경에 서 진화를 통해 적자생존의 경쟁에서 살아남기 위해 육상 미생물과는 다른 이화학적 특성을 가진 2차 대사산물을 생 성한다(McCarthy et al., 2004). 우리나라는 삼면이 바다로 둘러싸인 해양 국가이기 때문에 확보할 수 있는 다양한 해 양생물자원을 갖고 있고 이를 이용한 새로운 기능성 식품 이나 약효 식품의 개발은 많은 잠재력을 가진 분야로 주목 받고 있다(Mehbub et al., 2014). 특히 어류나 패류를 발효 시킨 젓갈류, 해초를 이용하여 제조하는 장아찌류나 무침, 젓이나 어류 등이 첨가되는 침채류 등의 다양한 발효식품 제조가 이루어지고 있다(Park et al., 2017).
수산발효식품은 염을 첨가하여 자가소화효소 및 미생물 이 분비하는 효소 반응에 의해 숙성된 것으로, 내염 또는 호염적인 특성을 나타내는 세균이 많이 자라며, g당 103- 105 CFU의 생균수를 나타내는 것으로 보고되어 있다(Park et al., 2017). 수산발효식품에서 분리한 호염성 미생물은 염분 함량이 높은 다양한 자연환경에서 유래하며 계통학적 다양성뿐만 아니라 소화효소 및 세제용 내염성 효소의 개 발과 더불어 수산 부산물과 폐기물을 처리 및 재활용 등 다양한 산업분야에 응용되고 있다. 특히 산업용 효소시장 에서 가장 큰 비율로 사용되고 있는 단백질가수분해효소는 식품, 세제, 섬유 펄프와 피혁 공업, 의약품 등에 이용된다 (Singh et al., 2016). 속성발효법으로 수산발효식품의 숙성 을 단축시키기 위한 starter로서 protease를 생성하는 호염 성 미생물의 군집분석(Mayuri et al., 2019), 염장 식품의 풍미 증가와 향미 발달, 육질 분해 등을 위한 protease를 생성하는 미생물의 특성 규명 등 활발한 연구가 수행되고 있다(Giyatmi and Irianto, 2017;Zang et al., 2020). Protease 뿐 아니라, 미생물 유래 효소를 이용하는 산업적 응용은 많은 수가 식품산업분야의 효소 활용에서 시작되었다. 이 에 식품 분야에 대한 연구와 함께 식품가공분야에 효소의 사용은 식품 보존, 식품유지 가공, 곡물 가공, 계란 가공, 생선 가공, 제과/제빵, 과일/야채주스, 시럽 제조, 향기 및 맛 제조, 맥주 등 알코올 발효, 낙농, 동물사료 등 많은 분 야에 이용되고 있다(Singh et al., 2016). 언급한 분야뿐 아 니라 거의 모든 식품관련 분야에 식품의 보존, 가공, 제조, 발효 등에 있어서 protease, amylase, lipase 등으로 적용되 고 있다(Mayuri et al., 2019).
본 연구에서는 다양한 발효식품으로부터 호염성 미생 물을 순수 분리 후, 산업적으로 응용 가능한 효소 4종 (amylase, lipase, cellulase, protease)의 활성을 탐색하였다. 또한 작물의 생육을 촉진하는 기능을 함으로써 화학비료를 대체하는 미생물비료로의 활용을 위한 auxin 생산능 분석 을 실시하여 호염성 미생물 자원의 가치를 제고하고 산업 용 효소 관련 생물공학 및 미생물비료 연구의 기본 생물 소재로 활용이 가능할 것으로 예상된다.
재료 및 방법
본 연구에 사용된 해양 유래 발효수산물은 남(서)해안의 쿠로시오 난류가 흐르는 지역에서 얻은 것으로 곰소 전통 젓갈 가게에서 구입한 밴댕이 젓 무침, 순천만에서 구입한 간장게장과 오징어 젓갈, 서귀포 올레시장에서 구입한 갈 치속젓, 통영에서 구입한 멸치젓, 대마도에서 구입한 조개 맛 오이로 총 6가지 시료를 사용하였다. 또한 본 연구를 통하여 분리한 모든 균주들은 KRIBB 미생물가치제고사업 단에 기탁하였다.
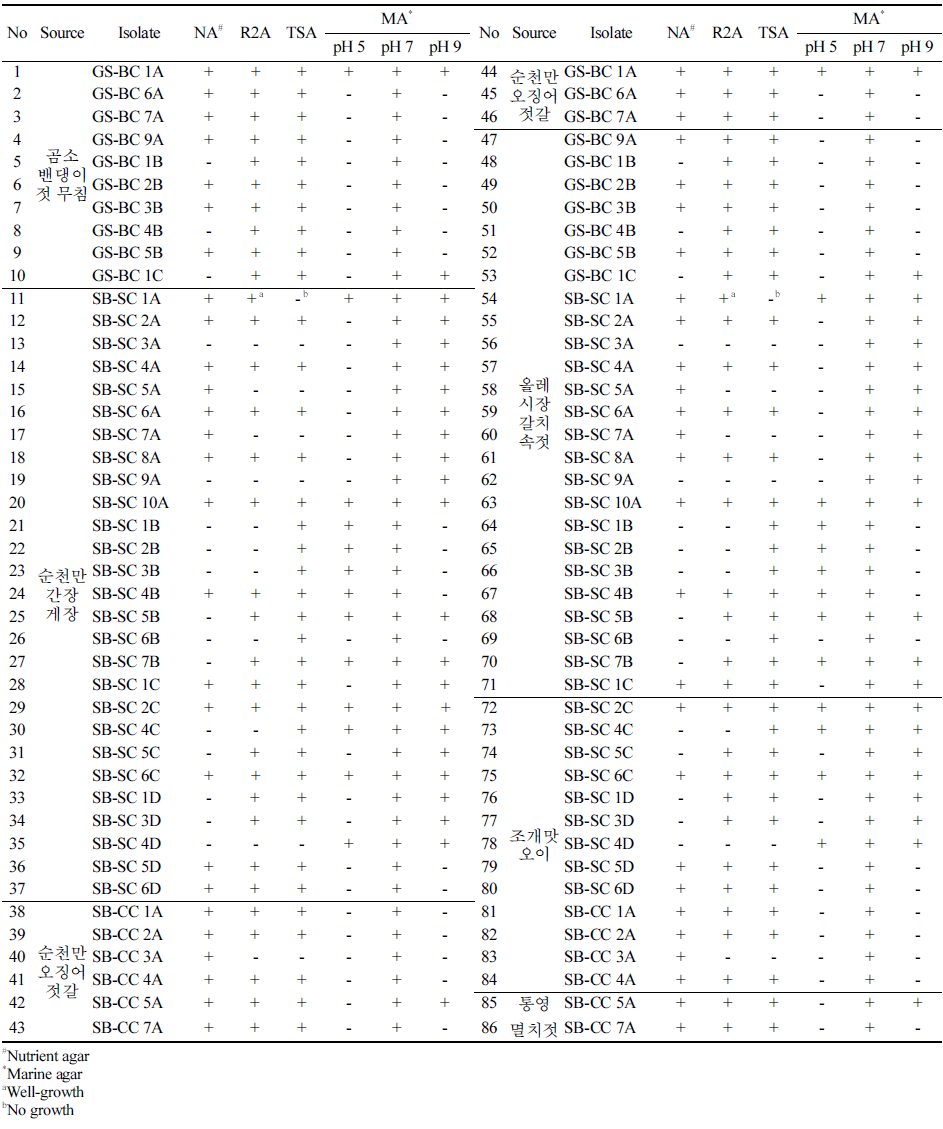
국내외 다양한 해양 유래 발효산물로부터 호염성 미생물 을 분리하기 위해 각 샘플을 멸균된 0.85% saline solution 에 첨가하여 vortexor로 현탁 후 10−1-10−4배로 단계 희석해 샘플을 준비하였다. 일반 증식배지로 해양미생물 전용배지 인 marine agar (BD, Franklin, NJ, USA)를 제작하여 희석 액을 도말하고 25, 37, 50°C에서 호기적으로 호염미생물을 배양한 후 선택적으로 배지상에 나타나는 균의 크기, 모양, 색깔 등 형태학적 모습에 따라 동일한 고체배지에 추가적 으로 single colony isolation을 수행하였다. 혼합배지에서의 생육 가능성을 확인하기 위해 순수 분리된 균주를 nutrient agar (BD), R2A agar (BD), 및 tryptic soy agar (BD)에 선상도말법을 이용하여 25, 37, 50°C에서 7일간 정치 배양 하였다.
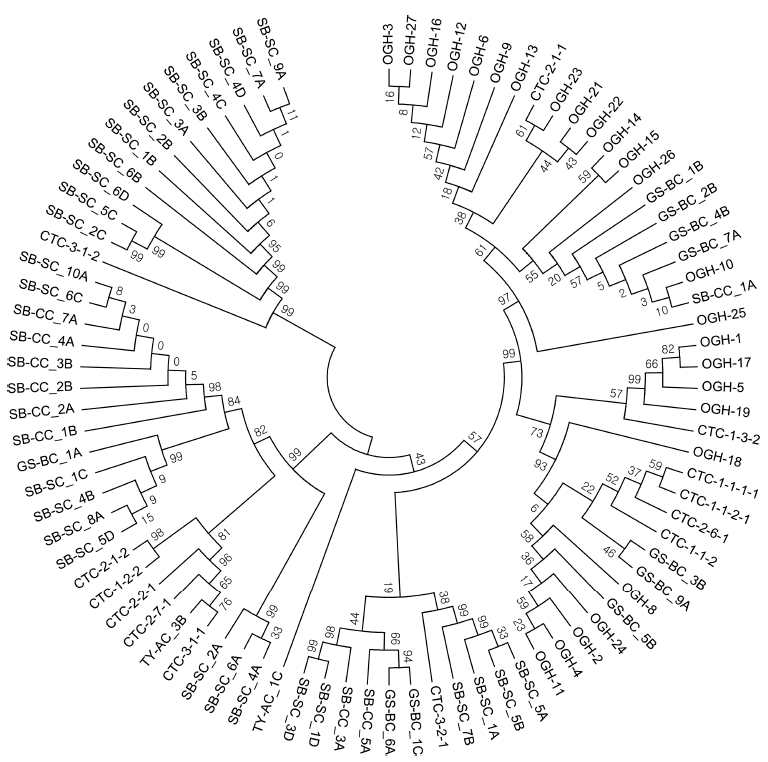
국내외 다양한 해양 유래 발효산물로부터 호기적 배양 조 건에서 분리된 균주들의 분자생물학적 동정을 위해 marine agar (BD) 배지에 각각 분리된 균주의 단일 colony가 배양 된 상태의 고체 배지를 ㈜바이오팩트에 보내어 16S rRNA 염기서열의 분석을 의뢰하였으며 분석된 16S rRNA 염기서 열로부터 가장 유사한 근연 균주의 확인을 위하여 ㈜천랩 의 EzBioCloud (https://www.ezbiocolud.net/)를 사용하였 다. 계통학적 분석은 Clustal W 및 MEGA 6 프로그램을 이용하여 phylogenetic tree를 작성하였다.
분리된 호염성 미생물의 세포외 분해 효소 amylase, lipase, cellulase, protease 활성 확인을 위하여 각각의 효소 와 특이적으로 반응할 기질 성분이 포함된 고체평판 선별 배지를 사용하였다. 먼저 amylase 활성은 0.2% soluble starch (BD)를, lipase 활성은 1% Tween 80 (BD)을, cellulase 활성은 1% CMC (BD)를, protease 활성은 20% skim milk (BD)를 기질로 선택하여 marine agar (BD) 배 지에 각각 첨가하여 제조하였으며 분리된 균주를 직접 접 종하여 25, 37, 50°C에서 7일 배양한 후 저지원(Clear zone)의 직경으로 조사하였다. 분리된 균주의 효소활성 분 해능 평가는 배양 후 나타나는 접종균 주위의 Clear zone 의 크기(+++: > 7 mm, ++: > 4-6 mm, +: 1-3 mm)로 나타 내었다.
결과 및 고찰
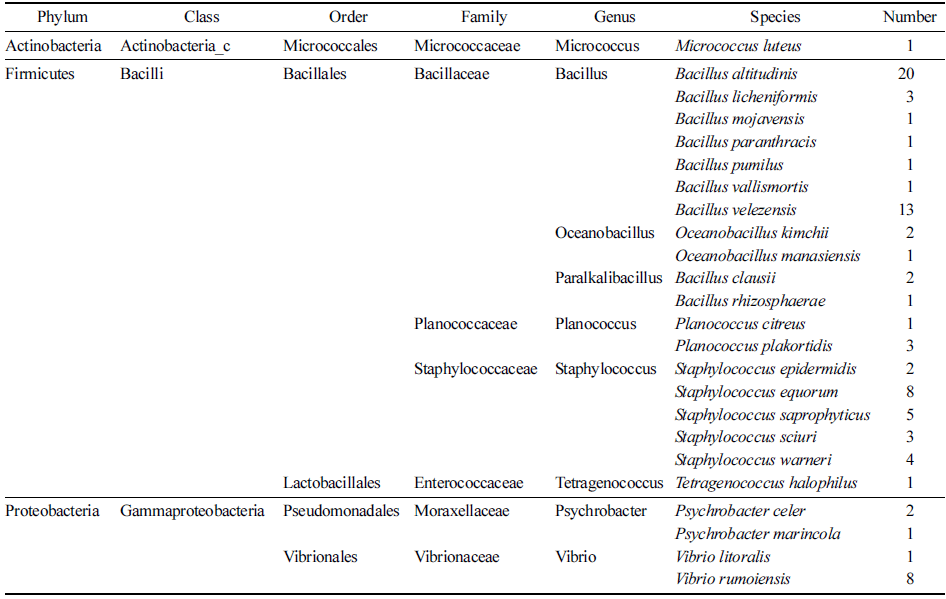
국내외 해양 유래 발효산물로부터 식품 산업에의 활용이 용이할 것으로 생각되는 호기적으로 생육 가능한 호염성 미생물을 분리하고자 marine agar 배지에 시료를 희석, 도 말하여 배양한 후, 배양된 colony의 모양, 색깔, 등 형태학 적 특징을 육안으로 구분이 가능한 균주들을 대상으로 동 일한 고체배지를 사용하여 2차로 단일 균주 분리를 수행하 였다. 그 결과 Table 1에서 나타낸 것처럼 곰소 밴댕이 젓 무침으로부터 10균주, 순천만 간장 게장으로부터 27균주, 순천만 오징어 젓갈로부터 9균주, 올레시장 갈치속젓으로 부터 25균주, 조개맛 오이로부터 13균주, 통영 멸치젓으로 부터 2균주가 각각 분리되었으며 본 실험을 통하여 호염성 미생물 총 86균주를 순수 분리하였다. 순천만 간장 게장으 로부터 가장 많은 27균주가 분리되었다. 이것은 발효시키 는 원료에 따라 호염성 미생물의 분리가 상이하게 나타난 결과로 예상된다. 또한 주요 성분이 무기염으로 이루어진 marine agar 배지는 해양 미생물 배양에 유리한 배지이기 에 산업적 활용가능성을 확인하기 위하여 대량 배양 등에 많이 활용되는 혼합배지(nutrient agar, R2A agar, tryptic soy agar)에서의 분리 균주들의 생육 가능성을 확인한 결 과 86균주 중 6균주를 제외한 80균주(93.0%)가 최소 1종 류 이상의 혼합 배지에서 생육이 가능한 것을 확인하였다. 이는 해양 유래 발효산물의 호염성 미생물 탐색에 있어서 분리 배지의 구성성분이 어느 정도 영향을 미치는 것으로 생각되며, 본 연구 결과를 바탕으로 해양 유래 발효산물 호염성 미생물 분리를 위한 최적 배지는 marine agar 배 지로 나타났다. 또한 최적의 생육 pH 조건을 확인하기 위 하여 pH를 5, 7, 9로 각각 조절한 marine agar 배지에 분 리 균주들의 생육을 확인해 본 결과 분리된 균주 중 Staphylococcus epidermidis CTC-1-2-2를 제외한 모든 균주 가 pH 7에서 생육이 가능하였으며 이중 31균주는 pH 5에 서도 생육이 가능하였으며 pH 9에서 생육이 가능한 균주 는 45균주였다(Table 1).
|
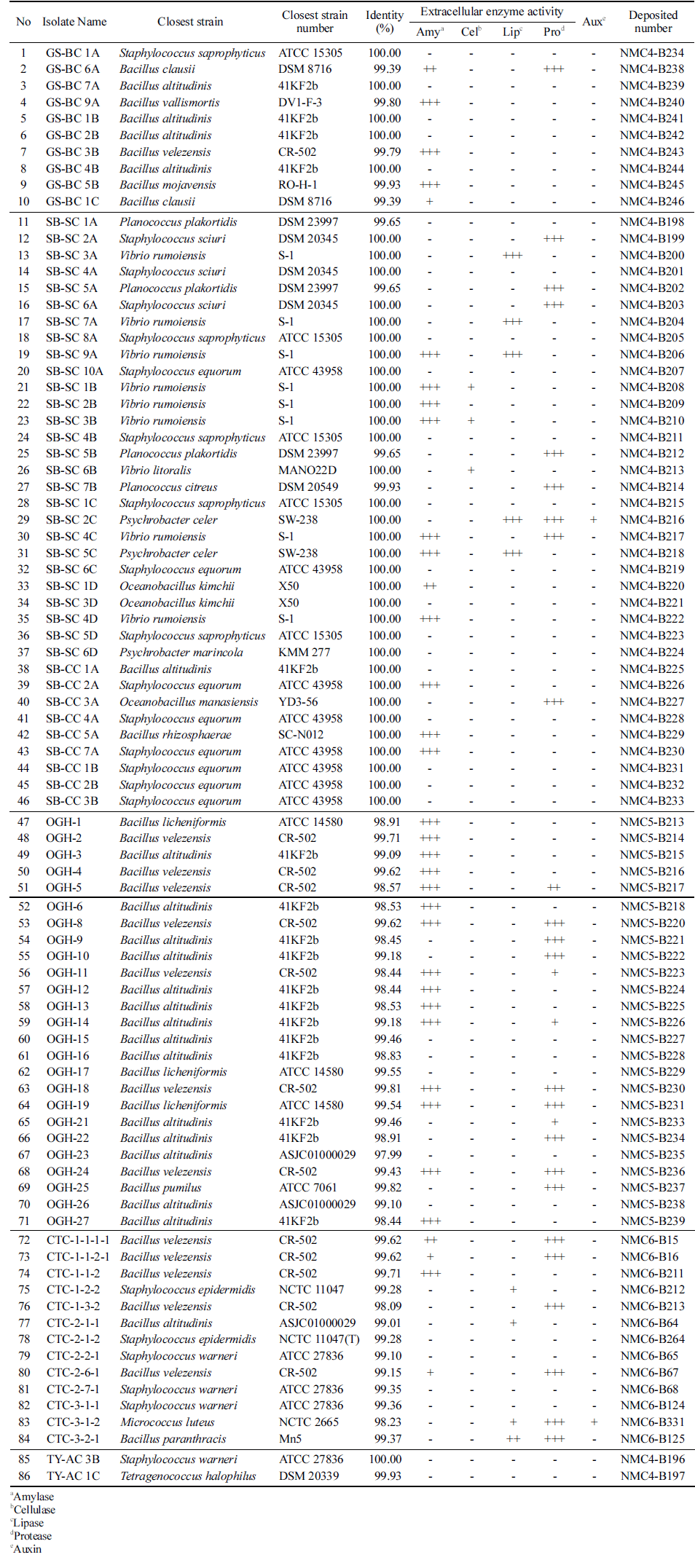
국내외 다양한 해양 유래 발효산물로부터 호기적으로 분 리된 86균주의 16S rRNA 염기서열을 바탕으로 NBLAST program과 EzBioCloud를 사용하여 미생물 동정을 실시 한 결과 크게 3문 5목 7과 9속 24종으로 나타났으며 (Table 2), 분리 균주와 근연 균주 및 상동성을 Table 3 에 나타내었다. 또한 분리 동정된 균주들 간의 유연관계 를 확인하기 위하여 계통수를 작성하였다(Fig. 1). Table 2에서 보는 바와 같이 Firmicutes (Bacilli)가 84.9%로 가 장 우점도가 높았고, Actinobacteria 1.1%, Proteobacteria (Gammaproteobacteria) 14.0%로 나타났다. 가장 우점도가 높은 Firmicutes 문은 Bacillaceae 63.0%, Staphylococcaceae 30.1%, Planococcaceae 5.5%, Enterococcaceae가 1.4%로 구성되었으며, 총 4과 6속 19종이 분리되었다. 본 연구에 서 가장 많이 분리된 Bacillaceae과는 Bacillus sp.이 해양 생물 중에서도 담수어에서 많이 분리되며 발효식품 중에 서도 젓갈류에서 주로 분리된다는 것이 알려져 있기에 해 양 유래 발효산물의 미생물 균총에서 가장 많이 분리된 것 으로 예상된다(Yang, 2005;Hur, 1996;Lee, 1969;Nam, 2012). 또한 metagenome 분석에 기반을 둔 미생물상 분석 이 전통발효식품에 적용되면서 기존 해양 유래 발효균총 연구에 알려지지 않았던 Staphylococcaceae과 Staphylococcus 속의 22종이 확인되었다(Guan et al, 2011). 이들 균주는 coagulase를 생산하지 않아 식품위해균 Staphylococcus aureus와 구분되며, 항생제 내성이 있고, 위해성은 보고되 지 않은 coagulase-negative staphylococci (CNS)로 분류된 다(Blaiotta et al., 2004). Actinobacteria 문에서 1속 1종 (Micrococcus luteus)이 분리되었으며 Proteobacteria 문에서 2속 4종이 분리되었다. 또한 분리 균주 중 근연 균주와의 16S rDNA 염기서열 상동성이 낮은 10균주(98.7% 이하)는 신종 균주 가능성을 나타내는 것으로 올레시장 갈치속젓(8 종)과 조개맛오이(2종)에서 분리(KRIBB기탁번호: NMC5- B217, 218, 221, 223, 224, 225, 235, 239, NMC6-B213 및 331) 되었으며, 동정관련 실험을 정밀하게 진행중이다. 이는 국내 생물 자원의 다양성 확보 차원에서 의미가 있을 것으로 생각된다.
수산 발효식품은 어류 또는 패류의 근육, 내장 등에 염 을 첨가하여 자가소화 및 미생물이 분비하는 효소반응에 의해 숙성시킨 전통 발효식품으로 알려져 있기에(Park et al., 2017) 분리된 호염성 미생물 균주들의 식품 산업에 적 용 가능한 신규 분해 효소 탐색 자원 및 미생물 비료로써 의 산업적 응용가능성을 확인하기 위하여 세포외 분해 효 소 생산능 및 옥신 생산능 분석을 실시하였다. Table 3에 나타낸 것처럼 분리된 86균주 중 55균주(64.0%)에서 한 가지 이상의 분해 효소 활성이 존재하는 것을 확인하였으 며, amylase 활성을 보이는 35균주, cellulase 활성을 보이 는 3균주, lipase 활성을 보이는 9균주, protease 활성을 보 이는 27균주를 확보하였고, 이 중 두 가지 이상의 효소 활 성을 가지는 19균주도 확인되었다. 특히 cellulase 활성을 보이는 3균주(Vibrio rumoiensis SB-SC 1B, 3B 및 V. litoralis SB-SC 6B)는 순천만 간장 게장에서 분리된 Vibrio sp. 이었다. 해양 서식처에 널리 분포하는 Vibrio는 현재 70종 이상이 보고되었으며 그 개체 수는 해마다 빠르게 증 가하고 있다(Park et al., 2003). 최근 다른 종의 Vibrio로부 터 cellulase가 분리되어 특성이 규명되었다는 보고와 함께 알코올 발효(맥주, 주정), 생선가공, 동물사료 등 다양한 식 품산업에 적용 가능할 것으로 생각된다(Li et al., 2003). 또한 옥신 생산능을 확인한 결과, 2균주(Psychrobacter celer SB-SC 2C와 Micrococcus luteus CTC-3-1-2)에서 생 산능이 확인되었으며 순천만 간장 게장과 조개맛 오이에서 각각 분리된 Psychrobacter celer와 Micrococcus luteus 모 두 생산능이 매우 높은 것을 알 수 있었다. 이는 식물 생 장을 촉진시킬 수 있는 미생물비료 연구에 활용할 수 있는 미생물 탐색 소재로써의 활용가치가 높을 것으로 예상된다. 본 연구에서 수행한 결과들은 국내 미생물 생물 자원의 다 양성 확보 차원에서 큰 의미를 찾을 수 있을 것이며 국내 외 해양유래 발효산물로부터 분해 효소 탐색을 위한 신균 주의 활용 가능성과 산업용 효소 관련 생물공학 및 미생물 비료 연구의 기본 생물소재로 활용이 가능할 것으로 예상 된다.
요 약
본 연구는 국내외 해양유래 발효산물 시료로부터 분리 한 호염성 미생물들의 다양성 및 특성에 관하여 조사하였 다. 호염성 미생물의 순수 분리를 위하여 marine agar 배 지를 사용하였으며 25, 37, 50°C에서 호기적으로 배양하 였다. 순수 분리 후, 86균주를 분리하였으며 16S rRNA 염기서열 분석 결과를 바탕으로 계통학적 분석을 실시한 결과, 3문, 7과, 9속, 24종으로 구성되어 있는 것을 확인하 였다. 특히 Firmicutes문 Bacilli강은 84.9%의 분포를 나타 내었으며 4과, 6속, 19종으로 Bacillaceae, Planococcaceae, Staphylococcaceae와 Enterococcaceae로 분포하는 것을 확인하였다. 그리고 분리한 균주들이 amylase, lipase, cellulase, protease 같은 산업적으로 유용한 효소를 생산하 는지 확인하기 위하여 효소 활성 평가를 실시하였으며, 55 균주가 최소 한 종류 이상의 효소 활성을 가지고 있는 것 을 확인하였다. 또한 옥신 생산능을 가지는 균주도 2 균주 가 확인되었으며 이는 본 연구를 통하여 분리한 미생물들 의 산업적 활용 가능성을 나타내었다. 그러므로 이번 연구 는 국내 유전자원 확보 및 시료의 호염성 미생물의 다양성 과 특성에 관한 과학적 지식 확장에 도움이 될 것으로 생 각된다.