서 론
단백질 분해효소는 세계 효소 생산의 약 25%를 차지하 고 있으며 산업용 효소 중 가장 중요한 효소 중 하나이다 (Kumar & Takagi, 1999). 단백질 분해효소(protease)는 생 물학적 진화에 있어서 가장 초기에 나타났을 것으로 추정 하고 있으며, 현재의 protease들도 미생물 기원의 단백질 분해효소와 공통점이 있으며 수억년 전에 출현했으리라 보 고 있다. 고분자 단백질의 흡수 기작이 없는 미생물에 있 어서 protease 분비능의 의미는 amylase나 lipase 등과는 또 다른 차원으로서 열량원 외에 질소원까지 얻을 수 있다 는 중요한 의미를 나타낸다(Kim et al., 2002). 촉매과정이 protein이나 peptide의 내부에 존재하는 peptide bond를 분 해하기 때문에 peptidase라 부르기도 하는데, 이때 효소가 작용하는 amide bond의 위치에 따라서 exopeptidase와 endopeptidase로 분류하며, 미생물이 체외로 분비하거나 동 물이 장내로 분비하는 대부분의 protease는 endopeptidase 에 속한다(Kim et al., 2002). 알칼리성 protease는 1974년 Linderstrom과 Othesen이 Bacillus licheniformis로부터 알칼 리성 protease인 subtilisin novo (Subtilis in BPN)를 발견 하였다(Choi et al., 1996). 1971년에는 호알칼리성 세균인 Bacillus sp.로부터 알칼리성 protease (Horikoshi, 1971)가 발견되었고, 이후 대부분의 연구진들은 세제, 연육가공, 탈 모공정 등에 있어서 알칼리성 단백질 분해효소의 탁월한 유용성으로 인해 활성이 높고, 안정된 성질을 갖는 새로운 균주 선별에 많은 노력을 기울여 왔다(Nakadai et al., 1973; Kobayashi et al., 1985; Durham et al., 1987). 또한 이들 효소에 대한 분비기작(Delepelaire & Wandersman, 1989), 삼차원구조(Wright et al., 1969; Duman & Löwe, 2010), 불활성 기작(Diermayr et al., 1987), 단백질 공학적 방법에 의한 구조적 안정화(Pantoliano et al., 1987), 면역 학적 비교연구(Hugenholtz et al., 1984), 아미노산 배열순 서 및 비교분석(Nedkov et al., 1985), 저해물질(Tanizawa et al., 1990), 유전자 조작 및 발현(Power et al., 1986) 등 에 관하여 폭넓은 연구가 이루어졌다. 호알칼리성 protease 는 알칼리 범위의 pH에서 최적 활성을 가지며, 대부분이 효소활성 부위에 serine 잔기를 포함하고 있어서 serine과 특이 반응을 하는 물질에 의해서 쉽게 활성을 잃는 특징을 갖는 효소로 알려져 있다(Ahn et al., 1990). 호알칼리성 protease에 관한 연구는 Horikoshi (1971)가 alkalophilic bacteria, Kobayashi et al. (1985)은 Pseudomonas maltophilia, Nakanishi et al. (1974)은 Streptomyces sp., Bang & Jeong (2011)은 B. pseudofirmus HS-54에서 호알칼리성 protease 가 분비된다고 보고하였다. 현재 생명공학 산업과 타 산업 이 연관되어 미생물 자원은 의학 분야의 경우 항생제 생산, 환경 분야에 있어서는 유해물질의 분해, 악취제거, 생활하수 및 산업폐수처리, 농업분야의 경우에는 미생물 살충제 및 미생물 비료로써 활용과 더불어 에너지 산업에서 대체 에 너지 개발 등에 이용되고 있다. 또한 다양한 식품 분야에 서 식품 첨가제, 효소 등으로 이용되는 등 많은 산업 분야 와 연계되어 활용되고 있다. 따라서 호알칼리성 protease 생산균주 등 산업적으로 유용한 다수의 미생물들이 확보될 경우 여러 산업의 발전을 유도하는 파급 효과를 발생시킬 수 있을 것이다.
본 연구에서는 호알칼리성 protease를 생산하는 미생물을 토양으로부터 분리하고, 동정하여 추후 식품 및 세제분야 등 산업으로 유용하게 이용될 수 있는 균주를 선발하고자 했다.
재료 및 방법
충청도, 강원도 일대의 해변가, 산악지, 축사주변의 퇴비, 채소밭 등으로부터 토양과 두엄을 채취하여 알칼리성 배지 에서 생육하는 균주를 선별하였다. 균주 선별 방법은 토양 또는 두엄시료 1 g을 생리식염수 10 mL에 현탁하고 30분 간 방치한 후 상등액 100 μL를 Horikosh I 평판배지에 도 말하였다. Horikosh I 배지는 별도로 멸균된 1% (w/v) glucose를 제외하고 멸균된 나머지 성분(0.5% (w/v) polypeptone, 0.5% (w/v) yeast extract, 0.1% (w/v) K2HPO4, 0.02% (w/v) MgSO4· 7H2O, 1% (w/v) Na2CO3, 2% (w/v) agar, pH 10.2)과 혼합하여 제조하였다. 분리시료를 도말 후 40°C 배양기에서 24시간 또는 48시간 배양하여 생육한 호알칼리성 균주를 1차로 선별하였다. 1차 선별된 균주들 중 단백질 분해효소 활성을 확인하기 위하여 2% (w/v) skim milk가 포함된 Horikosh I 평판배지에 분리된 균주를 접종한 후 40°C에서 배양하여 균의 집락 주위에 clear zone이 형성된 균주를 protease 활성을 갖는 균주로 2차 선정하였다. 2차 선정된 균주는 Horikosh I 액체배지에 재 접종하여 40°C에서 24-48시간 진탕배양(200 rpm)한 후 배 양 상등액에 존재하는 단백질 분해효소의 활성을 측정하여 상대적으로 효소활성도가 높은 균주를 최종 선별하였다.
단백질 가수분해활성의 기질로는 azocasein (Sigma, St. Louis, MO, USA)을 사용하였다. 10 mM sodium phosphate buffer (pH 9.0)에 녹인 2% azocasein 250 μL에 효소액 150 μL를 첨가하여 60°C에서 10분간 반응시켜 azo기를 유리시켰다. 여기에 600 μL의 20% (w/v) trichloroacetic acid를 가하여 잔존활성을 실활시키고, 9,000×g에서 10분 간 원심분리하여 반응하지 않은 azocasein은 침전, 제거하 였다. 상등액 450 μL에 700 μL의 1 N NaOH를 혼합하고 spectrophotometer (UV-1201, Shimadzu, Tokyo, Japan)를 이용하여 440 nm에서 흡광도를 측정하였다. 이때 효소 활 성도 1 unit은 10분간 반응에 의하여 유리되는 azo기에 의 해 440 nm에서의 흡광도를 0.001 증가시키는 양으로 정하 였다.
균주의 형태학적인 특성은 Gram staining 후 광학현미경 을 이용하여 관찰하였다. 또한 주사현미경(scanning electron microscope, SEM; S-800, Hitachi, Ltd., Tokyo, Japan)을 이용하여 균체형태를 보다 자세히 관찰하였다.
균주의 주요 생화학적 특성은 Bergey's manual of systematic bacteriology (Vos et al., 2009)와 Methods for general and molecular bacteriology (Gerhardt et al., 1994) 에 준하여 포자형성 유무, catalase, oxidase, nitrate 환원성 및 casein, gelatin, starch 가수분해능을 분석하였다. 모든 생화학적 실험은 B. pseudofirmus를 대조군으로 비교하여 실험하였다.
포자 형성 유무의 실험은 Strain DK1122 균체 배양액 일부를 취하여 100°C에서 20분간 중탕하였다. 이를 100 μL 취해 Horkosh I 배지에 pour plate법으로 접종하여 37°C에서 24시간동안 배양하여 그 생육여부로 포자 생성 능을 확인하였다. 또한 백금이로 균체를 취해 slide glass에 도말하고 malachite green으로 염색하여 30초간 증기 처리 한 후 증류수로 세척하였으며, 이를 safranine O로 20초간 염색하여 광학 현미경으로 관찰하여 포자형성 유무를 재확 인하였다. Catalase test는 균체 일부를 화염 멸균한 백금이 로 취해 slide glass에 도말하여 20 μL의 30% H2O2를 건 조된 균체에 가한 다음 기포의 생성 유무를 확인하였다. Oxidase test는 균체를 화염 멸균한 백금이로 취해 여과지 에 고르게 도말한 후 1% tetramethyl-p-phenylenediamine dihydrochloride 시약을 2-3방울 가하여 10초 이내 색이 청 색으로 변하는 것을 양성으로 하였다. Nitrate 환원성은 Horikoshi I 배지에 0.1% KNO3와 0.2% agar를 첨가한 후 균주를 접종하여 40°C에서 24시간 배양한 다음 기포생성 유무를 확인하여 1차 판정을 하였다. 기포 생성이 확인된 후 2차 판정을 위해, A 용액과 B 용액을 제조한 후 균배 양액 100 μL에 A 용액과 B 용액을 1 mL씩 가하여 적색 이나 분홍색을 띄는 것을 양성으로 판단하였다. A 용액은 1 N HCl 100 mL에 0.02 g N-(1-Naphthyl)ethylenediamine dihydrochloride를 첨가하여 제조하였으며, B 용액은 100 mL 1 N HCl에 1 g H2SO4를 첨가하여 제조하였다. 2차 확 인시 색의 변화가 관찰되지 않을 경우 zinc powder를 첨 가하여 3차 확인을 하는데, 이때 색이 변하면 음성으로 판 단하고 색변화가 없을시 양성으로 판단하였다. Casein, gelatin, starch의 가수분해 실험은 Horikoshi I 배지에 각각 0.5% casein, 20% gelatin, 1% starch를 첨가하여 사용하였 다. 균체를 접종하여 40°C에서 24시간 배양한 후 casein 분해활성은 clear zone의 생성 여부를 확인하였고, gelatin 분해활성은 배양 후 4°C의 저온에 방치하여 gelatin의 액화 여부를 확인하였다. Starch 분해활성은 iodine solution을 가 하여 교반시킨 뒤 clear zone의 생성여부를 확인하였다.
Strain DK1122 세포의 지방산 조성은 gas chromatography (GC, HP 6890, Hewlett Packard, Palo Alto, CA, USA) 를 사용하여 분석하였다. 표준품으로는 Hewlett-Packard사 의 표준지방산(free fatty acid calibration standard)을 이용 하였다. 균주의 배양은 Horikoshi I 배지를 사용하여 40°C 에서 24시간 동안 배양된 균체를 시험관에 취해 saponification을 위하여 reagent I (45 g sodium hydroxide, 150 mL methanol, 150 mL deionized water)을 1 mL 가하고 30 분간 100°C에서 중탕 후 흐르는 물로 냉각하였다. Methylation을 위하여 reagent II (325 mL 6 N hydrochloric acid, 275 mL methanol)를 2 mL 가한 후 80°C에서 10분 간 반응시키고 흐르는 물에 냉각하였다. Extraction을 위하 여 reagent III (200 mL n-hexane, 200 mL methyl-t-butyl ether)을 1.25 mL를 가한 후 10분간 상온에서 천천히 진탕 한 후 하층액을 제거하였다. Washing은 reagent IV (10.8 g sodium hydroxide, 150 mL deionized water)를 3 mL 가 하여 5분간 천천히 진탕하였으며, 층 분리를 위하여 포화 NaCl 용액 500 μL를 가하여 분리된 상등액을 취해 분석 용 시료로 사용하였다. 이것을 GC로 분석 후 Sherlock program (MIDI Inc., Newark, DE, USA)을 이용하여 분석 하였다.
16S rRNA 유전자의 염기서열을 분석하기 위하여 strain DK1122를 Horikoshi I 배지 5 mL에 접종하여 배양기 (KMC-8480S, Vision Science, Seoul, Korea)에 40°C, 250 rpm으로 24시간 동안 진탕배양하였다. 배양액을 13,000×g, 10분간 원심분리하여 균체를 회수하여, strian DK1122의 chromosomal DNA를 lysozyme-sodium dodecyl sulfateproteinase K 방법(Sambrook & Russell, 2001)으로 분리한 후, polymerase chain reaction (PCR)을 통해 16S rRNA 유전자를 증폭하기 위한 template로 사용하였다. PCR에 사 용된 primer는 16S rRNA 유전자 증폭에 사용하는 universal primer set (forward primer: Eubacterial 27F: 5'- AGAGTTTGATCATGGCTCAG-3'; reverse primer: universal 1492R: 5'-GGATACCTTGTTACGACTT-3')을 사용하였 다. PCR 반응액의 조성은 10 μL template (50 ng/μL), 5 μL 10× reaction buffer (100 mM Tris-HCl, 400 mM KCI, 500 μg/mL BSA, pH 8.3), 5 μL 1.5 mM MgCl2, 5 μL dNTP (2.5 mM each), 각각 1 μL primer (100 pmol/μL), 22 μL의 멸균 3차 증류수 혼합하여 최종 50 μL로 구성하였다. 이 혼합액을 thermocycler (GeneAmp 9600, Perkin Elmer, Waltham, MA, USA)를 사용하여 증폭하였다. 증폭을 위한 PCR 조건은 94°C에서 1분간 denaturation, 60°C에서 1분 간 annealing, 72°C에서 1.5분 동안 polymerization하는 조 건으로 30 cycle을 행하였으며, 최종 72°C에서 10분간 증 폭시켰다. 0.8% agarose gel에서 PCR product 크기를 확인 하였다. 확인된 PCR product는 Bio 101 gene cleaning kit (Bio-Rad, Hercules, CA, USA)를 사용하여 정제 후, pCRTM4-TOPO® vector (Invitrogen, Carlsbad CA, USA)에 ligation 시킨 후, Escherichia coli competent cell에 클로 닝하였다. PCR product를 클로닝한 vector를 갖고 있는 균주를 선별하고, plasmid DNA를 분리한 후, 이를 Tvector sequencing primer (M13 forward, M13 reverse)를 이용하여 ALFred automated DNA sequencer (Pharmacia, Uppsala, Sweden)를 사용, strain DK1122의 16 rRNA 유 전자 염기서열을 결정하였다. 그 결과는 Blastn program (http://blast.ncbi.nlm.nih.gov/Blast.cgi)을 이용하여 GenBank 와 RNA database project (RDP)의 염기서열과 비교하여 동정하였다.
Blastn program을 이용하여 16S rRNA 유전자의 염기서 열 분석 결과와 similarity가 높은 균주를 확인하였고, 유전 자 염기서열은 GenBank에 등록하였다. Dendrogram 작성 은 Clustal X program과 Tree View program을 이용하였다.
결과 및 고찰
호알칼리성 균주로부터 단백질 분해효소 활성을 갖는 균 주의 선별을 통해 alkaline 조건에서 생육하는 26균주를 1 차로 선별하였다. 1차 선별된 균주를 skim milk가 포함된 Horikosh I 평판 배지에 접종하여, 균체 주위의 clear zone 형성 유무에 의한 효소활성을 확인하여 20균주를 2차 선 별하였다. 2차 선별한 균주를 액체배양하여 배양 상등액으 로부터 단백질 효소활성을 측정한 결과 가장 활성이 높은 균주를 선별하여 이를 strain DK1122라 명명하였다.
호알칼리성 strain DK1122 균주의 형태학적 특성을 알아 보기 위해 균체를 Gram 염색하여 광학 현미경으로 관찰한 결과, Gram 양성의 간균임을 확인하였다. 또한 SEM으로 관찰하였을 때, 크기는 0.7×2-4 μm 정도였으며, 표면에 작 은 돌기가 형성되어 있었다(Fig. 1).
호알칼리성 strain DK1122의 포자형성 유무 실험 결과 균체 내부 포자의 색이 녹색으로 관찰되어 포자형성을 확 인하였다. 30% H2O2를 가하여도 기포형성이 되지 않은 것 으로 보아 catalase 음성으로 확인되었다. Oxidase test 결 과 균체의 색이 청색으로 변해 양성으로 판단하였다. Nitrate의 nitrite로의 환원성 실험에서는 48시간 배양한 strain DK1122가 기포를 형성한 것을 1차로 확인하였고, 2 차로 시약을 가한 균배양액 100 μL가 pink색으로 변하는 것을 확인하여 양성으로 판단하였다. Casein을 함유한 배 지에서 균체 주위에 clear zone이 형성되었으므로 casein 분해 능력이 있음을 확인할 수 있었으나, starch의 경우 clear zone이 형성되지 않았으며 gelatin도 액화되지 않아 starch와 gelatin분해 능력은 없는 것으로 확인되었다. 이상 의 결과로부터 본 균주는 Bergey’s manual에 기술된 Bacillus sp.의 특성을 나타냈다(Vos et al., 2009). 그 중 가 장 유사한 B. pseudofirmus를 대조군으로 모든 생화학적 실험을 비교하여 실험하였다. 분리균주와 비교한 결과 B. pseudofirmus는 gelatin 분해할 수 있었고, 생육온도에서 분 리균주는 50°C에서 자랐으나, 대조균주는 생육하지 못한 차이점을 보였다.
호알칼리성 strain DK1122의 whole cell fatty acid를 GC 로 분석하였다. 그 결과 Strain DK1122는 C15:0 ISO 7.56% 와 C15:0 ANTEISO 61.53%, C17:0 ANTEISO 14.15%로 구성된 것 으로 분석되었다(Table 1). 이는 Sherlock program 지방산 분석 결과 및 다른 연구에서 지방산이 분석된 Bacillus sp. 의 대표적인 지방산 분포(Cha & Park, 2001; Kim et al., 2009)와도 유사한 것으로 나타났다. 따라서 본 균주는 Bacillus sp.로 분류되었다.
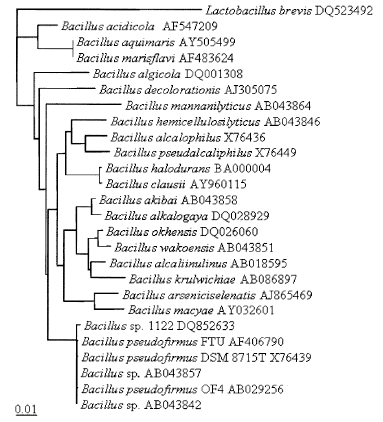
호알칼리성 균주 DK1122의 16S rRNA 유전자 염기서열 분석 결과 1,494 bp의 염기서열을 확인할 수 있었으며(Fig. 2), 이를 GenBank에서 유사성을 검증한 결과 등록된 균주 중 B. pseudofirmus와 99.7%의 유사성을 보였다. 그러나 생화학적 특성에서 B. pseudofirmus와는 gelatin 분해능과 생육온도에서 차이점을 보였으며, Genbank database 상에 서 16S rRNA 유전자 염기서열이 100% 일치하는 균주는 검색되지 않았으므로 기존에 발표되지 않은 새로운 균주로 판단되어 Bacillus sp. DK1122로 명명하였다. 균종간의 유 의성은 GenBank에 등록된 균주의 16S rRNA gene을 사용 하여 dendrogram을 작성하였고(Fig. 3), 유전자 염기서열은 GenBank에 등록하였다(accession number: DQ852633).
요 약
호알칼리성 단백질분해 효소를 분비하는 Bacillus sp. DK1122 균주를 토양으로부터 분리하여 동정하였다. 토양 으로부터 분리한 호알칼리성 균주 20균주 중 단백질 효소 활성이 가장 큰 균주인 strain DK1122를 선택하였다. Gram staining과 SEM을 통하여 형태학적인 특성을 관찰한 결과 본 균주는 Gram 양성, 간균으로 판단되었고, Bergey's manual of systematic bacteriology 등에 준하여 주요 생화 학적 특성을 검토한 결과 포자형성, catalase 음성, oxidase 양성, nitrate 환원력 양성으로 확인되었다. 균체의 지방산 조성 분석 결과 C15 ANTEISO 61.53%와 C17:0 ANTEISO 14.15% 로 이루어져 Bacillus sp.로 동정되었다. 16S rRNA 유전자 염기서열분석 결과 B. pseudofirmus와 99.7%의 유사성을 보였으나, Gelatin 분해능과 생장온도에서 차이를 보였다. 이상의 결과를 바탕으로 분리균주는 새로운 균주로 판단되 어, Bacillus sp. DK1122라 명명하고 Genbank에 균주를 등록하였다. 추후 식품 및 세제분야 등 산업으로 유용하게 이용될 수 있을 것으로 기대된다.









