Article
식물성 식품원료의 진위 판별을 위한 종 특이 프라이머의 개발
김규헌*, 김용상, 김미라, 이호연, 정유경, 이재황, 장혜숙, 박용춘1, 김상엽2, 최장덕, 장영미
Development of Species-Specific Primer to Determine the Authenticity of Vegetable Raw Materials in Food
Kyu-Heon Kim*, Yong-Sang Kim, Mi-Ra Kim, Ho-Yeon Lee, Yoo Kyung Jung, Jae-Hwang Lee, Hye-Sook Chang, Yong-Chjun Park1, Sang Yub Kim2, Jang Duck Choi, Young-Mi Jang
Author Information & Copyright ▼
식품의약품안전처 식품의약품안전평가원 신종유해물질팀
New Hazardous Substance Team, National Institute of Food and Drug Safety Evaluation, Ministry of Food and Drug Safety,
1Novel Food Division, Ministry of Food and Drug Safety
2Hazardous Substances Analysis Team, Busan Regional Food and Drug Administration
*Corresponding author: Kyu-Heon Kim, New Hazardous Substance Team, Food Safety Evaluation Department, National Institute of Food and Drug Safety Evaluation, Ministry of Food and Drug Safety, 187 Osongsaengmyeong2-ro, Osong-eup, Heungdeok-gu, Cheongju-si, Chungcheongbuk-do, 363-700, Korea Tel: +82-43-719-4453; Fax: +82-43-719-4450
khkim95@korea.kr
© Korean Society for Food Engineering. This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Received: Jul 10, 2014; Revised: Sep 29, 2014; Accepted: Oct 22, 2014
Abstract
In this study, a detection method was developed and optimized using a molecular biological technique to distinguish 21 vegetable food raw materials. The genes for the distinction of species in the raw materials were targeted at Internal Transcribed Spacer (ITS) in nucleus and the Maturase K (matK) gene in chloroplasts. The species-specific primers were designed for PCR products around 200 bp for their application to processed products. The amplicon size for the following: Winter mushrooms, Shiitakes, Button mushrooms, Lacquer tops, King oyster mushrooms, Oyster mushrooms, Chestnuts, Pine nuts, Walnuts, Aloe, Water dropwort, Scallions, Chinese chives, Cucumbers, Horseradish, Mustard, Mung beans, Red beans, Guarana, Korean dandelions, and Dandelions were confirmed at 166 bp ~ 245 bp, respectively. Non-specific PCR products were not detected in similar species by the species-specific primers. The detection method of raw materials developed in this study would be applied to food safety management for the eradication of adulterated food distribution and protection of consumers’ rights.
Keywords: Vegetable food raw material; PCR; Species-specific primer; Internal Transcribed Spacer; matK gene
서 론
일반적으로 가짜식품(Economically Motivated Adulteration, EMA)이란 경제적 이득을 취하기 위하여 원재료를 속이고, 의도적으로 저렴한 원료를 사용하는 등 부당한 방 법으로 제조된 식품을 의미한다. 최근 식품 원자재 값의 상승 및 경제성장과 산업화에 따른 식생활의 변화에 따라 가짜식품의 제조 및 유통이 크게 증가하여 소비자의 먹거 리 불안감은 점점 높아지고 있다. 가짜식품은 농어민과 정 상적인 식품 제조회사의 소득증대를 저하시키고 국민의 건 강을 위협할 뿐만 아니라 유통질서를 파괴시키는 주범으로 유럽에서는 European Union Regulation(EUR)을 규정하여 가짜식품으로부터 소비자를 보호하기 위한 식품안전과 이 력추적제를 추진하고 있고, 미국의 식품의약품청(FDA)과 영국식품기준청(FSA) 등에서도 늘어나고 있는 가짜식품에 대한 관리를 강화하고 있다. 이들과 마찬가지로 국가적인 차원에서 가짜식품의 유통을 근절시킬 수 있는 관리 감독 및 유통 체계를 마련하여야 하고 가공식품의 경우 사용원 료의 부정확한 표시나 제조 과정 중 원재료 외 다른 원료 들의 비의도적 혼입을 통제하기 위해서는 제품에 함유된 원료 물질을 모니터링 할 수 있는 효율적이고 정확한 분석 방법의 개발이 필요하다.
특히 이종 혼합식품이나 가짜식품 등을 가공하거나 처리 하였을 경우 외관이나 관능적 방법에 의한 원재료의 판별 이 거의 불가능하기 때문에 원재료의 정확한 표시 및 소비 자들의 신뢰성과 안전성 확보를 위하여 원재료 표시에 대 해 객관적으로 검증할 수 있는 과학적인 판별 기술 개발이 매우 중요하다. 현재 원재료 검증방법으로 광합성 중 대사 경로에 따른 탄소 동위원소 비율을 분석하는 방법과 분자 량이 작은 ion-fragment의 패턴 차이를 이용하여 비교분석 하는 MS-전자코 분석법(Padovan et al., 2003; Hong et al., 2011a,b), 그리고 원재료가 함유하고 있는 특정 성분 물질의 유무 및 함유량을 확인할 수 있는 크로마토그래피 (LC, GC, HPLC) 등을 포함하는 이화학적 분석법이 있고 (Schellenberg et al., 2010), 면역효소측정법과 등전점 전기 영동법으로 대표되는 단백질 분석법 그리고 분자생물학적 분석기술의 발달에 따라 DNA 분자 수준에서 염기서열 차 이에 기초한 PCR기법 등과 같은 유전자 분석법이 있다 (Folmer et al., 1994; Fabric et al., 2005). 하지만 이화학 적 분석법은 그 방법이 복잡하고 분석 기기가 고가이기 때 문에 쉽게 적용할 수 없고, 단백질 분석법은 동일 종 내에 서 단백질 발현이 유사하기 때문에 동종간 판별의 이용은 사실상 불가능할 뿐 아니라 열처리 과정을 거친 가공품인 경우 수용성 단백질 및 특이적 항원의 변성으로 감수성이 현저히 떨어지는 문제점이 있다. 최근에는 단백질에 비해 분석 방법이 간단하고 열에 비교적 안정한 유전자를 대상 으로 유전자 분석법을 사용하고 있다. 그 중 PCR을 이용 한 DNA 검출 방법은 민감성, 특이성이 매우 높을 뿐만 아니라 식품가공이나 제조 과정 중에서도 단백질에 비해 비교적 안정한 물질이므로 외부적 환경 요인에 큰 영향을 받지 않기 때문에 분석결과의 오차를 줄일 수 있다는 장점 을 가지고 있다(Hebert et al., 2003; Monaghan et al., 2003).
분자생물학 및 유전자 분석 기술의 발달로 종 특이적인 DNA 염기서열 차이에 따른 종 판별은 물론 동속 내에서 의 종 판별이 가능한 종 특이적 PCR 법이 이용되고 있다. 종의 특이적인 DNA를 증폭하기 위해서는 최적의 유전자 마커 선택(탐색)이 매우 중요하며, 최근에는 종 동정에 DNA barcoding marker를 활용하는 방법이 많이 연구되고 있다. DNA barcode라는 용어는 2003년 Hebert가 최초로 사용한 용어로 종 동정을 위한 최소한의 유전자를 의미하 며(Harmer et al., 1989), 이러한 DNA barcode 부위를 증 폭하기 위하여 일반적으로 보존력이 좋은 유전자를 선택하 고(White et al., 1990; Chase et al., 2007; Chen & Song, 2010), 이 유전자 부위를 증폭할 수 있는 일반프라이머 (universal primer)를 개발한다. 식물성은 모계 유전으로 인 해 유전적으로 비교적 안정적이고 유전자가 잘 보존되어 있는 엽록체에 존재하는 유전자 부위를 주로 이용한다. 그 중 Rubisco large subunit (rbcL) 및 Maturase K (matK) 등의 영역은 다른 영역에 비해 빠른 진화 속도를 보여 분 자계통분류학적으로 식물 종간 분류 및 종내 분류에 이용 되고 있다(Herbert et al., 2003). 또한 핵에 존재하는 Intergenic spacer (IGS) 및 Internal transcribed spacer (ITS) 영역은 다른 영역에 비해 빠른 진화 속도를 보여 종간, 종 내 및 집단 내에서 변이조사 등 DNA barcoding marker로 서 광범위하게 이용되고 있다(Kress et al., 2005; Kress & Erickson, 2007; Kress & Erickson, 2008; Lahaye et al., 2008)(Fig. 1).
Fig. 1.
Genes from three genomes in plants that are candidate barcodes. (Blue markers are potential barcodes, green markers are poor candidates and red markers are pending to be investigated.)
Download Original Figure
식품원료의 종 판별을 위한 DNA barcoding marker 유 전자 증폭에는 크게 두 가지로 구분할 수 있다. 첫 번째, 일반 프라이머(universal primer)를 이용하여 증폭 후 염기 서열을 결정하고 미국 국립보건원에서 운영하는 유전자은 행(www.ncbi. nlm.nih.gov/blast.cgi)에 등록되어 있는 염기 서열과 비교하여 종을 판별하는 방법(Kocher et al., 1989), 두 번째, 종 특이 프라이머(species-specific primer)를 개발 하여 유전자 증폭 유무로 종을 판별하는 방법이 있다 (Tomoaki et al., 2009). 특히, 가공식품은 제조 가공과정에 서 고압살균, 화학처리 및 발효작용 등을 거쳐야 하기 때 문에 식품 속의 DNA가 손상될 우려가 있는데 이를 효율 적으로 분석할 수 있는 기술 확보가 매우 중요하다. 일반 프라이머를 이용한 염기서열 분석은 PCR 증폭 산물의 크 기가 700 bp 이상이므로 가공식품에 적용하기가 어렵지만, PCR 증폭 산물의 크기를 200 bp 내외가 되도록 설계하고 이를 이용하여 증폭 여부로 종을 확인하는 방법이 활용된 다(Park et al., 2012a to 2012e; Park et al., 2013).
식품의약품안전처는 2010년부터 2012년까지 불량식품 근절을 위해 유전자 분석법을 이용하여 식품 중 동물성(축 산물 12종, 수산물 29종) 및 식물성(33종) 사용원료 74종에 대한 진위 판별법을 개발하여 지방식품의약품안전 청, 보건환경연구원, 시·도 지자체, 식품위생검사기관 및 산 업계 등에 보급하여 활용하고 있다. 본 연구에서는 2013년 도에 버섯류를 포함한 21종의 식물성 식품원료에 대한 진 위 판별을 위하여 종 특이 프라이머를 설계하고, PCR 조 건을 최적화하여 식품원료에 직접 적용 및 유효성을 확인 하였으며, 향후 식물성 식품원료를 포함한 가공식품에 대하 여 진위를 판별하는데 과학적인 근거를 제시하고자 하며, 불 량식품 근절에 적극 활용될 것으로 기대된다.
재료 및 방법
시료 선정
국내·외에서 기사화 된 불량식품의 유통 사례와 다소비 식품에 대한 정보를 토대로 대상종을 선정하였다. 대상종 은 총 21종으로 버섯류 6종[팽이버섯(Flammulina velutipes), 표고버섯(Lentinula edodes), 양송이버섯(Agaricus bisporus), 영지버섯(Pleurotus eryngii), 새송이버섯(Pleurotus ostreatus) 및 느타리버섯(Ganoderma lucidum)], 견과류 3종[밤(Castanea crenata Sieb), 잣(Pinus koraiensis) 및 호 두(Juglans regia)], 과실류 1종[대추(Ziziphus jujuba)], 채 소류 6종[알로에(Aloe vera), 미나리(Oenanthe javanica), 부추(Allium tuberosum), 오이(Cucumis sativus), 고추냉이 (Eutrema wasabi) 및 겨자(Brassica juncea)], 콩류 2종[녹 두(Vigna radiatus), 팥(Vigna angularis)] 및 기타 3종[과라 나(Paullinia pachycarpa), 흰민들레(Taraxacum coreanum) 및 민들레(Taraxacum platycarpum)]을 포함한다. 선정한 식 품원료와 유전적으로 근연종이거나, 식감 및 색이 유사하 여 상호간에 육안으로 구분이 어려운 식품군을 비교종으로 선정하였다.
시약 및 시료 구입
PCR을 위한 프라이머는 바이오니아(Bioneer, Korea)에 의뢰하여 합성하였으며, 유전자 증폭을 위해 Taq DNA polymerase 및 dNTPs, MgCl2은 TAKARA (TaKaRa Co., JAPAN)에서 구매하였고, PCR 장비는 C1000 Touch™ Thermal Cycler (Bio-Rad Laboratories, Inc. USA)을 사용 하였다. 실험에 사용된 시료는 청주 인근 대형마트 및 재 래시장에서 구입하여 사용하였다.
유전자 추출
식물성 식품원료에서의 유전자 추출은 DNeasy Plant mini kit (Qiagen GmbH, Hilden, Germany)를 사용하였고, 추출 방법은 제조사에서 제공하는 방법에 의거하여 추출하 였으며, 최종 용출단계에서는 멸균증류수를 사용하였다.
종 특이 프라이머 설계
종 특이 프라이머 설계를 위해 미국 국립보건원에서 운영 하는 유전자은행에 등록되어 있는 식물성 원료에 대한 유전 정보를 확인하였고, 엽록체에 존재하는 matK와 핵에 존재 하는 ITS 유전자 부위에 대한 염기서열을 대상으로 시료 간 비교·분석을 통해 종 특이 프라이머를 설계하였으며, 비교· 분석에는 BioEdit ver. 7.0.9.0 프로그램을 사용하였다.
PCR 반응 및 결과 확인
PCR을 위한 반응액의 조성은 추출한 주형 DNA 50~100 ng/μl, dNTPs 200 μM, MgCl2 2.0 mM 그리고 10 pM의 정방향 및 역방향 프라이머 각각 1 μl를 혼합하였 으며, 최종 용량은 20 μl가 되도록 증류수를 첨가하였다. 본 연구에서 개발된 종 특이 프라이머의 염기서열 및 검출 을 위한 최적의 PCR 반응조건은 Table 1에 나타내었다. 최종 산물의 확인은 반응액 5 μl를 취하여 EtBr이 첨가된 (1 μl/ml) 아가로즈 젤로 100V, 30분간 전기영동하였다. PCR 산물의 크기 확인은 100 bp DNA ladder(Bioneer, Korea)를 사용하였으며, 전기영동 완료 후 UV 투영기를 이용하여 결과를 확인하였다.
Table 1.
Information and PCR conditions of species-specific primers used in this study.
| Species |
Primer |
Primer Sequnce (5‘→3’) |
Length (bp) |
PCR condition |
Target region |
| Step |
Temp. |
Time |
Cycle |
|
Flammulina velutipes
|
SFI13-Enokitake-F |
ATGTAACGAATGTCATTGATTA |
210 |
1st denaturation |
94°C |
5 m |
1 |
ITS
|
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
55°C |
30 s |
| SFI13-Mushroom-R |
SFI13-Mushroom-R |
Extention |
72°C |
1 m |
|
| Elongation |
72°C |
7 m |
1 |
| Lentinula edodes |
SFI13-Shiitake-F |
ATAGAATGWTCTKGTTATTGGG |
210 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
55°C |
30 s |
| SFI13-Mushroom-R |
TGACACTCAAACAGGCATGC |
Extention |
72°C |
1 m |
|
| Elongation |
72°C |
7 m |
1 |
|
Agaricus bisporus
|
| SFI13-Agaricus-F |
CATGGGCTATGCCTATGAAA |
190 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
55°C |
30 s |
| SFI13-Mushroom-R |
TGACACTCAAACAGGCATGC |
Extention |
72°C |
1 m |
|
| Elongation |
72°C |
7 m |
1 |
|
Ganoderma lucidum
|
SFI13-Ganoderma-F |
TTGCGATGTAACACATCTATAT |
188 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
55°C |
30 s |
| SFI13-Mushroom-R |
TGACACTCAAACAGGCATGC |
Extention |
72°C |
1 m |
|
| Elongation |
72°C |
7 m |
1 |
|
Pleurotuseryngii
|
SFI13-King oyster-F |
CTCAAACTCACTCTGGTTTTTCC |
197 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
20 s |
35 |
|
Annealing |
65°C |
10 s |
| SFI13-King oyster-R |
GGTTAGAGAGCCAGACTCTATTC |
Extention |
72°C |
30 s |
|
| Elongation |
72°C |
7 m |
1 |
| Pleurotus ostreatus |
SFI13-oyster-F |
CTCAAACTCACTTTGGTTTCTTT |
206 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
62°C |
30 s |
| SFI13-oyster-R |
TTGCGGACGATTRGAGAGCTG |
Extention |
72°C |
1 m |
|
| Elongation |
72°C |
7 m |
1 |
|
Castanea crenata
|
| SFI13-Chestnut-F |
TAGAACATTTTGCCGAAGTC |
181 |
1st denaturation |
94°C |
5 m |
1 |
matK |
|
| Denaturation |
94°C |
20 s |
35 |
|
Annealing |
65°C |
10 s |
| SFI13-Chestnut-R |
CATTSCCATAAATTGACAAGG |
Extention |
72°C |
30 |
|
| Elongation |
72°C |
72°C |
1 |
|
Pinus koraiensis
|
SFI13-Pine nut-F |
TTCTCGTCGAAATTCACTGA |
211 |
1st denaturation |
94°C |
5 m |
1 |
matK |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
60°C |
30 s |
| SFI13-Pine nut-R |
GGATAATTGATGAGAACAGAC |
Extention |
72°C |
1 m |
|
| Elongation |
72°C |
7 m |
1- |
|
uglans regi
|
SFI13-Juglans-F |
CTTTTTGAGCGAATTTA |
245 |
1st denaturation |
94°C |
5 m |
1 |
matK |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
55°C |
30 s |
| SFI13-Juglans-R |
TATATCTAAGAAGGGTT |
Extention |
72°C |
1 m |
|
| Elongation |
72°C |
7 m |
1 |
| Aloe vera |
SFI13-Aloe-F |
AAGGACGACCGCGAACCATT |
175 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
62°C |
10 s |
| SFI13-Aloe-R |
CCTCCGACCGWTGTGTTCCTT |
Extention |
72°C |
45 s |
|
| Elongation |
72°C |
7 m |
1 |
|
Oenanthe javanica
|
| SFI13-Water parsley-F |
ACGACCCGCTAACGTGTAAAC |
193 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
62°C |
30 s |
| SFI13-Water parsley-R |
TACAYGAAGCGCGCGYGCATT |
Extention |
72°C |
1 m |
|
| Elongation |
72°C |
7 m |
1 |
|
Allium tuberosum
|
SFI13-Chives-F |
TATTGTTTAGAAGGGTTTCC |
235 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
58°C |
30 s |
| SFI13-Chives-R |
AAGTGTCGCATTTCGCTA |
Extention |
72°C |
1 m |
|
| Elongation |
72°C |
7 m |
1 |
|
Cucumis sativus
|
SFI13-Cucumber-F |
TCGTACCGGCCTCGGTGGTGC |
178 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
15 s |
35 |
|
Annealing |
60°C |
5 s |
| SFI13-King oyster-R |
TTCAAAGACTCGGTGGTTCAC |
Extention |
72°C |
30 s |
|
| Elongation |
72°C |
7 m |
1 |
| Eutrema wasabi |
SFI13-Wasabi-F |
GTYTYGGTYGGATCGTRCGCA |
166 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
15 s |
35 |
|
Annealing |
67°C |
10 s |
| SFI13-Wasabi-R |
TTGCCGAGAGTCGTTTTAGACT |
Extention |
72°C |
30 m |
|
| Elongation |
72°C |
7 m |
1 |
|
Brassica juncea
|
| SFI13-Mustard-F |
TATCTCGGCTGGGTCATGCGTG |
167 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
65°C |
15 s |
| SFI13-Mustard-R |
TTGCCGAGAGTCGTTTTAGACT |
Extention |
72°C |
45 s |
|
| Elongation |
72°C |
72°C |
1 |
|
Vigna radiatu
|
SFI13-Mung bean-F |
KSGCAACAACTTGTGTGA |
201 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
20 s |
35 |
|
Annealing |
65°C |
10 s |
| SFI13-Mushroom-R |
GATGCAAGAGCCGAGATA |
Extention |
72°C |
30 s |
|
| Elongation |
72°C |
7 m |
1 |
|
Vigna angularis
|
SFI13-Red bean-F |
TCACCTCTCCAGGCAAAAC |
202 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
62°C |
15 s |
| SFI13-King oyster-R |
ACACCAAGTATCGCATTT |
Extention |
72°C |
45 m |
|
| Elongation |
72°C |
7 m |
1 |
| Ziziphus jujuba |
SFI13-date-F |
AAACCGCGCCAAGGAACACCT |
196 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
62°C |
30 s |
| SFI13-Mushroom-R |
GATGGTTCACGGGATTCTGC |
Extention |
72°C |
1 m |
|
| Elongation |
72°C |
7 m |
1 |
|
Paullinia cupana
|
| SFI13-Guarana-F |
CATCGTTGCCCCCAAACCT |
219 |
1st denaturation |
94°C |
5 m |
1 |
ITS |
|
| Denaturation |
94°C |
30 s |
35 |
|
Annealing |
65°C |
15 s |
| SFI13-Guarana-R |
CTCCCAGCCCTCGGACCCTCT |
Extention |
72°C |
45 s |
|
| Elongation |
72°C |
7 m |
1 |
|
Taraxacum coreanum
|
SFI13-Platycarpum-F |
TCTTTTTTGAGGGCTTCCCTGG |
196 |
1st denaturation |
95°C |
5 m |
1 |
ITS |
|
| Denaturation |
95°C |
30 s |
35 |
|
Annealing |
60°C |
5 s |
| SFI13-Platycarpum-R |
CTGTGTGTCGTGAGCTGCA |
Extention |
72°C |
30 |
|
| Elongation |
72°C |
5 m |
1naza |
|
Taraxacum platycarpum
|
SFI13-Platycarpum-F |
CTGTGTGTCGTGAGCTGCA |
195 |
1st denaturation |
95°C |
5 m |
1 |
ITS |
|
| Denaturation |
95°C |
30 s |
35 |
|
Annealing |
60°C |
5 s |
| SFI13-Platycarpum-R |
GATTTTCAAGCTGGGCTAG |
Extention |
72°C |
30 |
|
| Elongation |
72°C |
5 m |
1 |
Download Excel Table
결과 및 고찰
본 연구에서는 버섯류를 포함한 21종의 대상시료에 대 하여 식품 중 식물성 사용원료 진위 판별을 위한 유전자 판별법을 개발하였다. 이를 위하여 유전자은행에 등록되어 있는 대상 시료 및 비교 시료의 ITS 또는 matK 유전자 부 위의 염기서열 데이터를 확보하고 비교 분석하여 종 특이 프라이머를 설계하였다. 먼저, 버섯류에 속하는 팽이버섯, 표고버섯, 양송이버섯, 새송이버섯, 느타리버섯 및 영지버 섯은 ITS 유전자 부위에서 종간 상동성이 비교적 높은 부 위에서 6종의 역방향 프라이머를 설계하였고, 정방향 프 라이머는 염기서열이 비교적 다양한 부위를 이용하여 각각 설계하였으며, 대상종과 비교종은 6종의 버섯류를 상호 교차 실험하였다. 견과류 중 밤은 고구마, 울금, 호두, 잣 및 도토리, 잣은 호두, 포도, 호박씨, 해바라기씨 및 땅콩, 호두는 잣, 아몬드, 땅콩 및 밤을 각각 비교종으로 하였으 며 matK 유전자 부위에서 비교종들과 염기서열이 상이한 부위를 프라이머로 설계하여 실험에 이용하였다. 과실류에 속하는 대추는 블루베리, 포도를 비교종으로 하여 ITS 유 전자 부위에서 프라이머를 설계하였고 채소류 중 알로에는 오이, 양파, 배추, 마늘, 호박, 수박 및 참외, 미나리는 배 추, 녹차, 시금치, 클로렐라 및 스피루리나, 부추는 파와 쪽 파, 오이는 알로에, 양파, 배추, 마늘, 호박, 수박 및 참외, 고추냉이는 후추, 겨자 및 무, 겨자는 후추, 고추냉이 및 무를 각각 비교종으로 하여 ITS 유전자 부위에서 비교종들 과 염기서열이 상이한 부위를 프라이머로 설계하였다. 콩 류 또한 ITS 유전자 부위에서 프라이머를 설계하였는데 녹 두는 팥, 옥수수, 고구마, 완두, 감자 및 밀, 팥은 녹두, 옥 수수, 고구마, 완두, 감자 및 밀을 비교종으로 하였다. 마지 막으로 건강식품의 원료로 많이 이용되는 과라나는 단풍나 무, 커피 및 카카오, 흰민들레와 민들레는 서양민들레를 비 교종으로 하여 ITS 유전자 부위에서 각각의 프라이머를 설 계하였다. 21종의 식물원료 프라이머는 가공식품에 대하 여 적용이 가능하도록 하기 위하여 가급적 200 bp 내외가 되도록 설계하였고, 이를 이용하여 선정된 식품원료에 대 한 PCR 조건을 최적화 하였다(Table 1).
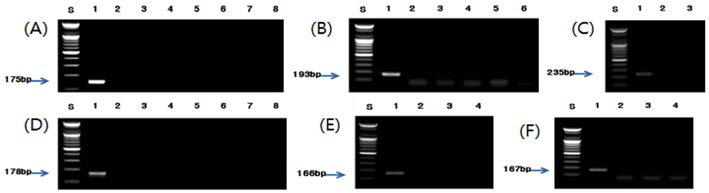
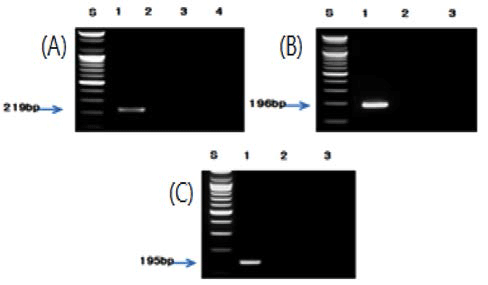
각각의 종 특이 프라이머를 사용하여 최적화 된 조건으 로 PCR을 수행한 결과, 버섯류인 팽이버섯, 표고버섯, 양 송이버섯, 영지버섯, 새송이버섯 및 느타리버섯은 각각 210, 210, 190, 188, 197 및 206 bp(Fig. 2), 견과류인 밤, 잣 및 호두는 각각 181, 211 및 245 bp(Fig. 3), 과실류인 대추는 196 bp(Fig. 4), 채소류인 알로에, 미나리, 부추, 오이, 고추 냉이 및 겨자는 각각 175, 193, 235, 178, 166 및 167 bp(Fig. 5), 두류인 녹두와 팥은 201 bp와 202 bp(Fig. 6), 그리고 과라나, 흰민들레 및 민들레에 대하여 각각 219, 196 및 195 bp(Fig. 7)의 예상된 크기의 증폭 산물을 확인 할 수 있었고, 선정된 대상종 이외의 비교종에서는 비 특 이적인 밴드는 생성되지 않았다.
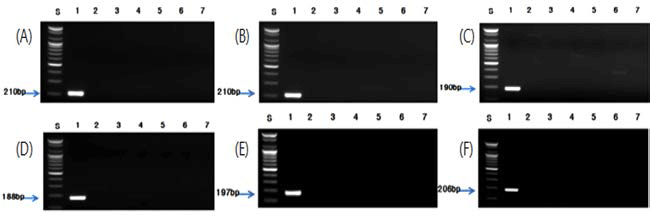
Fig. 2.
PCR results from various mushrooms by PCR using optimised species-specific primer. (A) Lane 1; Flammulina velutipes, Lane 2; Pleurotus ostreatus, Lane 3; Auricularia auricula-judae, Lane 4; Lentinula edodes, Lane 5; Ganoderma lucidum, Lane 6; Agaricus bisporus, Lane 7; Pleurotus eryngii. (B) Lane 1; Lentinula edodes, Lane 2; Pleurotus ostreatus, Lane 3; Auricularia auriculajudae, Lane 4; Flammulina velutipes, Lane 5; Ganoderma lucidum, Lane 6; Agaricus bisporus, Lane 7; Pleurotus eryngii. (C) Lane 1; Agaricus bisporus, Lane 2; Pleurotus ostreatus, Lane 3; Auricularia auricula-judae, Lane 4; Flammulina velutipes, Lane 5; Lentinula edodes, Lane 6; Ganoderma lucidum, Lane 7; Pleurotus eryngii. (D) Lane 1; Pleurotus eryngii, Lane 2; Pleurotus ostreatus, Lane 3; Auricularia auricula-judae, Lane 4; Flammulina velutipes, Lane 5; Lentinula edodes, Lane 6; Ganoderma lucidum, Lane 7; Agaricus bisporus. (E) Lane 1; Pleurotus ostreatus, Lane 2; Auricularia auricula-judae, Lane 3; Flammulina velutipes, Lane 4; Lentinula edodes, Lane 5; Ganoderma lucidum, Lane 6; Agaricus bisporus, Lane 7; Pleurotus eryngii. (F) Lane 1; Ganoderma lucidum, Lane 2; Pleurotus ostreatus, Lane 3; Auricularia auricula-judae, Lane 4; Flammulina velutipes, Lane 5; Lentinula edodes, Lane 6; Agaricus bisporus, Lane 7; Pleurotus eryngii.
Download Original Figure
Fig. 3.
PCR results from various nut seeds by PCR using optimised species-specific primer. (A) Lane 1; Castanea crenata Sieb, Lane 2; Ipomoea batatas, Lane 3; Curcuma longa, Lane 4; Juglans regia, Lane 5; Pinus koraiensis, Lane 6; Quercus acutissima Carr. (B) Lane 1; Pinus koraiensis, Lane 2; Juglans regia, Lane 3; Vitis vinifera L., Lane 4; Cucurbita moschata, Lane 5; Helianthus annus, Lane 6; Arachis hypogaea. (C) Lane 1; Juglans regia, Lane 2; Pinus koraiensis, Lane 3; Prunus dulcis, Lane 4; Arachis hypogaea, Lane 5; Castanea crenata Sieb.
Download Original Figure
Fig. 4.
PCR results from fruits by PCR using optimised speciesspecific primer. Lane 1; Ziziphus jujuba, Lane 2; Vaccinium corymbosum, Lane 3; Vitis vinifera L.
Download Original Figure
Fig. 5.
PCR results from various vegetables by PCR using optimised species-specific primer. (A) Lane 1; Aloe vera, Lane 2; Cucumis sativus, Lane 3; Allium cepa, Lane 4; Brassica campestris, Lane 5; Allium scorodorpasum, Lane 6; Cucurbita moschata, Lane 7; Citrullus lanatus, Lane 8; Cucumis melo var. makuwa. (B) Lane 1; Oenanthe javanica, Lane 2; Brassica campestris, Lane 3; Camellia cinensis, Lane 4; Spinacia oleracea, Lane 5; Chlorella, Lane 6; Spirulina platensis. (C) Lane 1; Allium tuberosum, Lane 2; Allium fistulosum L., Lane 3; Allium ascalonicum. (D) Lane 1; Cucumis sativus, Lane 2; Aloe vera, Lane 3; Allium cepa, Lane 4; Brassica campestris, Lane 5; Allium scorodorpasum, Lane 6; Cucurbita moschata, Lane 7; Citrullus lanatus, Lane 8; Cucumis melo var. makuwa. (E) Lane 1; Eutrema wasabi, Lane 2; Piper longum, Lane 3; Brassica juncea, Lane 4; Raphanus sativus. (F) Lane 1; Brassica juncea, Lane 2; Piper longum, Lane 3; Eutrema wasabi, Lane 4; Raphanus sativus.
Download Original Figure
Fig. 6.
PCR results from various pulse crops by PCR using optimised species-specific primer. (A) Lane 1; Vigna radiatus L., Lane 2; Vigna angularis, Lane 3; Zea may, Lane 4; Ipomea batatas, Lane 5; Pisum sativum, Lane 6; Solanum tuberosum, Lane 7; Triticum aestivum. (B) Lane 1; Vigna angularis, Lane 2; Vigna radiatus L., Lane 3; Zea may, Lane 4; Ipomea batatas, Lane 5; Pisum sativum, Lane 6; Solanum tuberosum, Lane 7; Triticum aestivum.
Download Original Figure
Fig. 7.
PCR results from guarana and dandelion by PCR using optimised species-specific primer. (A) Lane 1; Paullinia pachycarpa, Lane 2; Acer palmatum, Lane 3; Coffea arabica, Lane 4; Theobroma cacao. (B) Lane 1; Taraxacum coreanum, Lane 2; Taraxacum platycarpum, Lane 3; Taraxacum officinale. (C) Lane 1; Taraxacum platycarpum, Lane 2; Taraxacum coreanum, Lane 3; Taraxacum officinale.
Download Original Figure
따라서 본 연구에서 개발된 21종의 식물성 식품원료에 대한 종 특이 프라이머는 육안 확인이 어려운 가공식품 등 을 대상으로 간단하고 정확하게 사용원료의 진위 여부를 확인할 수 있으며, 일반 프라이머가 가지고 있는 식품 적 용 한계를 극복할 수 있을 것이라고 판단하였다. 하지만 대상종에 대한 비교종의 선택에 있어 색, 식감 등에 기준 을 두었지만 비교종과의 유전적 의연관계를 추가적으로 확 인할 필요가 있다. 현재까지 개발된 판별법을 활용하여 건 전한 식품제조업체를 보호하고 정확한 표시사항 준수를 통 한 소비자 보호와 불량식품 근절에 적극 활용될 수 있을 것으로 사료된다.
요 약
본 연구에서는 식품 중 식물성 식품원료의 진위 판별을 위하여 분자생물학적 기법을 이용한 판별법을 개발하였다.
종 판별을 위한 유전자로 엽록체에 존재하는 matK 유전자 와 핵 내에 존재하는 ITS 유전자 부분을 대상으로 하였으 며, 가공식품에의 적용을 고려하여 PCR 산물의 크기는 200 bp 내외가 되도록 종 특이 프라이머(species-specific primer)를 설계하였다. 대상종으로는 버섯류 6종(팽이버섯, 표고버섯, 양송이버섯, 영지버섯, 새송이버섯 및 느타리버 섯), 견과류 3종(밤, 잣 및 호두), 과실류 1종(대추), 채소 류 6종(알로에, 미나리, 부추, 오이, 고추냉이 및 겨자), 콩 류 2종(녹두, 팥) 및 기타 3종(과라나, 흰민들레 및 민들 레), 총 21종을 선정하였으며, 종 특이 프라이머를 이용하 여 예상되는 PCR 산물의 생성 유무를 확인하였다. PCR 분석 결과, 21종의 식물성 식품원료에 대하여 각각 예상 된 PCR 산물을 확인하였으며, 프라이머별로 비교종에서 비 특이적 PCR 산물(non-specific PCR product)이 생성되 지 않음을 확인하였다. 본 연구에서 개발된 종 특이 프라 이머는 가열 및 가공된 식품 중 21종의 식물성 식품원료 의 진위 판별에 이용될 것이며, 불량식품 근절에 적극 활 용될 것으로 기대된다.
감사의 글
본 연구는 식품의약품안전평가원 2013년도 연구개발사 업지원비(13161소비자102)에 의해 수행되었으며 이에 감사 드립니다.
REFERENCES
Chase MW, Cowan RS, Hollingsworth PM, Van den Berg C, Madrinan S. A proposal for a standardised protocol to barcode all land plants. Taxon. 2007; 56:295-299.

Chen S, Song J. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species. PLoS One. 2010; 5-e8613.

Choi SJ, Lee YS, Kim JK, Lim SS. Physiological activities of extract from edible mushrooms. J. Korean Soc. Food Sci. Nutr. 2010; 39:1087-1096.

Choi YH, Kim MJ, Lee HS, Yun BS, Hu C, Kwak SS. Antioxidative compounds in aerial parts of Potentilla fragariodes. Korean J. Pharmacogn. 1998; 29:79-8.

Fabric T, Celia M, Catherine H. Food and forensic molecular identification: update and challenges. Trends Biotechnol. 2005; 23:359-366.

Folmer O, Black M, Hoeh W, Lutz R, Vrijenhoek R. DNA primers for amplification of mitochondrial cytochrome C oxidase subunit from diverse metazoan invertebrates. Mol. Mar. Bio. Biotechnol. 1994; 3:294-299.

Harmer JE, Farrall L, Ortach MJ, Valent B, Chunmley FG. Host species-specific conservation of a family of repeated sequence in the genomic of a fungal plant pathogen. Proc. Natl. Acad. Sci. U.S.A. 1989; 86:9981-9985.

Hebert PDN, Cywinska A, Shelley LB, Jeremy R. Biological identifications through DNA Barcodes. Proc. R. Soc. Lond. B. 2003; 270:313-321.

Herbert PDN, Retnasingham S, DeWaard JR. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species. Proc. R. Soc. Lond. B. 2003; 270:S96-S99.

Hong EJ, Kim KH, Park SJ, Kang JW, Kim DS, Lee HJ, Kim EJ, Lee JH, Lee KH, Noh BS. Discrimination of shreds of frozen and dried alaska pollack, dried pollack and cod using electronic nose. Food Eng. Progress. 2011a; 15:162-168.

Hong EJ, Kim KH, Park SJ, Kang JW, Kim DS, Lee HJ, Kim EJ, Lee JW, Kim SH, Lee KH, Noh BS. Discrimination of the salted cuttle fish and the salted mitra squid in economically motivated authentication food using electronic nose based on mass spectrometer. Food Eng. Progess. 2011b; 15:122-129.

Kocher TD, Thomas WK, Meyer A, Edwards SV, Paabo S, Vellablanca FX. Dynamics of mitochondrial DNA evolution in animals: amplification and sequencing with conserved primers. Proc. Natl. Acad. Sci. U.S.A. 1989; 86:6196-6200.

Kress WJ, Erickson DL. A two-locus global DNA barcode for land plants: the coding rbcL gene complements the noncoding trnH-psbA spacer region. PLoS ONE. 2007; 2-e508.

Kress WJ, Erickson DL. DNA barcodes : genes, genomics, and bioinformatics. Proc. Natl. Acad. Sci. U.S.A. 2008; 105:2761-2762.

Kress WJ, Wurdack KJ, Zimmer EA, Weigt LA, Janzen DH. Use of DNA barcodes to identify flowering plants. Proc. Natl. Acad. Sci. U.S.A. 2005; 102:8369-8374.

Lahaye R, Van der Bank M, Bogarin D, Warner J, Pupulin F. DNA barcoding the floras of biodiversity hotspots. Proc. Natl. Acad. Sci. U.S.A. 2008; 105:2923-2928.

Maua JL, Lina HC, Song SF. Antioxidant properties of several specialty mushrooms. Food Res. Int. 2002; 35:519-526.

Monaghan MT, Michael B, Gregory TR, Vogler AP. DNA-based species delineation in tropical beetles using mitochondrial and nuclear markers. Philos. Trans R. Soc. Lond. B. 2003; 270:313-321.

Padovan GJ, De Jong D, Rodrigues LP, Marchini JS. Detection of adulteration of commercial honey samples by the 13C/12C isotope ratio. Food Chem. 2003; 82:633-636.

Park YC, Jin SO, Lim JY, Kim KH, Lee JH, Cho TY, Lee HJ, Han SB, Lee SJ, Lee KH, Yoon HS. Application for identification of food raw materials by PCR using universal primer. J. Fd. Hyg. Safety. 2012a; 27:317-324.

Park YC, Lim JY, Kim MR, Park YE, Lim JD, Hwang CR, Kim KH, Lee JH, Cho TY, Lee HJ, Lee SJ, Han SB. Identification of faulty red pepper powder containing seasoned red pepper sauce. J. Fd. Hyg. Safety. 2012b; 27:182-187.

Park YC, Kim MR, Lim JY, Park YE, Shin JH, Hwang CR, Lim JD, Kim KH, Lee JH, Cho TY, Lee HJ, Han SB. A comparison of gene extraction methods for the Identification of raw materials from meat products. J. Fd. Hyg. Safety. 2012c; 27:146-151.

Park YC, Ahn CY, Jin SO, Lim JY, Kim KH, Lee JH, Cho TY, Lee HJ, Park KS, Yoon HS. Identification of Raw Materials in Processed Meat Products by PCR Using Species-Specific Primer. J. Fd. Hyg. Safety. 2012d; 27:68-73.

Park YC, Jin SO, Kim MR, Kim KH, Lee JH, Cho TY, Lee HJ, Han SB. Detection Method for Identification of Pueraria mirifica (Thai kudzu) in Processed Foods. J. Fd. Hyg. Safety. 2012e; 27:466-472.

Park YC, Kim MR, Jung YH, Shin JH, Kim KH, Lee JH, Cho TY, Lee HJ, Han SB. Development of Detection Method for Oilfish (Ruvettus pretiosus and Lepidocybirium flavobrunneum) as a Food Materials not Usable in Foods. J. Fd. Hyg. Safety. 2013; 28:50-55.

Schellenberg A, Chmielus S, Schlicht C, Camin F, Perini M, Bontempo L, Heinrich K, Kelly SD, Rossmann A, Thomas F, Jamin E, Horacek M. Multielement stable isotope ratio (H, C, N, S) of honey from different european regions. Food Chem. 2010; 121:770-777.

Tomoaki M, Atsushi A, Takuma T, Sumitaka Y, Yutaka O, Yoshida M. Identification of animal species using the partial sequences in the mitochondrial 16S rRNA Gene. Legal Med. 2009; 11:449-450.

White JJ, Bruns J, Lee S, Taylor J. Amplification and direct sequencing of fungus ribosomal RNA genes for phylogenics. A guide to methods and application. 1990; Academic press. p. 315-322.

Yanga JH, Lina HC, Mau JL. Antioxidant properties of several commercial mushroom. Food Chem. 2002; 77:229-23.