Article
Pediococcus pentosaceus의 Multilocus Sequence Typing 분석에서 신규 Sequence Type의 확인
이설희, 박영서*
Identification of New Sequence Types for Multilocus Sequence Typing of Pediococcus pentosaceus
Sulhee Lee, Young-Seo Park*
Department of Food Science and Biotechnology, Gachon University
*Corresponding author: Young-Seo Park, Department of Food Science and Biotechnology, Gachon University, Seongnam 13120, Korea Tel: +82-31-750-5378, Fax: +82-31-750-5273
ypark@gachon.ac.kr
© Korean Society for Food Engineering All rights reserved. This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Received: Nov 9, 2015; Revised: Nov 15, 2015; Accepted: Nov 16, 2015
Abstract
The genus Pediococcus belongs to the lactic acid bacteria and includes 15 species which are used in the food industry as both starter and probiotic cultures. The importance of Pediococcus spp. is due to their use as starter cultures in fermented meat as well as to their presence as the natural microbiota in vegetables. The availability of P. pentosaceus in the food industry increases the need for reliable molecular techniques for strain identification. To date, the reliable molecular methods for definite identification at strain level of microorganisms used in food industry has not been developed. Molecular identification based on suitable marker genes could be a promising alternative to conventional molecular typing methods such as ribotyping. In this study, the applicability of seven housekeeping genes gyrB, pyc, pgm, leuS, glnA, and dalR in combination with the pgi gene in multilocus sequence typing of P. pentosaceus was assessed. Sequencing and comparative analysis of sequence data were performed on 6 strains isolated from various vegetables. In addition to 17 sequence types, two new sequence types were identified and these fortified sequence types and seven marker genes allowed for a clear differentiation of the strains analyzed, indicating their applicability in molecular typing.
Keywords: multilocus sequence typing; Pediococcus pentosaceus; DNA fingerprinting; sequence type
서 론
현재까지 박테리아를 strain 수준으로 동정 또는 분류하기 위한 분자 수준의 typing 기술이 여러 가지 개발되어 왔는 데, 이러한 기술들 중에서 가장 강력한 기술은 유전자 지문 분석법(DNA fingerprinting) 기술로서, 여기에는 절단 빈도 가 낮은 제한효소를 이용한 pulsed-field gel electrophoresis (PFGE) (Olson, 1989), ribotyping (Kostman et al., 1992), randomly amplified polymorphic DNA (RAPD) (O’Sullivan, 1999), amplified fragment length polymorphism (AFLP) (Vos et al., 1995), multilocus sequence typing (MLST) (Stackebrandt et al., 2002; Gevers et al., 2005) 등이 있다. 이러한 기술들은 발효식품이나 인간 장내에서 분리된 유산 균이나 bifidobacteria를 종 이하의 수준에서 typing하는 데 광범위하게 사용되어 왔는데(McCartney, 2002), 각각의 기 술들은 분류 범위, 재현성, 해석의 용이성, 표준화 정도가 서로 다르며, 분석 기술의 정밀성과 정확성도 실험 방법, 실험실, 실험 재료에 따라서 매우 차이가 난다. 이 중에서 ribotyping, PFGE, polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE) (Muyzer et al., 1993), t-restriction fragment length polymorphism (t-RFLP) (Marsh, 1999; Dunbar et al., 2001) 등은 재현성은 높지만 고가의 장비와 많은 분석 시간이 요구되고 분석과정이 복 잡하여 고도의 숙련도가 필요하기 때문에 일반 실험실에서 는 분석이 어려운 단점이 있다. 반면에 repetitive-PCR (rep-PCR), RAPD, AFLP 등은 조작이 비교적 간단하고 신 속하지만 재현성이 낮은 단점이 있다(Higgins et al., 1982; William et al., 1993; Gancheva et al., 1999; Ventura & Zink, 2002; Healy et al., 2005).
최근 특정 균주의 유래나 이동경로를 추적하기 위해 PFGE나 PCR 등의 band-based typing 방법의 단점을 보 완할 수 있는 multilocus sequence typing (MLST) 방법이 고안되어 사용되고 있다(Stackebrandt et al., 2002; Gevers et al., 2001). MLST 방법은 염색체 DNA에서 약 6-8개의 housekeeping 유전자의 염기서열을 결정하고 MLST database에 입력한 후 대립유전자 프로파일들을 조 합하여 sequence type (ST)을 결정하는 방법이다(Homan et al., 2002).
Pediococcus sp.는 정상발효하는 유산균으로서 사워크라 우트와 같은 채소발효에 사용되고, 일부 균주는 프로바이 오틱스로 사용되어 치즈나 요구르트 제조에 사용되는 식품 발효에 중요한 유산균이다. 따라서 본 연구에서는 대표적 인 Pediococcus sp.인 P. pentosaceus를 다양한 분리원으로 부터 분리한 후, MLST 분석을 수행하여 P. pentosaceus의 분류학적 다양성을 확인하고자 하였다.
재료 및 방법
사용균주
본 연구에 사용된 P. pentosaceus는 한국프로바이오틱스 은행(Seongnam, Korea)에서 총 12주를 분양받았으며, 이 균주들의 분리원은 ON-82A, ON-188은 양파, GO-005, GO-008은 파, SC27, SC12는 젓갈, RP-008, RP-003은 고 추, TA-027, TA-035는 귤, 그리고 SP-019, SP-026은 고구 마로 총 6종류의 분리원을 사용하였다.
Genomic DNA 분리
P. pentosaceus로부터 genomic DNA를 분리하기 위하여 P. pentosaceus의 단일 집락을 Lactobacilli MRS 액체배지 (Difco, Detroit, MI, USA)에 접종하여 37°C에서 16시간 정치배양하였다. 배양액 3 mL을 16,100×g에서 1분간 원심 분리하여 균체를 수획하고, 상등액을 제거하였다. 이후 균 체에 1 mL의 TEN 완충용액(10 mM Tris-HCl, pH 7.6, 1 mM EDTA, 10 mM NaCl)을 첨가하여 현탁하고, 원심분리 하여 배지성분을 제거하였다. SET 완충용액(20% sucrose, 50 mM Tris-HCl, pH 7.6, 50 mM EDTA) 500 μL를 첨가하 여 현탁하고, 50 μL의 lysozyme (60 mg/mL in TEN 완충 용액, w/v)과 3 μL의 RNase A용액을 첨가하여 37°C에서 1시간동안 반응시켰다. 이 반응 용액에 25 μL의 SDS (25%, w/v)와 20 μL의 proteinase K를 첨가하여 조심스럽 게 혼합하고, 37°C에서 30분간 반응시켰다. 반응 종료 후, AccuPrep Genomic DNA Extraction kit (Bioneer, Daejeon, Korea) 내의 GC binding buffer 400 μL를 첨가하여 60°C 에서 10분간 반응시키고, 200 μL의 iso-propanol을 첨가하 여 조심스럽게 혼합하였다. 반응액을 column tube에 옮겨 서 원심분리를 통하여 DNA를 column에 부착시키고, 500 μL의 washing buffer 1과 washing buffer 2를 순차적으로 첨가하여 순수하게 DNA만을 남긴 후, 50 μL의 멸균 3차 증류수로 DNA를 용출하였다.
MLST 분석
P. pentosaceus의 MLST 분석을 위한 PCR은 상기 조건 으로 분리된 genomic DNA를 template DNA로 사용하였 고, Table 1에 나타낸 7개의 marker 유전자(gyr, pyc, pgm, leuS, gln, dal, pgi)를 증폭시키기 위한 primer를 제작하여 PCR을 수행하였다. PCR은 AccuPrep PreMix (Bioneer, Daejeon, Korea)를 사용하여 최종 부피가 20 μL가 되도록 하였고, My Cycler (BIO-RAD Laboratories, Hercules, CA, USA)로 반응시켰으며, PCR 조건은 94°C에서 2분동안 1 cycle, 95°C에서 20초, 56°C에서 30초, 72°C에서 30초 조 건에서 35 cycle, 그리고 마지막 단계로 72°C에서 7분간 반 응시켰다. PCR 산물의 염기서열 결정은 Macrogen사(Seoul, Korea)에 의뢰하여 ABI PRISM 3700 DNA Analyzer를 이용하여 수행하였다. 분석된 염기서열의 homology 분석은 BioEdit program (Tom Hall Ibis Biosciences, Carlsbad, CA, USA)을 이용하였고, 염기서열의 상동성은 Clustal X (http:/ /www.clustal.org/clustal2)와 Mega 5 program (Center for Evolutionary Functional Genomics, The Biodesign Institute, Tempe, AZ, USA)에 의해 phylogenetic tree를 작성하였다.
Table 1.
MLST primers for Pediococcus pentosaceus
|
Gene |
Primers |
Protein |
Sequence (5’ → 3’) |
Alleles |
Standard length (bp) |
|
gyrB
|
gyrB_F |
Gyrase B |
AGGGTGATTCTGCCGGTGG |
6 |
462 |
|
gyrB_R |
ACCTTGTCACCCATCAACAT |
|
pyc
|
pyc_F |
Pyruvate carboxylase |
AACAATTGCGGGCTAAAATG |
9 |
603 |
|
pyc_R |
GGCGCAGGTTGGTATACTGT |
|
pgm
|
pgm_F |
Phosphoglucomutase |
TTACGAAGAAAATGGCGAC |
8 |
321 |
|
pgm_R |
AATTTCAAAACATCCGCTTT |
|
leuS
|
leuS_F |
Leucyl-rRNA synthetase |
GGGTGGAACGGTAGTTGCTA |
10 |
522 |
|
leuS_R |
GCGAGAGAAGACCCAGTCAC |
|
glnA
|
glnA_F |
Glutamine synthetase |
AATCAATGGTTCAGGGATGC |
5 |
495 |
|
glnA_R |
TTGACGGTAAGAAGCCCATTC |
|
dalR
|
dalR_ F |
Glycil-tRNA synthetase |
TGTCGAACTGGCTAATGCTG |
10 |
570 |
|
dalR_ R |
CATGTAATGTTCGCGAATGG |
|
pgi
|
pgi_F |
Glucose-6-phosphate |
ATCGGTGACCGTGACTTCTC |
9 |
417 |
|
pgi_R |
isomerase |
ATCAAATTGCGACGACCTTC |
Download Excel Table
결과 및 고찰
Pediococcus pentosaceus의 MLST 분석
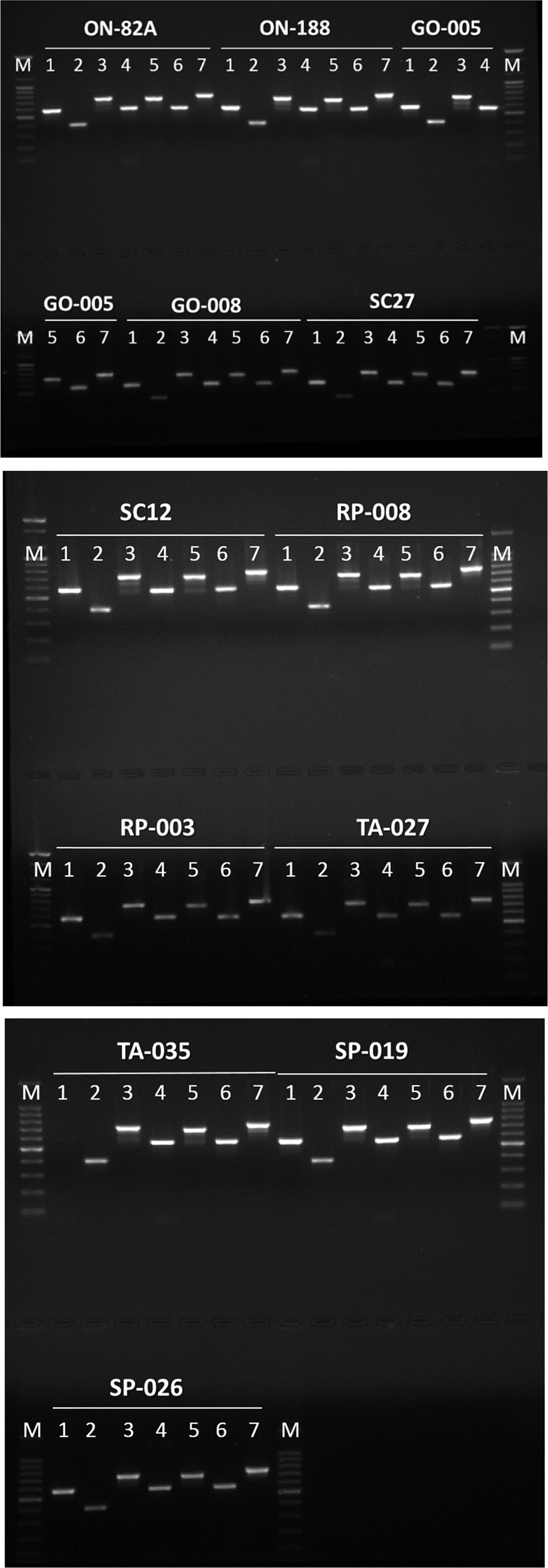
P. pentosaceus의 MLST 분석을 위한 primer와 각각의 대립 유전자들을 Table 1에 나타내었다. MLST 분석을 위 한 marker 유전자로는 gyrB, pyc, pgm, leuS, glnA, dalR, pgi 등 총 7개를 사용하였는데, 각각의 marker 유전자들은 5-10개의 대립 유전자들을 지니고 있었다(PubMLST, 2015). 분양받은 12개의 균주에 대한 MLST marker 유전 자들을 증폭하여 agarose gel 전기영동한 결과는 Fig. 1에 나타내었다. 사용된 12균주 각각에 대하여 모든 marker 유 전자들이 증폭이 되어 전기영동 상에 나타났으나, pgm 유 전자를 제외한 6개의 marker 유전자의 크기는 PubMLST database (PubMLST, 2015) 상의 크기와는 상이한 것으로 나타났다. 그러나 이들 12균주 중에서 6균주(ON-82A, ON-188, GO-005, GO-008, RP-003, RP-008)를 선정하여 marker 유전자들에 대한 염기서열을 분석한 결과 증폭된 유전자는 해당 marker 유전자인 것으로 판명되어 MLST 분석에 이용하였다.
Fig. 1.
Agarose gel electrophoresis of MLST for P. pentosaceus strains.
M, 100 bp DNA ladder; 1, Pgi; 2, Pgm; 3, Pyc; 4, GyrB; 5, DalR; 6, GlnA; 7, LeuS.
Download Original Figure
MLST sequence type의 분석
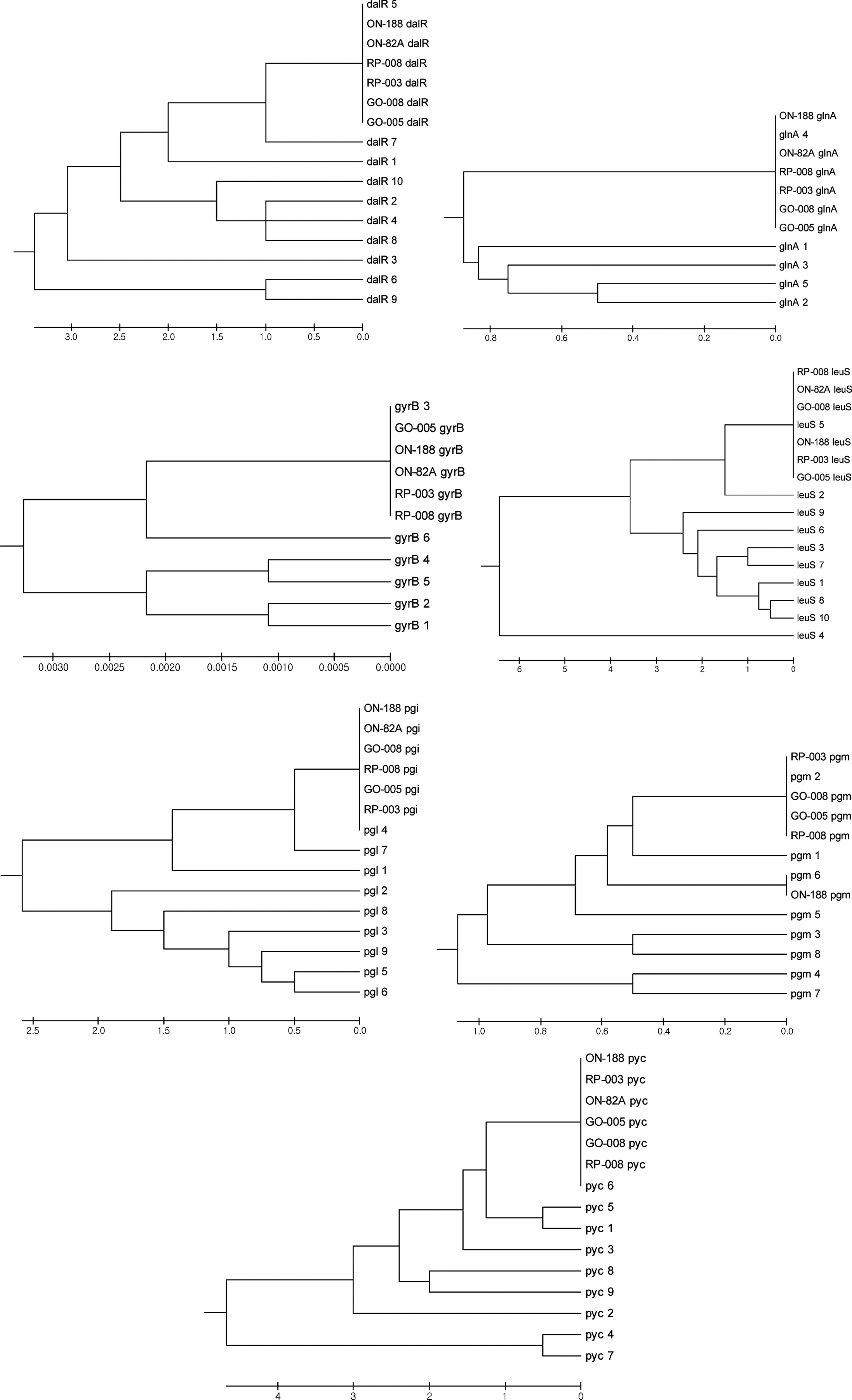
분석에 사용된 6균주의 MLST marker 유전자의 대립 유 전자 종류를 결정하기 위하여 해당 균주의 marker 유전자 와 현재까지 알려진 대립 유전자들의 염기서열을 이용하여 phylogenetic tree를 작성한 결과는 Fig. 2에 나타내었고, 이 결과에 따라 결정된 sequence type을 Table 2에 정리하였 다. 분석된 6균주 모두는 dalR 유전자는 5번 대립 유전자, glnA 유전자는 4번 대립 유전자, gyrB 유전자는 3번 대립 유전자, leuS 유전자는 5번 대립 유전자, pgi 유전자는 4번 대립 유전자, pyc 유전자는 6번 대립 유전자를 동일하게 지니고 있었으나, pgm 유전자의 경우에는 ON-188 균주의 경우 6번 대립 유전자를 지니고 있었고, 다른 균주들은 2번 대립 유전자를 지니고 있었는데 이를 이용하여 sequence type을 결정한 결과 기존의 sequence type과 일치하지 않는 새로운 sequence type으로 확인되었다. 따라서 ON-188 균 주의 sequence type을 18번으로, 나머지 균주들의 sequence type을 19번으로 지정하였다(PubMLST, 2015).
Table 2.
Sequence types of MLST for P. pentosaceus strains
|
Strain |
Allele type |
Sequence Type |
|
dalR
|
glnA
|
gyrB
|
leuS
|
pgi
|
pgm
|
pyc
|
|
ON-188 |
5 |
4 |
3 |
5 |
4 |
6 |
6 |
18 |
|
ON-82A |
5 |
4 |
3 |
5 |
4 |
2 |
6 |
19 |
|
SP-008 |
5 |
4 |
3 |
5 |
4 |
2 |
6 |
19 |
|
SP-003 |
5 |
4 |
3 |
5 |
4 |
2 |
6 |
19 |
|
GO-008 |
5 |
4 |
3 |
5 |
4 |
2 |
6 |
19 |
|
GO-005 |
5 |
4 |
3 |
5 |
4 |
2 |
6 |
19 |
Download Excel Table
Martino et al. (2013)은 여러 종류의 분리원으로부터 29주의 P. pentosaceus 균주를 분리하여 표현형적 특성과 bacteriocin 생산 여부를 조사하였고, MLST 분석을 통하여 17개의 sequence type을 확인하여 gyrB 유전자는 6개, pyc 유전자는 9개, pgm 유전자는 8개, leuS 유전자는 10개, glnA 유전자는 5개, dalR 유전자는 10개, pgi 유전자는 9 개의 대립 유전자가 존재함을 보고하였다. 또한 sequence type간의 관계를 조사하여 MLST 분석에 사용된 7개의 target 유전자는 유전자 내 재조합이 발생하지 않았지만 유 전자간 재조합이 광범위하게 발생되었음을 확인하였고, sequence type과 분리원간에는 높은 상관관계가 있어 같은 유전적 계통에 속하는 sequence type은 동일한 분리원으로 부터 유래됨을 밝힌 바 있다.
본 연구에서 새롭게 밝힌 sequence type 18은 dalR 유전 자의 5번 대립유전자, glnA 유전자의 4번 대립유전자, gyrB 유전자의 3번 대립유전자, leuS 유전자의 5번 대립유 전자, pgi 유전자의 4번 대립유전자, pgm 유전자의 6번 대 립유전자, pyc 유전자의 6번 대립유전자가 존재하는 것으 로, sequence type 18인 균주는 양파에서 분리된 ON-188 균주 밖에 존재하지 않아 sequence type과 분리원과의 상 관관계를 분석할 수 없었다. Sequence type 19는 dalR 유 전자의 5번 대립유전자, glnA 유전자의 4번 대립유전자, gyrB 유전자의 3번 대립유전자, leuS 유전자의 5번 대립유 전자, pgi 유전자의 4번 대립유전자, pgm 유전자의 2번 대 립유전자, pyc 유전자의 6번 대립유전자가 존재하는 것으 로, 총 5균주 중에서 1균주는 양파로부터, 2균주는 고구마 로부터, 나머지 2균주는 파로부터 분리된 것으로 다양한 분리원에 존재하는 것으로 나타났다.
현재까지 P. pentosaceus 균주의 MLST 분석은 Martino et al. (2013)이 수행하여 17개의 sequence type을 확인한 것을 제외하면 본 연구가 유일하며, 본 연구를 통하여 17 개의 sequence type 이외에 추가로 2개의 sequence type이 존재한다는 것을 밝힘으로써 P. pentosaceus의 분류 및 추 적을 위한 유전자 지문의 다양성을 확인할 수 있었다. P. pentosaceus 이외의 다른 유산균에 대한 MLST 분석의 최 근 연구들을 보면, Sun et al. (2015)은 중국과 몽골의 발 효 유제품으로부터 분리한 L. helveticus의 population structure를 MLST 분석을 기반으로 한 연구에서 총 11개 의 유전자를 분석한 바 있다.
본 연구결과로 MLST 분석법은 매우 간단하고 신속한 방법으로 미생물 분류 및 추적을 위한 유용한 유전자 지문 법인 것으로 확인되었으며, P. pentosaceus의 경우 6균주를 대상으로 MLST 분석을 한 결과 지금까지 알려져 있던 17 개의 sequence type 이외에 추가로 2개의 sequence type이 밝혀진 바, 보다 많은 균주를 대상으로 MLST 분석을 할 경우 아직까지 밝혀져 있지 않은 sequence type을 확인할 수 있을 것으로 판단된다.
요 약
Pediococcus 속은 유산균의 한 속(genus)으로 15종(species) 이 존재하며 식품산업에서 스타터 균주 또는 프로바이오틱 스 균주로 사용된다. Pediococcus 속은 발효육에서 스타터 균주로 사용이 되며, 채소에 존재하는 균총의 일부로 존재 하는 중요한 균주이다. Pediococcus pentosaceus의 식품산 업에서의 이용성이 증대됨에 따라 이들을 동정 및 분류할 수 있는 분자유전학적 기술이 요구되고 있다. 지금까지 식 품산업에서 미생물을 균주 수준에서 명확하게 식별할 수 있 는 신뢰할만한 방법이 개발되지 않고 있다. 적당한 marker 유전자를 이용한 분자유전학적 동정 방법이 ribotyping과 같은 전통적인 분자 typing을 대체할 수 있는 대안으로 여 겨진다. 본 연구에서는 gyrB, pyc, pgm, leuS, glnA, dalR 등의 7개의 필수 유전자를 사용하여 P. pentosaceus의 multilocus sequence typing을 분석하였다. 다양한 식품으로 부터 분리된 6균주에 대한 marker 유전자의 염기서열을 이용하여 분석에 사용하였다. 그 결과 기존에 보고된 17개 의 sequence type과는 상이한 2개의 sequence type이 추가 로 확인되었다. MLST 분석을 위한 sequence type의 종류 가 보강됨에 따라 해당 균주의 분류학적 분석이 보다 정확 하게 이루어질 것으로 사료된다.
감사의 글
본 연구는 농림축산식품부 고부가가치식품기술개발사업( 과제번호: 314073-03-1-HD040)에 의해 이루어진 것으로 연구비 지원에 감사드립니다.
REFERENCES
Dunbar J, Ticknor LO, Kuske CR. Phylogenetic specificity and reproducibility and new method for analysis of terminal restriction fragment profiles of 16S rRNA genes from bacterial communities. Appl. Environ. Microbiol. 2001; 67 p. 190-197.

Gancheva A, Pot B, Vanhonacker K, Hoste B, Kersters K. A polyphasic approach towards the identification of strains belonging to Lactobacillus acidophilus and related species. Syst. Appl. Microbiol. 1999; 22 p. 573-585.

Gevers D, Huys G, Swings H. Applicability of rep-PCR fingerprinting for identification of lactobacillus species. FEMS Microbiol. Lett. 2001; 205 p. 31-36.

Healy M, Huong J, Bittner T, Lising M, Frye S, Raza S, Schrock R, Manry J, Renwick A, Nieto R, Woods C, Versalovic J, Lupski JR. Microbial DNA typing by automated repetitive-sequence-based PCR. J. Clin. Microbiol. 2005; 43 p. 199-207.

Higgins CF, Ames GFL, Barnes WM, Clement JM, Hofnung M. A novel intercistronic regulatory element of prokaryotic operons. Nature. 1982; 298 p. 760-762.

Homan WL, Tribe D, Poznanski S, Li M, Hogg G, Spalburg E, Van Embden JD, Willems RJ. Multilocus sequence typing scheme for Enterococcus faecium. J. Clin. Microbiol. 2002; 40 p. 1963-1971.

Kostman JR, Elliot TD, Lipuma JJ, Stull TL. Molecular epidermiology of Pseudomonas cepacia determined by polymerase chain reaction ribotyping. J. Clin. Microbiol. 1992; 30 p. 2084-2087.

Martino ME, Maifreni M, Marino M, Bartolomeoli I, Carraro L, Fasolato L, Cardazzo B. Genotypic and phenotypic diversity of Pediococcus pentosaceus strains isolated from food matrices and characterisation of the penocin operon. Anton. Leeuw. 2013; 103 p. 1149-1163.

Marsh TL. Terminal restriction fragment length polymorphism (T-RFLP): an emerging method for characterizing diversity among homologous populations of amplification products. Curr. Opin. Microbiol. 1999; 2 p. 323-327.

McCartney A. Application of molecular biological methods for studying probiotics and the gut flora. Br. J. Nutr. 2002; 88 p. S29-37.

Muyzer G, de Waal EC, Uitterlinden AG. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction amplified genes coding for 16S rRNA. Appl. Environ. Microbiol. 1993; 59 p. 695-700.

Olson MV. Separation of large DNA molecules by pulsed field gel electrophoresis. A review of the basic phenomenology. J. Chromatogr. 1989; 470 p. 377-383.

O'Sullivan DJ. In: Tannock GW, editor. Methods for analysis of the intestinal microflora. Probiotics: a critical review. 1999Horizon Scientific PressNorfolk, UK.

PubMLST Databases hosted on PubMLST. Available from. http://pubmlst.org/. Acccessed Apr. 05. 2015.

Stackebrandt E, Frederiksen W, Garrity GM, Grimont PAD, Kampfer P, Maiden MCJ, Nesme X, Rossello-Mora R, Swings J, Truper HG, Vauterin L, Ward AC, Whitman WB. Report of the ad hoc committee for the re-evaluation of the species definition in bacteriology. Int. J. Syst. Evol. Micr. 2002; 52 p. 1043-1047.

Sun Z, Liu W, Song Y, Xu H, Yu J, Bilige M, Zhang H, Chen Y. Population structure of Lactobacillus helveticus isolates from naturally fermented dairy products based on multilocus sequence typing. J. Dairy Sci. 2015; 98 p. 2962-2972.

Ventura M, Zink R. Specific identification and molecular typing analysis of Lactobacillus johnsonii by using PCR-based methods and pulsed-field gel electrophoresis. FEMS Microbiol. Lett. 2002; 217 p. 141-154.

Vos P, Hogers R, Bleeker M, Reijans M, van de Lee T, Hornes M, Frijters A, Pot J, Peleman J, Kuiper M. AFLP: a new technique for DNA fingerprinting. Nucleic Acids Res. 1995; 23 p. 4407-4414.

William JGK, Hanafey MK, Rafolski JA, Tingey SV. Genetics analysis using random amplified polymorphic DNA markers. Methods Enzymol. 1993; 218 p. 704-740.