서 론
세계적으로 육류 소비량은 크게 증가하고 있다. 우리나 라의 경우에도 급격한 경제 발전 이후에 식육, 육가공품, 유가공품 등의 식품 소비가 증가하였다(Bratosin et al., 2021). 세계적으로 인구는 꾸준히 증가하고 있어 2050년에 는 연간 4억톤 이상의 육류 생산이 필요할 것으로 예상된 다(Boland et al., 2013). 또한, 동물복지, 건강, 지속 가능 성에 대한 관심 증가와 더불어 가축 전염병 발병에 따른 육류의 공급 안정성 문제가 대두되면서 육류 단백질을 대 체할 수 있는 대체식품에 대한 관심이 증대되고 있다 (Bajic et al., 2022). 식물성 대체육 시장의 급격한 성장과 더불어 주원료로 사용되는 식물성 단백질 시장 또한 급격 히 성장하고 있다(Boland et al., 2013). 그러나 국내 생산 식물성 단백질은 가격 경쟁력에서 밀려 국내 일반식품 및 대체식품의 원료로 이용되는 식물성 단백질은 전량 수입에 의존하고 있는 실정이다(KISTEP, 2023). 이러한 문제점을 해결하기 위한 방안으로 품질이 우수하고, 대량생산이 용 이한 미생물 유래 단백질을 원료로 사용하는 방법이 세계 적으로 주목받고 있다(Bajic et al., 2022).
미생물 유래 단백질은 “균체 단백질(single cell protein)” 로도 불리며, 세균, 곰팡이, 조류, 효모 등의 미생물 배양을 통해 생산된다. 다른 대체 단백질 소재와 비교하여 균체 단백질 생산은 단시간 대량생산에 유리한 장점이 있기 때 문에 2050년에는 균체 단백질의 세계시장 규모가 2조 5천 억 달러에 이를 것으로 예측된다(Bajic et al., 2022). 다양 한 미생물 중에서 Saccharomyces cerevisiae 효모는 단백질 함량이 높을 뿐만 아니라 베타글루칸, 글루타치온, 무기질, 비타민 B군 등을 함유하고 있어 활용성이 높다. 또한, S. cerevisiae는 오랜 시간에 걸쳐 인체에 무해한 것으로 알려 진 대표적 GRAS (generally recognized as safe) 균주이기 때문에 다양한 대체식품 개발에 용이하다(Zhu et al., 2016;Bratosin et al., 2021). 그러나 일반적으로 세균과 비 교하여 효모의 단백질 함량이 낮아(Bratosin et al., 2021) 효모 유래 균체 단백질 상용화를 위해서는 효모의 단백질 함량을 최대화시키는 연구가 필요하다. 이와 관련된 선행연 구로 단백질 함량이 높은 야생형 효모 균주 선별(Ritala et al., 2017;Natesuntorn et al., 2019)과 더불어 단백질 함량 증대를 위한 발효 조건 최적화(Aggelopoulos et al., 2014;Yadav et al., 2014)가 수행되었다.
미생물을 이용한 고부가가치 소재 생산에는 주로 대사공 학(metabolic engineering)적 접근법이 활용된다. 대사공학 은 대사 물질의 생산경로 조절을 통해 목적하는 물질의 생 산을 최적화하는 기술을 일컫는다. 이러한 기술이 활용되 기 위해서는 목적 물질 생산에 관여되는 속도결정단계 (rate-limiting step)에 대한 정보가 필요하다. 그러나 균체 단백질 생산에 관여된 유전인자들의 높은 복잡성으로 인해 현재까지 균체 단백질 생산 관련 속도결정단계에 대해서는 아직 보고 되지 않았다.
본 연구에서는 식용 가능한 야생형 효모 균주 중에서 단 백질 함량이 높은 균주를 선별하고 무작위 돌연변이 (random mutagenesis)를 통해 단백질 함량이 더욱 증대된 변이체를 구축하고자 하였다. 무작위 돌연변이를 통해 구 축된 변이체의 경우 인위적으로 조작된 유전자변형생물체 (genetically modified organism) 규제 대상이 아니기 때문에 대체 단백질 소재로 활용되기에 용이할 것으로 판단된다.
재료 및 방법
30종의 야생형 S. cerevisiae 및 근연종은 농업유전자원정 보센터와 한국미생물보존센터로부터 분양받았다(Table 1). 실험용 효모 균주로는 S. cerevisiae D452-2 (Hosaka et al., 1992)를 이용하였다.
YPD 고체배지(10 g/L yeast extract, 20 g/L bacto peptone, 15 g/L bacto agar, 20 g/L glucose)에 형성된 효모 콜로니 (colony)를 멸균 이쑤시개로 찍어 200 μL YP50D 액체배지 (10 g/L yeast extract, 20 g/L bacto peptone, 50 g/L glucose) 에 접종하였다. 접종 후에 30 °C, 250 rpm에서 48시간 동안 진탕 배양한 효모 세포는 5,000 rpm에서 10분간 원심분리 하여 회수하였다.
초저온 내동고(deep freezer)에 보관 중인 효모 글리세롤 스탁(stock) 100 μL을 5μL YPD 액체배지(10 g/L yeast extract, 20 g/L bacto peptone, 20 g/L glucose)에 접종하여 30 °C, 250 rpm에서 48시간 동안 진탕 배양하였다. 이렇게 전배양된 배양액을 600 nm에서의 광학밀도(OD600)가 1.0이 되도록 100mL YP50D 액체배지가 포함되어 있는 플라스 크에 접종하였다. 본 배양은 30 °C, 250 rpm에서 48시간 동안 진행하였다. 단백질 농도 측정을 위해 본 배양액 200 μL을 96-well plate에 옮기고 5,000 rpm에서 10분간 원 심분리하여 효모 세포를 회수하였다.
회수된 효모 세포에 100μL Y-PERTM yeast protein extraction reagent (Thermo Fisher Scientific Inc., Rockford, IL, USA)를 첨가하여 세포를 파쇄하였다. 멸균수를 이용하여 10배 희석한 효모 파쇄액 10 μL에 protein assay reagent (Bio-Rad Laboratories, Inc., Hercules, CA, USA) 40 μL, 멸균수 150 μL를 첨가하여 반응시킨 후 595 nm에서 흡광 도를 측정하였다. 효모 파쇄액에 존재하는 단백질 농도는 농도를 이미 알고 있는 알부민 표준 용액을 이용해 계산하 였다. 효모 파쇄액에 존재하는 구성 아미노산(total amino acid) 농도와 유리 아미노산(free amino acid) 농도는 서울 대학교 농생명과학공동기기원(NICEM)에 의뢰하여 분석하 였다. 분석은 선행연구(Lee et al., 2013)에 기반하여 Inno C18 컬럼(Young Jin Biochrom Co., Seongnam, Republic of Korea)이 설치된 고성능 액체 크로마토그래피(Thermo Fisher Ultimate 3000)를 이용하여 수행되었다.
5 mL YPD 액체배지에서 12시간 배양된 S. cerevisiae KCCM 34709 균주를 OD600*mL = 0.2가 되도록 회수하였 다. 예를 들어, OD600 수치가 1.0인 경우에 0.2mL의 효모 배양액을 원심분리하여 효모 세포를 회수하였다. 회수된 효모 세포를 200 μL YPD 액체배지로 resuspension하고 자 외선 램프(G30T8, Sankyo Denki, Hiratsuka, Japan)로부터 20 cm 떨어지도록 위치시켰다. 24시간 동안 자외선을 조사 하면서 시간에 따른 효모의 생존률을 측정하였다. 생존률 은 자외선을 조사한 효모 세포 현탁액 50 μL를 회수하여 serial dilution하고 YPD 고체배지에 도말하여 형성되는 콜 로니 수를 기반으로 계산하였다.
결과 및 고찰
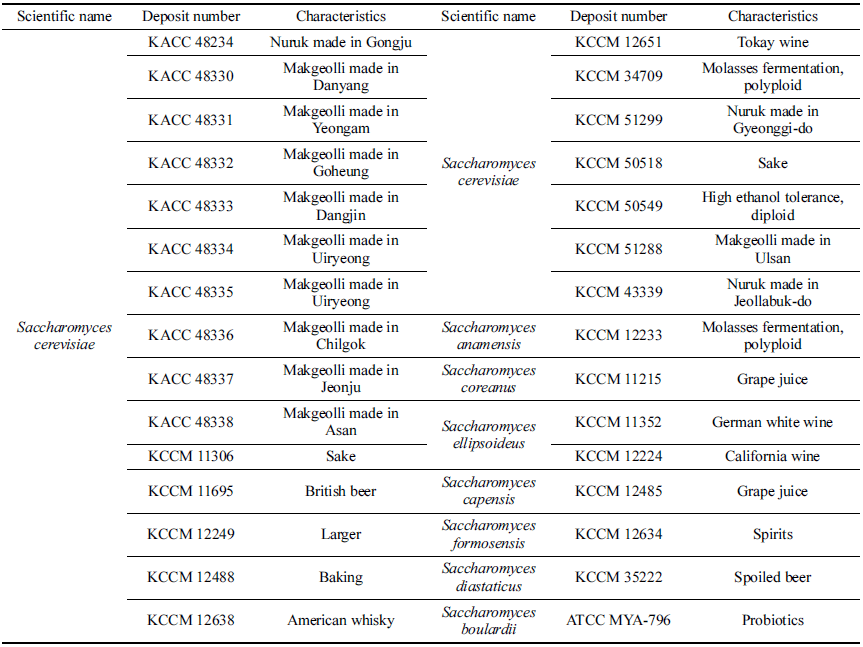
식품산업에 적용 가능한 단백질 고함량 효모 균주를 탐 색하기 위해 양조, 제빵 등 다양한 제품 개발에 이용되고 있는 식용 S. cerevisiae 및 근연종을 농업유전자원정보센터 와 한국미생물보존센터로부터 분양받았다(Table 1). 실험용 효모 균주인 S. cerevisiae D452-2 (Hosaka et al., 1992)를 포함하여 Table 1에 명시된 30종의 식용 효모 균주의 단백 질 농도를 측정하였다. S. cerevisiae D452-2 대비 단백질 농도가 4배 이상 높은 야생형 효모 균주는 S. cerevisiae KCCM 34709와 S. anamensis KCCM 12233으로 나타났 다(Fig. 1). S. cerevisiae KCCM 34709와 S. anamensis KCCM 12233의 단백질 농도는 S. cerevisiae D452-2 대비 각각 4.1배, 5.0배 높은 수치를 나타냈다. 단백질 농도가 높은 두 종의 균주 모두 당밀(molasses) 발효용 효모 균주 로 나타나(Table 1) 당밀 발효에 최적화된 효모 균주의 단 백질 함량이 높은 것으로 보인다. 진화론적으로 판단했을 때, 당밀에 일부 존재하는 질소원을 이용하여 고효율로 단 백질을 생산해 낼 수 있는 효모 균주만이 당밀 발효 시에 우점종으로 생장했을 것으로 생각되나 아직 정확한 분자적 인 메커니즘은 규명되지 않아 관련된 추가 연구가 필요할 것으로 판단된다. S. cerevisiae와 S. anamensis 중에서 국 내 식품원료로 등재되어 있는 효모 균주는 S. cerevisiae이 므로 최종적으로 S. cerevisiae KCCM 34709 균주를 단백 질 고생산용 식용 효모 균주로 선별하였다.
선별된 S. cerevisiae KCCM 34709 균주의 단백질 함량 을 증대시키기 위해 무작위 돌연변이 방법을 이용하기로 하였다. 무작위 돌연변이를 통한 균주 개량 방법은 크게 두 단계 과정을 거친다. 먼저 다양한 화학적 및 물리적인 방법을 통해 수많은 서로 다른 변이체들을 포함하는 돌연 변이 라이브러리 제작한다. 그 이후에는 구축된 돌연변이 라이브러리로부터 실험자가 원하는 형질을 지니는 변이체 를 선택적으로 선별하게 된다. 보통은 원하는 형질을 지니 는 변이체를 단시간에 효과적으로 선별해 낼 수 있는 방법 의 유무에 따라 무작위 돌연변이를 통한 균주 개량방법의 효율성이 결정된다. 그러나 단백질 고함량 효모 변이체만 을 선택적으로 선별하거나 우점종이 될 수 있게 만드는 방 법은 현재까지 보고되지 않았다. 따라서, 본 연구에서는 96-well plate를 이용하여 최소한의 부피에서 다양한 변이체 를 배양하고 효모 세포 파쇄 용액인 Y-PERTM yeast protein extraction reagent (Thermo Fisher Scientific Inc.)를 첨가하 여 세포 파쇄액을 제조한 후에 브래드퍼드 단백질 정량법 (Bradford protein assay)을 이용하여 변이체간 상대적인 단 백질 함량(%) 및 농도(mg/L)를 결정하였다. 브래드퍼드 단 백질 정량법의 반응성은 단백질 농도뿐만 아니라 아미노산 의 종류 등의 다른 인자들에 의해서도 영향을 받기 때문에 (Sapan et al., 1999) 본 연구에서는 변이체간 상대적인 단 백질 함량 및 농도 비교에만 이용하였다.
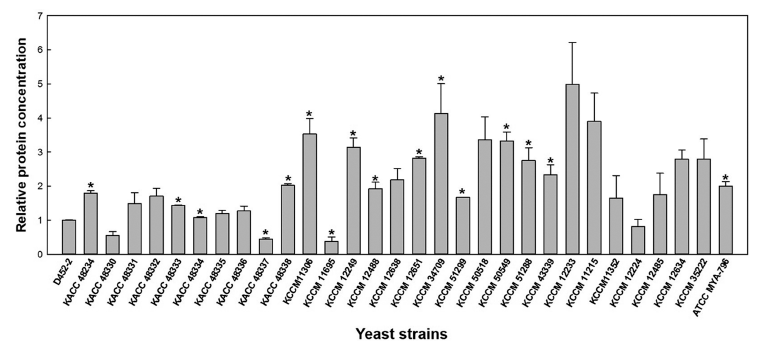
돌연변이 라이브러리는 S. cerevisiae KCCM 34709 균주 에 자외선을 조사하여 구축하였다. 자외선 조사시간이 증 가할수록 효모 유전체에 발생하는 변이빈도는 증가하는 반 면 효모의 생존률은 감소한다. 선행연구에 따르면 효모의 생존률 2-5%를 나타내는 자외선 조사조건이 효모 유전체 의 다양한 위치에 변이를 유발하여 목적하는 형질을 나타 내는 변이체 선별을 위한 돌연변이 라이브러리 구축에 용 이하였다(Taloria et al., 2012). 이에 따라 본 연구에서도 S. cerevisiae KCCM 34709 균주에 다양한 시간별로 자외선 을 조사하여 5.0%의 생존률을 나타낸 조건의 시료를 채취 하였다. 이렇게 구축된 돌연변이 라이브러리를 YPD 고체 배지에 도말하여 생성된 콜로니(colony) 중에서 1,000개를 무작위로 선별하여 200 μL의 YP50D 액체배지를 포함하는 96-well plate에 접종하였다. 효모의 최적 생장 온도인 30 °C 에서 250 rpm의 교반 속도로 48시간 동안 진탕 배양한 후 에 변이체들의 상대 단백질 함량(relative protein content) 및 상대 단백질 농도(relative protein concentration)를 측정 하였다. Fig. 2에서 볼 수 있듯이 대다수의 변이체는 모균 주인 S. cerevisiae KCCM 34709와 비슷한 수준의 단백질 함량 및 농도를 나타내었다. 그러나 일부 변이체들은 S. cerevisiae KCCM 34709대비 단백질 함량 및 농도가 50% 이상 향상되었다. 최종적으로 단백질 함량이 50% 이상 향 상된 변이체 중에서는 11종(#92, #176, #182, #294, #359, #654, #669, #688, #694, #855, #991)을 선별하였고(Fig. 2A), 단백질 농도가 50% 이상 향상된 변이체 중에서는 5 종(#177, #180, #394, #680, #893)을 선별하였다(Fig. 2B).
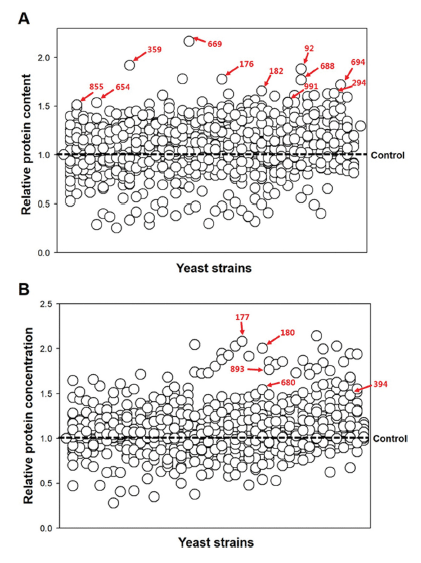
배양액의 부피에 따라 산소전달 및 교반 효율 등의 인자 들이 변화하기 때문에 배양액의 부피를 100 mL로 증가시 킨 플라스크 수준에서 2차 스크리닝을 진행하고자 하였다. 96-well plate 수준의 1차 스크리닝에서 선별된 16종의 변 이체를 100mL의 YP50D 액체배지를 포함하는 플라스크 에 접종하였다. 30 °C, 250 rpm의 조건에서 48시간 동안 진탕 배양한 후 변이체들의 상대 단백질 함량 및 농도를 측정하였다. 그 결과 #180, #394 변이체는 2차 스크리닝 조건에서도 모균주인 S. cerevisiae KCCM 34709보다 유의 미하게 높은 단백질 함량 및 농도를 나타내는 것으로 관찰 되었다(Fig. 3). 그중에서도 특히 #180 변이체는 모균주 S. cerevisiae KCCM 34709대비 각각 28%, 29% 증대된 단백 질 함량 및 농도를 나타냈다.
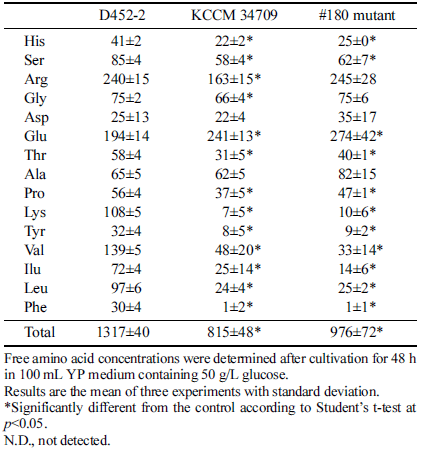
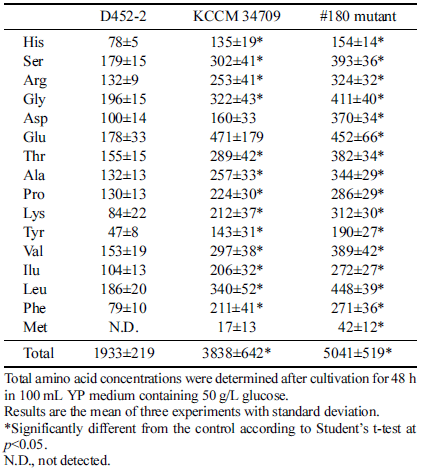
브래드퍼드 단백질 정량법은 모균주 및 변이체들 사이의 상대적인 단백질 농도 측정에만 이용될 수 있기 때문에 효 모 내에 포함되어 있는 정확한 단백질 정량은 고성능 액체 크로마토그래피를 이용해 수행하였다. 그 결과, 유리 아미 노산의 경우 실험용 효모 균주인 S. cerevisiae D452-2 대 비 S. cerevisiae KCCM 34709와 #180 변이체에서 전체적 으로 농도가 미량 감소하였지만 큰 차이는 없었다(Table 2). 반면 구성 아미노산의 경우 S. cerevisiae D452-2와 비 교하여 S. cerevisiae KCCM 34709 및 #180 변이체에서의 농도가 측정된 모든 아미노산에 대해 월등하게 높게 나타 났다(Table 3). #180 변이체는 글루탐산을 제외한 측정된 모든 구성 아미노산에 대해서 모균주 S. cerevisiae KCCM 34709보다 높은 농도를 나타냈다. 이에 따라, 구성 아미노 산 전체 농도는 #180 변이체의 경우 5,041±519 mg/L로 3,838±642 mg/L를 나타낸 S. cerevisiae KCCM 34709보다 31% 높은 수치를 나타냈다. 이러한 결과는 자외선 조사 및 브래드퍼드 단백질 정량법 기반의 무작위 돌연변이 및 선별 기법을 통해 단백질 고함량 S. cerevisiae KCCM 34709 균주의 단백질 생산 농도를 더욱 증대시킬 수 있음 을 보여주었다. 효모의 제한 아미노산은 메티오닌으로 알 려져 있으며(Bratosin et al., 2021), 본 연구에서도 S. cerevisiae KCCM 34709 및 #180 변이체 모두 메티오닌 함량이 다른 아미노산에 비해 낮은 것으로 확인되었다 (Table 3). 따라서, 후속 연구에서는 유도진화(directed evolution), 대사공학과 같은 기법을 통해 #180 변이체의 메티오닌 함량을 더욱 증대시킬 필요가 있을 것으로 생각 된다.
|
|
요 약
본 연구에서는 단백질 고생산 효모 균주를 개발하고자 하였다. 이를 위해 먼저 식용 가능한 야생형 S. cerevisiae 및 근연종 30종의 단백질 농도를 측정하여 비교하였다. 그 결 과, 실험용 효모 균주인 S. cerevisiae D452-2 대비 세포 내 단백질 농도가 4.1배 높은 당밀 발효용 효모 S. cerevisiae KCCM 34709를 단백질 고함량 식용 효모로 선별하였다. S. cerevisiae KCCM 34709 균주의 단백질 함량을 더욱 증 대시키기 위해 자외선 조사 및 브래드퍼드 단백질 정량법 기반의 무작위 돌연변이 및 선별을 수행하였다. 96-well plate 수준의 1차 스크리닝 및 플라스크 수준의 2차 스크 리닝을 통해 최종적으로 #180 변이체를 선별하였다. #180 변이체는 글루탐산을 제외한 모든 구성 아미노산에 대해 모균주인 S. cerevisiae KCCM 34709보다 높은 수치를 나 타냈다. #180 변이체의 구성 아미노산 농도는 5,041±519 mg/L로 나타나 S. cerevisiae KCCM 34709보다 31% 증대 된 수치를 보였다. 본 연구를 통해 개발된 S. cerevisiae KCCM 34709의 #180 변이체는 다양한 대체식품의 단백질 원료로 활용될 수 있을 것으로 기대된다.