Article
발효 균주 탐색을 위한 목장 주변 토양에서 분리한 내염성 미생물의 다양성 및 효소 생산능 분석
이용직, 신기선1, 이상재2,*
A Research on the Diversity and Enzyme Productivity of Halophilic Microorganisms Isolated from the Soil around the Ranch for the Exploration of Fermentation Strains
Yong-Jik Lee, Kee-Sun Shin1, Sang-Jae Lee2,*
Department of Bio-Cosmetics, Seowon University
1Industrial Bio-materials Research Center, Korea Research Institute of Bioscience and Biotechnology (KRIBB)
2Major in Food Biotechnology and Research Center for Extremophiles & Marine Microbiology, Silla University
*Corresponding author: Sang-Jae Lee, Major in Food Biotechnology and Research Center for Extremophiles & Marine Microbiology, Silla University, 140 Baegyang-daero, 700beon-gil, Sasang-Gu, Busan 46958, Korea Tel: +82-51-999-5447: Fax: +82-51-999-5458 E-mail:
sans76@silla.ac.kr
© Korean Society for Food Engineering. All rights reserved.
Received: Jan 30, 2020; Revised: Feb 20, 2020; Accepted: Feb 21, 2020
Abstract
The diversity and characterization of microorganisms isolated from the soil around several ranches in Korea were confirmed in this study. To isolate halophilic microorganisms, the marine agar medium was basically used and cultivated at 37ºC for several days. After single colony isolation, a total of 116 pure colonies were isolated and phylogenetic analysis was carried out based on the result of 16S rDNA sequencing, indicating that isolated strains were divided into 4 phyla, 23 families, 30 genera and 51 species. To confirm whether isolated strain can be a candidate for the fermentation of diverse food ingredients, amylase, lipase, and protease enzyme assays were performed individually, showing that 92 strains possessed at least one enzyme activity. Especially 4 strains, identified to Jonesia quinghaiensis (isolate name: JSF 19-2), Halomonas alkaliantarctica (isolate name: JSF 21), Bacillus velezensis (isolate name: NWFY-36), and Staphylococcus capitis subsp. urealyticus (isolate name: MSY-5), showed all enzyme activity tested. Moreover, 17 strains showed the ability for auxin production. This result indicated that isolated strains have shown the possibility of the application for the food and feed industries. Therefore, this study has contributed to securing domestic biological resources and the improvement of hydrolytic enzyme activity by using isolated strains.
Keywords: ranch; diversity; microorganisms; enzyme; fermented strains
서 론
효소는 고도의 초정밀성, 특이성, 선택성 및 고효율성의 특성을 가진 생산 수단으로 미래 친환경산업에 그 활용이 확대되고 있으며 이미 많은 식품소재의 가공은 효소반응을 통하여 이루어지고 있고, 그 활용범위는 크게 확대되고 있 다(Singh et al., 2016). 미생물 유래의 효소를 이용한 산업 적 응용은 대다수가 식품산업에서 사용하는 효소의 응용에 서 시작되었기 때문에 이 분야에 대한 연구는 많이 되어 왔다. 효소의 식품가공에의 사용은 시럽 제조, 맥주 등 알 코올 발효, 낙농, 제빵, 과일 및 야채주스, 곡물 가공, 식품 보존, 계란 가공, 식품유지 가공, 생선 가공, 향기 및 맛 제조, 동물사료 등 많은 분야에 이용되고 있으며(Singh et al., 2016), 식품유지, 생선가공, 향료 및 맛 제조와 동물사 료를 제조하는데 있어서 protease, amylase, lipase 등을 적 용하고 있다(Mayuri et al., 2019).
산업계에서 이용되고 있는 효소는 단백질 분해효소(약 60%)와 탄수화물 분해효소(약 28%)가 대부분 사용되고 있 으며 특히 단백질 분해효소의 경우 식품산업에서 어육단백 질 분해를 통한 액젓의 속성발효(Park et al., 2005) 및 조 미료의 제조(Kim et al., 2000), 식물단백질의 가수분해에 의한 조미 소재의 제조(Jeong et al., 2005; Chae et al., 1997) 등이 보고되고 있다. 또한 건강증진 식품으로 가장 많이 사용되고 있는 홍삼의 효소 처리에 의한 추출 수율 향상과 항산화 활성 향상 및 우수한 진세노사이드로 전환 을 위하여 amylase와 glucoamylase를 활용한 연구(Kim et al., 2011)와 함께 최근에는 홍삼 추출물의 제조에서 단백 질 분해효소를 활용하는 연구도 보고되고 있다(Kim et al., 2019).
또한, 호염성 미생물이 생산하는 protease는 염장식품뿐 만 아니라 수산부산물을 이용한 분야에서도 널리 사용되어 호염성 protease를 이용하여 수산물의 껍질에서 동물성 콜 라겐의 가수분해산물을 얻을 수 있으며 수산 폐기물을 처 리하는 산업에 있어서도 이러한 protease 또는 protease를 생산하는 미생물을 이용하는 방법이 연구되고 있다(Wu et al., 2008). 그리고 염장식품의 풍미를 증가시키고 숙성 기 간을 줄여주기 위한 starter로서 protease를 생성하는 호염 성 미생물은 염장 산업에서 중요하며, protease에 의한 분 해산물이 풍미를 증진시킬 뿐만 아니라 여러 가지의 생리 활성이 있음이 밝혀짐에 따라 다양한 분야에서 protease의 활용을 위한 연구가 진행되고 있다(Mayuri et al., 2019). 염장식품을 위한 미생물은 고염농도 뿐만 아니라 다양한 온도범위 및 산성 또는 염기성 조건에서도 생장이 가능하 며 강력한 protease 활성을 지닌 것이 보다 이용 가능성이 높다. 식물호르몬 auxin은 다양한 미생물에 의해 식물의 세포신장, 발아, 기관의 분화 등에 관여하는 식물호르몬으 로 작물의 생육을 촉진하는 기능이 있다(Jung et al., 2006; Jung et al., 2007).
따라서 본 연구에서는 목장 주변의 토양으로부터 호염성 미생물을 분리하고 식품 산업용 효소시장에 응용 가능한 효소 3종(amylase, lipase, protease)의 활성을 중심으로 분 리된 미생물의 다양한 분해 효소 생산 특성 탐색과 화학비 료를 대체하는 미생물비료로써의 활용 가능성을 확인하기 위한 auxin 생산능 분석을 실시하였다. 이는 국내 호염성 미생물 자원의 가치를 제고하고 식품 산업용 효소 및 미생 물비료 연구의 기본 생물소재로 활용이 가능할 것으로 예 상된다.
재료 및 방법
호염성 세균 분리 및 배양
국내의 제주 양떼 목장(제주시 애월읍 소재), 녹원 목장( 제주시 한림읍 소재), 아침미소 목장(제주시 월평동 소재) 의 토양으로부터 호염성 미생물 분리를 위해 각각의 샘플 을 멸균된 0.85% 생리식염수에 첨가하여 voltexing으로 현 탁하였다. 현탁한 샘플 1 mL을 사용하여 10−1-10−4 배로 단 계희석 한 후, 일반 증식배지로 해양미생물 전용배지인 marine agar (BD, Difco Laboratories, Detroit, MI, USA) 배지에 3% NaCl을 첨가한 고체배지를 제작하여 희석액을 도말하여 37°C에서 호기적으로 호염미생물을 배양하였다. 배양 후 선택적으로 배지상에 나타나는 균의 크기, 모양, 색깔 등 형태학적 모습을 관찰한 후 동일한 고체배지를 사 용하여 추가적으로 single colony isolation을 수행하였다. 순수분리된 균주의 혼합배지에서의 생육 가능성을 확인하 기 위하여, nutrient agar (BD, Difco Laboratories), R2A agar (BD, Difco Laboratories), 및 tryptic soy agar (BD, Difco Laboratories)에 평판 도말법을 이용하여 37°C에서 7 일간 정치 배양을 하였다. 그리고 분리된 균주들이 호염성 균주로써 5%와 10% NaCl 농도에서도 생육이 가능한지를 확인하기 위하여 marine agar 배지에 5%와 10% NaCl을 첨가한 배지를 제작하여 분리 균주들의 생육을 확인하였으 며 최적의 생육 pH 조건을 확인하기 위하여 pH를 5, 7, 9 로 각각 조절한 marine agar 배지에 분리 균주들의 생육을 확인하였다.
16S rDNA 염기서열의 계통학적 분석
국내외 다양한 염전으로부터 호기적 배양 조건에서 분리 된 균주들의 분자생물학적 동정을 위해 marine agar (BD, Difco Laboratories) 배지에 각각 분리된 균주의 colony가 배양된 상태의 고체 배지를 ㈜바이오팩트에 보내어 16S rDNA 염기서열의 분석을 의뢰하였으며 분석된 16S rDNA 염기서열로부터 가장 유사한 근연 균주의 확인을 위하여 ㈜천랩의 Ezbiocloud (https://www.ezbiocloud.net/)를 사용 하였다. 계통학적 분석은 Clustal W 및 MEGA X 프로그 램을 이용하여 확인하였다.
세포외 분해 효소 생산능 분석
분리된 호염성 미생물의 세포외 분해 효소 amylase, lipase, protease 생산능 확인을 위하여 각각의 효소와 특이 적으로 반응할 기질 성분이 포함된 고체평판 선별배지를 사용하였다. 먼저 amylase 생산능은 0.2% soluble starch (BD, Difco Laboratories)를, lipase 생산능은 1% Tween 80 (Sigma Chemical Co., St. Louis, MO, USA)을, protease 생산능은 2% skim milk (BD, Difco Laboratories)를 기질 로 선택하여 marine agar (BD, Difco Laboratories) 배지에 각각 첨가하여 제조하였으며 분리된 균주를 직접 접종하여 37°C에서 7일 배양한 후 투명환(Clear zone)의 직경으로 조사하였다. 분리된 균주의 효소활성 분해능 평가는 배양 후 나타나는 접종균 주위의 투명환의 크기(+++: > 7 mm, ++: 4-6 mm, + : 1-3 mm)로 나타내었다.
옥신(Auxin) 생산능 분석
분리된 균주의 auxin 생산능은 0.1% L-tryptophan이 첨 가된 Marin broth (BD, USA) 배지에 순수분리된 colony를 tooth-picking 한 후 37°C에서 5일 배양하여 Salkowski 시 약(35% HClO4 50 mL + 0.5 M FeCl3 1 mL) 800 μL을 배 양 상등액 400 μL에 섞어준 후 어두운 곳에서 30 min 반 응시켜 육안으로 확인하여 붉은 색깔 변화 정도에 따라 옥 신 생성능 정도를 표시하였다(짙은 붉은색: +++, 일반 붉 은색: ++, 옅은 붉은색: +, 주황색: w, 무색: -).
결과 및 고찰
호염성 세균 분리
국내의 제주 양떼 목장, 녹원 목장, 아침미소 목장의 토 양으로부터 식품 산업에의활용이 용이할 것으로 생각되는 호기적으로 생육 가능한 호염성 미생물을 분리하고자 marine agar 배지에 시료를 희석, 도말하여 배양한 후, 배 양된 colony의 모양, 색깔 등 형태학적 특징을 육안으로 구분이 가능한 균주들을 대상으로 동일한 고체배지를 사용 하여 2차로 단일 균주 분리를 수행하였다. 그 결과 Table 1에서 나타낸 것처럼 제주 양떼 목장 토양으로부터 38 균 주, 녹원 목장 토양에서 64 균주, 아침미소 목장의 토양으 로부터 14 균주가 각각 분리되었으며 본 실험을 통하여 호염성 미생물 총 116 균주를 순수 분리하였다. 또한 주 요성분이 무기염으로 이루어진 marine agar 배지는 해양 미생물 배양에 유리한 배지이기에 산업적 활용가능성을 확 인하기 위하여 대량 배양 등에 많이 활용되는 혼합 배지 (nutrient agar, R2A agar, tryptic soy agar)에서의 분리 균 주들의 생육 가능성을 확인한 결과 116균주 중 4균주(No. 58, 80, 107, 116)을 제외한 112균주(96.5%)가 최소 1 종 류 이상의 혼합 배지에서 생육이 가능(weak growth 포함) 한 것을 확인하였다. 그리고 분리 균주가 높은 염농에서도 생육이 가능한지를 확인하기 위하여 marine agar 배지에 5%와 10% NaCl을 각각 첨가한 배지를 제작하여 분리 균 주들의 생육을 확인한 결과, 5% NaCl 조건에서는 2균주를 제외한 모든 균주에서 생육이 가능(weak growth 포함)하였 으며 10% NaCl 조건에서는 78균주가 생육이 가능(weak growth 포함)한 것을 확인하였다. 또한 최적의 생육 pH 조 건을 확인하기 위하여 pH를 5, 7, 9로 각각 조절한 marine agar 배지에 분리 균주들의 생육을 확인해본 결과 분리된 균주 중 1균주(No. 95)를 제외한 모든 균주가 pH 7에서 생육이 가능하였으며 이중 25균주는 pH 5에서도 생육이 가능(weak growth 포함)하였으며, pH 9에서 생육 가능 (weak growth 포함)한 균주는 78균주였다(Table 1). 특히 제주 양떼 목장에서 분리한 균주들은 모두 pH 9에서 생육 이 가능한 것을 확인하였는데, 이는 섬이라는 지리적 위치 로 인하여 토양이 내륙에 위치하는 목장의 토양보다 알칼 리화되어 있는 것으로 추측된다.
Table 1.
Isolation and identification of halophilic microorganisms isolated from the soil around the several ranches in Korea
|
No. |
Source |
Isolate number |
NA# |
R2A |
TSA |
MA* |
|
NaCl (5%) |
NaCl (10%) |
pH 5 |
pH 7 |
pH 9 |
|
1 |
|
JSF-1 |
Wb |
-c |
+a |
+ |
+ |
- |
+ |
+ |
|
2 |
|
JSF-3 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
3 |
|
JSF-4 |
+ |
+ |
+ |
+ |
+ |
W |
+ |
+ |
|
4 |
|
JSF-5 |
+ |
+ |
+ |
+ |
+ |
W |
+ |
+ |
|
5 |
|
JSF-6 |
+ |
+ |
+ |
+ |
W |
W |
+ |
+ |
|
6 |
|
JSF-7 |
+ |
+ |
+ |
+ |
W |
- |
+ |
+ |
|
7 |
|
JSF-8-1 |
+ |
+ |
+ |
+ |
- |
- |
+ |
+ |
|
8 |
|
JSF-8-2 |
+ |
+ |
+ |
+ |
+ |
W |
+ |
W |
|
9 |
|
JSF-8-3 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
W |
|
10 |
|
JSF-9 |
+ |
+ |
+ |
+ |
+ |
W |
+ |
+ |
|
11 |
|
JSF-10 |
+ |
+ |
+ |
+ |
w |
- |
+ |
+ |
|
12 |
|
JSF-11-1 |
W |
+ |
+ |
+ |
- |
- |
+ |
W |
|
13 |
|
JSF-11-2 |
+ |
+ |
+ |
+ |
w |
- |
+ |
W |
|
14 |
|
JSF-11-3 |
W |
+ |
+ |
+ |
- |
- |
+ |
W |
|
15 |
|
JSF-12-1 |
+ |
+ |
+ |
+ |
w |
- |
+ |
+ |
|
16 |
|
JSF-13-1 |
- |
W |
+ |
+ |
- |
- |
+ |
+ |
|
17 |
|
JSF-13-2 |
- |
+ |
+ |
+ |
+ |
- |
+ |
W |
|
18 |
|
JSF-13-3 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
W |
|
19 |
Jeju yangtte farm |
JSF-13-4 |
- |
W |
+ |
+ |
- |
- |
+ |
+ |
|
20 |
|
JSF-13-5 |
- |
+ |
+ |
+ |
+ |
- |
+ |
W |
|
21 |
|
JSF-13-6 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
W |
|
22 |
|
JSF-14-1 |
+ |
+ |
+ |
+ |
w |
- |
+ |
+ |
|
23 |
|
JSF-16 |
W |
- |
+ |
+ |
+ |
- |
+ |
+ |
|
24 |
|
JSF-17-1 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
25 |
|
JSF-17-2 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
26 |
|
JSF-18 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
27 |
|
JSF-19-1 |
+ |
+ |
+ |
+ |
- |
- |
+ |
W |
|
28 |
|
JSF-19-2 |
+ |
+ |
+ |
+ |
- |
- |
+ |
W |
|
29 |
|
JSF-20 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
30 |
|
JSF-21 |
- |
- |
W |
+ |
+ |
- |
+ |
W |
|
31 |
|
JSF-23 |
- |
W |
W |
+ |
+ |
- |
+ |
W |
|
32 |
|
JSF-24-1 |
W |
+ |
+ |
+ |
- |
- |
+ |
+ |
|
33 |
|
JSF-24-2 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
34 |
|
JSF-24-3 |
W |
+ |
+ |
+ |
- |
- |
+ |
+ |
|
35 |
|
JSF-27-1 |
W |
W |
+ |
+ |
+ |
- |
+ |
+ |
|
36 |
|
JSF-27-2 |
W |
+ |
+ |
+ |
w |
- |
+ |
+ |
|
37 |
|
JSF-28 |
+ |
+ |
+ |
+ |
W |
W |
+ |
+ |
|
38 |
|
JSF-29 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
39 |
Nokwon farm |
NWFY-8 |
- |
+ |
+ |
- |
- |
- |
W |
- |
|
40 |
NWFY-9 |
+ |
+ |
+ |
+ |
+ |
- |
- |
+ |
|
41 |
|
NWFY-11-1 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
42 |
|
NWFY-11-2 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
43 |
|
NWFY-11-3 |
+ |
- |
+ |
+ |
w |
W |
+ |
- |
|
44 |
|
NWFY-11-4 |
+ |
- |
+ |
+ |
- |
- |
+ |
- |
|
45 |
|
NWFY-12 |
- |
+ |
- |
+ |
- |
- |
+ |
- |
|
46 |
|
NWFY-14 |
W |
W |
+ |
+ |
- |
- |
W |
- |
|
47 |
|
NWFY-16 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
48 |
|
NWFY-17 |
W |
+ |
+ |
+ |
- |
- |
+ |
+ |
|
49 |
|
NWFY-18-1 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
50 |
|
NWFY-18-2 |
- |
+ |
+ |
+ |
w |
- |
+ |
- |
|
51 |
|
NWFY-19 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
52 |
|
NWFY-20-1 |
- |
+ |
- |
+ |
- |
- |
+ |
- |
|
53 |
|
NWFY-20-2 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
W |
|
54 |
|
NWFY-20-3 |
- |
+ |
- |
+ |
w |
- |
+ |
- |
|
55 |
|
NWFY-20-4 |
W |
+ |
- |
w |
- |
- |
+ |
- |
|
56 |
|
NWFY-20-5 |
W |
+ |
- |
+ |
- |
- |
+ |
- |
|
57 |
|
NWFY-21 |
- |
+ |
- |
+ |
- |
- |
+ |
- |
|
58 |
|
NWFY-22 |
- |
- |
- |
+ |
+ |
- |
+ |
+ |
|
59 |
|
NWFY-23-1 |
- |
+ |
- |
+ |
- |
- |
+ |
- |
|
60 |
|
NWFY-23-2 |
- |
W |
- |
+ |
- |
- |
+ |
- |
|
61 |
|
NWFY-23-3 |
- |
W |
- |
+ |
w |
- |
+ |
- |
|
62 |
|
NWFY-25 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
W |
|
63 |
|
NWFY-26 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
64 |
|
NWFY-27 |
+ |
+ |
+ |
+ |
w |
W |
+ |
+ |
|
65 |
Nokwon farm |
NWFY-29 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
66 |
|
NWFY-30-1 |
+ |
+ |
+ |
+ |
+ |
W |
+ |
+ |
|
67 |
|
NWFY-30-2 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
68 |
|
NWFY-31 |
W |
W |
- |
+ |
+ |
- |
+ |
+ |
|
69 |
|
NWFY-32 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
70 |
|
NWFY-33-1 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
71 |
|
NWFY-33-2 |
+ |
+ |
+ |
+ |
- |
- |
+ |
+ |
|
72 |
|
NWFY-33-3 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
73 |
|
NWFY-35 |
+ |
+ |
+ |
+ |
w |
+ |
+ |
- |
|
74 |
|
NWFY-36 |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
75 |
|
NWFY-37 |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
76 |
|
NWFY-38 |
+ |
+ |
+ |
+ |
- |
- |
+ |
+ |
|
77 |
|
NWFY-39 |
+ |
+ |
+ |
+ |
w |
+ |
+ |
- |
|
78 |
|
NWFY-40 |
+ |
+ |
+ |
+ |
- |
+ |
+ |
- |
|
79 |
|
NWFY-41 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
80 |
|
NWFY-42 |
- |
- |
- |
+ |
+ |
- |
+ |
- |
|
81 |
|
NWFY-43-2 |
W |
W |
+ |
+ |
- |
- |
+ |
- |
|
82 |
|
NWFY-43-3 |
+ |
+ |
+ |
+ |
- |
- |
+ |
+ |
|
83 |
|
NWFY-43-4 |
+ |
+ |
+ |
+ |
- |
- |
+ |
- |
|
84 |
|
NWFY-43-5 |
+ |
+ |
+ |
+ |
- |
- |
+ |
W |
|
85 |
|
NWFY-44 |
+ |
+ |
+ |
+ |
- |
+ |
+ |
- |
|
86 |
|
NWFY-45 |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
|
87 |
|
NWFY-46 |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
|
88 |
|
NWFO-1 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
89 |
|
NWFO-2 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
W |
|
90 |
|
NWFO-5-2 |
- |
+ |
W |
+ |
- |
- |
+ |
W |
|
91 |
|
NWFO-6 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
W |
|
92 |
|
NWFO-7 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
93 |
|
NWFO-8 |
+ |
+ |
+ |
+ |
- |
- |
+ |
+ |
|
94 |
|
NWFO-9 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
95 |
|
NWFO-13 |
- |
+ |
- |
+ |
- |
- |
- |
- |
|
96 |
Nokwon farm |
NWFO-14-1 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
- |
|
97 |
|
NWFO-14-2 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
- |
|
98 |
|
NWFO-15 |
+ |
+ |
+ |
+ |
+ |
W |
+ |
+ |
|
99 |
|
NWFO-16 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
100 |
|
NWFO-17 |
+ |
+ |
W |
+ |
+ |
W |
+ |
+ |
|
101 |
|
NWFO-18 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
102 |
|
NWFO-19 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
+ |
|
103 |
|
MSY-5 |
+ |
+ |
+ |
+ |
+ |
- |
+ |
- |
|
104 |
|
MSY-7 |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
105 |
|
MSY-9 |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
106 |
|
MSY-10 |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
107 |
|
MSY-14-1 |
- |
- |
- |
+ |
+ |
W |
+ |
+ |
|
108 |
|
TY-SF 3A |
+ |
- |
+ |
+ |
+ |
- |
+ |
- |
|
109 |
Morning-smile farm |
TY-SF 4A |
+ |
+ |
+ |
+ |
- |
- |
+ |
- |
|
110 |
|
TY-SF 2B |
+ |
+ |
+ |
+ |
- |
W |
+ |
- |
|
111 |
|
TY-SF 4B |
+ |
W |
+ |
+ |
- |
- |
+ |
- |
|
112 |
|
TY-SF 1C |
- |
+ |
- |
- |
- |
- |
+ |
- |
|
113 |
|
TY-SF 2C |
+ |
+ |
+ |
+ |
- |
- |
+ |
- |
|
114 |
|
TY-SF 3C |
+ |
+ |
+ |
+ |
+ |
- |
+ |
- |
|
115 |
|
TY-SF 1D |
- |
- |
+ |
+ |
+ |
- |
+ |
- |
|
116 |
|
TY-SF 2D |
- |
- |
- |
+ |
+ |
W |
+ |
- |
Download Excel Table
16S rDNA 염기서열의 계통학적 분석
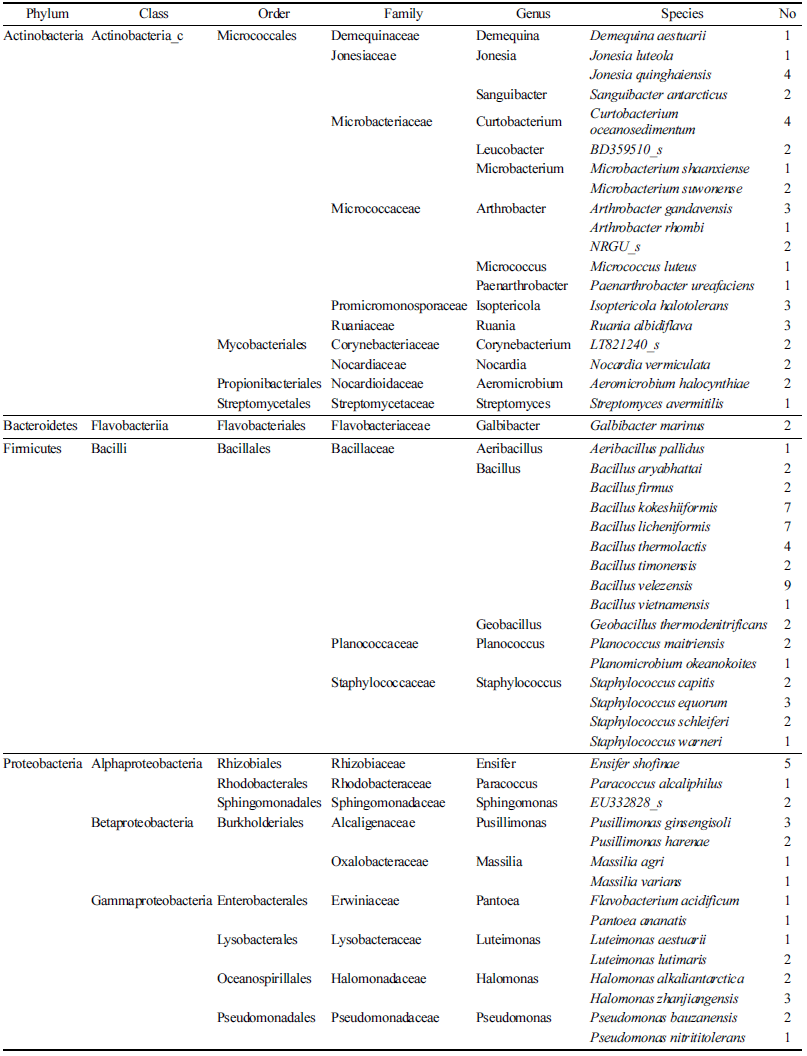
국내의 제주 양떼 목장, 녹원 목장, 아침미소 목장의 토 양으로부터 호기적으로 분리된 116균주의 16S rDNA 염기 서열을 바탕으로 Ezbiocloud 분석서비스를 사용하여 미생물 동정을 실시한 결과 크게 4문 6강 14목 23과 30속 51종으 로 나타났으며(Table 2), 분리 균주와 근연 균주 및 상동성 을 Table 3에 나타내었다. 또한 분리 동정된 균주들 간의 유연관계를 확인하기 위하여 계통수를 작성하였다(Fig. 1). Table 2에서 보는 바와 같이 Firmicutes (Bacilli)가 41.4% 로 가장 우점도가 높았고, Actinobaceria (Actinobacteria_c) 가 32.8%, Proteobacteria (Gammaproteobacteria 11.2%, Alphaproteobacteria 6.9%, Betaproteobacteria 6.0%)가 24.1%, Bacteroidetes는 1.7%로 나타났다. 가장 우점도가 높은 Firmicutes 문은 Bacillaceae 77.1%, Staphylococcaceae 16.7%, Planococcaceae 6.2%로 구성되었으며, 총 3과로 5 속 16종이 분리되었다. 총 38균주가 분리된 Actinobacteria 문에서는 Microbacteriaceae 23.7%, Micrococcaceae 21.1%, Jonesiaceae 18.4%, Promicromonosporaceae와 Ruaniaceae 각 각각 7.9%, Corynebacteriaceae, Nocardiaceae, Nocardioidaceae가 각각 5.3%, Demequinaceae와 Streptomycetaceae 가 각각 2.6%로 구성되었으며, 총 10과로 15속 19종이 분 리되었다.
Fig. 1.
Evolutionary relationships of taxa by using the 16S rDNA sequences of bacteria from the soil around the several ranches in Korea. The evolutionary history was inferred using the Neighbor-Joining method (Saitou and Nei, 1987).
The optimal tree with the sum of branch length = 2.72807696 is shown. The percentage of replicate trees in which the associated taxa clustered together in the bootstrap test (1000 replicates) are shown next to the branches (Felsenstein, 1985). The evolutionary distances were computed using the Maximum Composite Likelihood method (Tamura et al., 2004) and are in the units of the number of base substitutions per site. This analysis involved 116 nucleotide sequences. Codon positions included were 1st+2nd+3rd+Noncoding. All ambiguous positions were removed for each sequence pair (pairwise deletion option). There were a total of 1599 positions in the final dataset. Evolutionary analyses were conducted in MEGA X (Kumar et al., 2018).
Download Original Figure
Table 2.
Phylum analysis of halophilic microorganisms isolated from the soil around the several ranches in Korea
Download Excel Table
Table 3.
Representative sequences of halophilic microorganisms isolated from the soil around the several ranches in Korea
|
No |
Isolate Name |
Closed strain |
Closed strain number |
Similarity (%) |
Extracellular enzyme activity |
Auxin |
Deposited number |
|
Amylase |
Lipase |
Protease |
|
1 |
JSF-1 |
Halomonas zhanjiangensis
|
JSM 078169 |
99.35 |
+++ |
-a |
- |
+++ |
NMC6-B390 |
|
2 |
JSF-3 |
Isoptericola halotolerans
|
YIM 70177 |
98.45 |
+++ |
- |
++ |
++ |
NMC6-B391 |
|
3 |
JSF-4 |
Staphylococcus equorum subsp. Equorum
|
ATCC 43958 |
98.89 |
- |
- |
+++ |
- |
NMC6-B392 |
|
4 |
JSF-5 |
Staphylococcus equorum subsp. Equorum
|
ATCC 43958 |
99.09 |
- |
- |
++ |
- |
NMC6-B393 |
|
5 |
JSF-6 |
Arthrobacter gandavensis
|
R 5812 |
98.33 |
- |
+ |
- |
- |
NMC6-B394 |
|
6 |
JSF-7-1 |
Pseudomonas nitrititolerans
|
GL14 |
98.18 |
- |
- |
- |
- |
NMC6-B395 |
|
7 |
JSF-8-1 |
Microbacterium shaanxiense
|
CCNWSP60 |
99.81 |
+++ |
- |
+ |
- |
NMC6-B396 |
|
8 |
JSF-8-2 |
Bacillus aryabhattai
|
B8W22 |
97.96 |
+++ |
- |
- |
+ |
NMC6-B397 |
|
9 |
JSF-8-3 |
Bacillus aryabhattai
|
B8W22 |
98.63 |
+++ |
- |
+ |
- |
NMC6-B398 |
|
10 |
JSF-9 |
Staphylococcus equorum subsp. equorum
|
ATCC 43958 |
98.52 |
+++ |
- |
+ |
- |
NMC6-B399 |
|
11 |
JSF-10 |
Paracoccus alcaliphilus
|
DSM 8512 |
99.72 |
- |
- |
+++ |
- |
NMC6-B400 |
|
12 |
JSF-11-1 |
Sanguibacter antarcticus
|
DSM 18966 |
99.27 |
+++ |
- |
+++ |
- |
NMC6-B401 |
|
13 |
JSF-11-2-1 |
Planomicrobium okeanokoites
|
IFO 12536 |
98.64 |
- |
- |
+++ |
- |
NMC6-B402 |
|
14 |
JSF-11-3 |
Sanguibacter antarcticus
|
DSM 18966 |
99.27 |
+++ |
- |
+++ |
- |
NMC6-B403 |
|
15 |
JSF-12-1 |
Arthrobacter gandavensis
|
R 5812 |
98.8 |
- |
- |
- |
- |
NMC6-B404 |
|
16 |
JSF-13-1-1 |
LT821240_s
|
Marseille-P2417 |
98.07 |
- |
- |
+ |
- |
NMC6-B405 |
|
17 |
JSF-13-2-1 |
Planococcus maitriensis
|
S1 |
97.89 |
- |
- |
+++ |
- |
NMC6-B406 |
|
18 |
JSF-13-3 |
Staphylococcus schleiferi subsp. coagulans
|
GTC 510 |
97.66 |
- |
- |
++ |
- |
NMC6-B407 |
|
19 |
JSF-13-4 |
LT821240_s
|
Marseille-P2417 |
98.07 |
- |
- |
+ |
- |
NMC6-B412 |
|
20 |
JSF-13-5 |
Planococcus maitriensis
|
S1 |
97.89 |
- |
- |
+++ |
- |
NMC6-B417 |
|
21 |
JSF-13-6 |
Staphylococcus schleiferi subsp. coagulans
|
GTC 510 |
97.66 |
- |
- |
++ |
- |
NMC6-B418 |
|
22 |
JSF-14-1 |
Demequina aestuarii
|
NBRC 106260 |
100 |
- |
- |
- |
- |
NMC5-B320 |
|
23 |
JSF-16 |
Halomonas zhanjiangensis
|
JSM 078169 |
98.9 |
- |
- |
+ |
- |
NMC5-B321 |
|
24 |
JSF-17-1 |
BD359510_s
|
|
98.34 |
- |
- |
++ |
- |
NMC5-B322 |
|
25 |
JSF-17-2 |
BD359510_s
|
|
98.34 |
- |
- |
++ |
- |
NMC5-B323 |
|
26 |
JSF-18 |
Pseudomonas bauzanensis
|
CGMCC 1.9095 |
99.28 |
- |
- |
- |
- |
NMC5-B324 |
|
27 |
JSF-19-1 |
Jonesia luteola
|
YIM 93067 |
97.8 |
+++ |
- |
+ |
- |
NMC5-B325 |
|
28 |
JSF-19-2 |
Jonesia quinghaiensis
|
DSM 15701 |
99.37 |
+++ |
+ |
+++ |
- |
NMC5-B326 |
|
29 |
JSF-20 |
Aeromicrobium halocynthiae
|
KME 001 |
99.13 |
- |
- |
+++ |
- |
NMC5-B327 |
|
30 |
JSF-21 |
Halomonas alkaliantarctica
|
CRSS |
98.63 |
+++ |
+ |
+ |
+++ |
NMC5-B328 |
|
31 |
JSF-23 |
Halomonas alkaliantarctica
|
CRSS |
97.91 |
+++ |
- |
- |
+++ |
NMC5-B329 |
|
32 |
JSF-24-1 |
Jonesia quinghaiensis
|
DSM 15701 |
98.45 |
++ |
- |
+ |
- |
NMC5-B330 |
|
33 |
JSF-24-2 |
Pseudomonas bauzanensis
|
CGMCC 1.9095 |
99.28 |
- |
- |
- |
- |
NMC5-B331 |
|
34 |
JSF-24-3 |
Jonesia quinghaiensis
|
DSM 15701 |
99.19 |
+++ |
- |
- |
- |
NMC5-B332 |
|
35 |
JSF-27-1 |
Halomonas zhanjiangensis
|
JSM 078169 |
99.27 |
- |
- |
- |
- |
NMC5-B333 |
|
36 |
JSF-27-2 |
Jonesia quinghaiensis
|
DSM 15701 |
97.7 |
+++ |
- |
+ |
- |
NMC5-B334 |
|
37 |
JSF-28 |
Arthrobacter gandavensis
|
R 5812 |
98.33 |
- |
+ |
- |
- |
NMC5-B335 |
|
38 |
JSF-29 |
Aeromicrobium halocynthiae
|
KME 001 |
99.13 |
- |
- |
+++ |
- |
NMC5-B336 |
|
39 |
NWFY-8 |
Bacillus velezensis
|
CR-502 |
99.33 |
+++ |
- |
++ |
- |
NMC5-B337 |
|
40 |
NWFY-9 |
Bacillus licheniformis
|
ATCC 14580 |
99.18 |
+ |
- |
+ |
- |
NMC5-B338 |
|
41 |
NWFY-11-1 |
Bacillus licheniformis
|
ATCC 14580 |
98.91 |
- |
- |
+++ |
- |
NMC5-B339 |
|
42 |
NWFY-11-2 |
Bacillus licheniformis
|
ATCC 14580 |
98.54 |
+++ |
- |
+++ |
- |
NMC5-B340 |
|
43 |
NWFY-11-3 |
Bacillus thermolactis
|
R-6488 |
99 |
- |
- |
- |
- |
NMC5-B341 |
|
44 |
NWFY-11-4-1 Bacillus kokeshiiformis |
MO-04 |
98.14 |
- |
- |
- |
- |
NMC5-B342 |
|
45 |
NWFY-12 |
Bacillus thermolactis
|
R-6488 |
98 |
+ |
- |
+ |
- |
NMC5-B343 |
|
46 |
NWFY-14 |
Bacillus thermolactis
|
R-6488 |
99.54 |
- |
- |
++ |
- |
NMC5-B344 |
|
47 |
NWFY-16 |
Ruania albidiflava
|
DSM 18029 |
98 |
- |
- |
++ |
- |
NMC5-B345 |
|
48 |
NWFY-17 |
Microbacterium suwonense
|
M1T8B9 |
99.25 |
+++ |
- |
- |
- |
NMC5-B346 |
|
49 |
NWFY-18-1 |
Staphylococcus warneri
|
ATCC 27836 |
99.91 |
- |
- |
- |
- |
NMC5-B347 |
|
50 |
NWFY-18-2 |
Ensifer shofinae
|
CCBAU 251167 |
96.99 |
- |
- |
- |
+++ |
NMC5-B348 |
|
51 |
NWFY-19 |
Bacillus velezensis
|
CR-502 |
98.07 |
+++ |
- |
+++ |
- |
NMC5-B349 |
|
52 |
NWFY-20-1-1 EU332828_s |
LnR5-44 |
97.2 |
- |
- |
- |
- |
NMC5-B350 |
|
53 |
NWFY-20-2 |
Luteimonas lutimaris
|
G3 |
96.95 |
- |
- |
++ |
- |
NMC5-B351 |
|
54 |
NWFY-20-3-1 Ensifer shofinae |
CCBAU 251167 |
96.7 |
- |
- |
- |
+++ |
NMC5-B352 |
|
55 |
NWFY-20-4 |
Luteimonas aestuarii
|
B9 |
98.5 |
- |
- |
++ |
+++ |
NMC5-B353 |
|
56 |
NWFY-20-5 |
Luteimonas lutimaris
|
G3 |
98.73 |
- |
- |
++ |
- |
NMC5-B354 |
|
57 |
NWFY-21 |
EU332828_s
|
LnR5-44 |
97.4 |
- |
- |
- |
- |
NMC5-B355 |
|
58 |
NWFY-22-1 |
Galbibacter marinus
|
ck-I2-15 |
95.47 |
++ |
- |
- |
- |
NMC5-B356 |
|
59 |
NWFY-23-1 |
Ensifer shofinae
|
CCBAU 251167 |
97.39 |
- |
- |
- |
+++ |
NMC5-B357 |
|
60 |
NWFY-23-2 |
Ensifer shofinae
|
CCBAU 251167 |
97.29 |
- |
- |
+ |
+++ |
NMC5-B358 |
|
61 |
NWFY-23-3 |
Ensifer shofinae
|
CCBAU 251167 |
97.29 |
- |
- |
- |
+++ |
NMC5-B359 |
|
62 |
NWFY-25 |
Arthrobacter rhombi
|
F.98.3HR.69 |
98.19 |
- |
- |
+ |
- |
NMC5-B360 |
|
63 |
NWFY-26 |
NRGU_s
|
BW78 |
98.53 |
- |
- |
++ |
- |
NMC5-B361 |
|
64 |
NWFY-27 |
Ruania albidiflava
|
DSM 18029 |
97.59 |
- |
- |
+ |
- |
NMC5-B362 |
|
65 |
NWFY-29 |
Bacillus velezensis
|
CR-502 |
98.56 |
+++ |
- |
+ |
- |
NMC5-B363 |
|
66 |
NWFY-30-1 |
Bacillus velezensis
|
CR-502 |
99.42 |
+++ |
- |
+ |
- |
NMC5-B364 |
|
67 |
NWFY-30-2 |
Bacillus velezensis
|
CR-502 |
98.84 |
+++ |
- |
+ |
- |
NMC5-B365 |
|
68 |
NWFY-31 |
NRGU_s
|
BW78 |
98.15 |
- |
- |
+ |
- |
NMC5-B366 |
|
69 |
NWFY-32-1 |
Ruania albidiflava
|
DSM 18029 |
97.45 |
- |
- |
++ |
- |
NMC5-B367 |
|
70 |
NWFY-33-1 |
Curtobacterium oceanosedimentum ATCC 31317
|
99.26 |
- |
- |
+++ |
+++ |
NMC5-B368 |
|
71 |
NWFY-33-2 |
Pusillimonas ginsengisoli
|
DCY25 |
98.54 |
- |
- |
- |
++ |
NMC5-B369 |
|
72 |
NWFY-33-3 |
Curtobacterium oceanosedimentum ATCC 31317
|
99.26 |
- |
- |
+++ |
+++ |
NMC5-B370 |
|
73 |
NWFY-35 |
Nocardia vermiculata
|
NBRC 100427 |
99.08 |
- |
- |
- |
- |
NMC5-B371 |
|
74 |
NWFY-36 |
Bacillus velezensis
|
CR-502 |
99.71 |
+++ |
+ |
+++ |
- |
NMC5-B372 |
|
75 |
NWFY-37 |
Bacillus licheniformis
|
ATCC 14580 |
99.08 |
++ |
- |
+++ |
- |
NMC5-B373 |
|
76 |
NWFY-38 |
Pusillimonas harenae
|
B201 |
97.65 |
- |
- |
- |
+++ |
NMC5-B374 |
|
77 |
NWFY-39 |
Nocardia vermiculata
|
NBRC 100427 |
99.08 |
- |
- |
- |
- |
NMC5-B375 |
|
78 |
NWFY-40 |
Curtobacterium oceanosedimentum ATCC 31317
|
99.72 |
- |
- |
+ |
- |
NMC5-B376 |
|
79 |
NWFY-41 |
Bacillus velezensis
|
CR-502 |
99.62 |
+++ |
- |
+++ |
- |
NMC5-B377 |
|
80 |
NWFY-42 |
Galbibacter marinus
|
ck-I2-15 |
98.24 |
- |
- |
- |
- |
NMC5-B378 |
|
81 |
NWFY-43-2 |
Microbacterium suwonense
|
M1T8B9 |
99.15 |
- |
- |
+ |
- |
NMC5-B379 |
|
82 |
NWFY-43-3 |
Pusillimonas ginsengisoli
|
DCY25 |
98.82 |
- |
- |
- |
- |
NMC5-B380 |
|
83 |
NWFY-43-4 |
Pusillimonas ginsengisoli
|
DCY25 |
98.81 |
- |
- |
- |
- |
NMC5-B381 |
|
84 |
NWFY-43-5 |
Pusillimonas harenae
|
B201 |
97.17 |
- |
- |
- |
- |
NMC5-B382 |
|
85 |
NWFY-44 |
Curtobacterium oceanosedimentum ATCC 31317
|
99.72 |
- |
- |
+ |
- |
NMC5-B383 |
|
86 |
NWFY-45 |
Pantoea ananatis
|
LMG 2665 |
99.09 |
+++ |
- |
+++ |
+++ |
NMC5-B384 |
|
87 |
NWFY-46 |
Flavobacterium acidificum
|
LMG 8364 |
98.62 |
- |
++ |
+++ |
+++ |
NMC5-B385 |
|
88 |
NWFO-1 |
Bacillus velezensis
|
CR-502 |
98.09 |
- |
- |
+++ |
- |
NMC5-B386 |
|
89 |
NWFO-2 |
Bacillus firmus
|
NBRC 15306 |
99.09 |
- |
- |
+++ |
- |
NMC5-B387 |
|
90 |
NWFO-5-2 |
Streptomyces avermitilis
|
MA-4680 |
99.54 |
- |
- |
- |
- |
NMC5-B388 |
|
91 |
NWFO-6 |
Bacillus firmus
|
NBRC 15306 |
98.81 |
- |
- |
+++ |
- |
NMC5-B389 |
|
92 |
NWFO-7 |
Isoptericola variabilis
|
MX5 |
99.35 |
+++ |
- |
+ |
- |
NMC5-B390 |
|
93 |
NWFO-8-1 |
Paenarthrobacter ureafaciens
|
DSM 20126 |
98.44 |
- |
- |
+ |
- |
NMC5-B391 |
|
94 |
NWFO-9 |
Isoptericola variabilis
|
MX5 |
99.35 |
+++ |
- |
+ |
- |
NMC5-B392 |
|
95 |
NWFO-13 |
Bacillus thermolactis
|
R-6488 |
98.26 |
- |
- |
++ |
- |
NMC5-B393 |
|
96 |
NWFO-14-1 |
Bacillus velezensis
|
CR-502 |
98.18 |
+++ |
- |
+++ |
- |
NMC4-B482 |
|
97 |
NWFO-14-2 |
Bacillus licheniformis
|
ATCC 14580 |
98.37 |
+++ |
- |
+++ |
- |
NMC4-B483 |
|
98 |
NWFO-15 |
Bacillus timonensis
|
10403023 |
98.09 |
+++ |
- |
- |
- |
NMC4-B484 |
|
99 |
NWFO-16 |
Bacillus licheniformis
|
ATCC 14580 |
98.63 |
+++ |
- |
- |
- |
NMC4-B485 |
|
100 |
NWFO-17 |
Bacillus vietnamensis
|
15-1 |
98.92 |
+++ |
- |
- |
- |
NMC4-B486 |
|
101 |
NWFO-18 |
Bacillus licheniformis
|
ATCC 14580 |
99.18 |
+++ |
- |
- |
- |
NMC6-B408 |
|
102 |
NWFO-19 |
Bacillus licheniformis
|
ATCC 14580 |
98.44 |
+++ |
- |
++ |
- |
NMC6-B427 |
|
103 |
MSY-5 |
Staphylococcus capitis subsp. Urealyticus
|
GTC 727 |
99.26 |
+++ |
+ |
+ |
- |
NMC6-B428 |
|
104 |
MSY-7 |
Staphylococcus capitis subsp. Urealyticus
|
GTC 727 |
99.26 |
- |
+ |
+ |
- |
NMC6-B429 |
|
105 |
MSY-9 |
Massilia varians
|
CCUG 35299 |
98.39 |
+++ |
- |
- |
- |
NMC6-B430 |
|
106 |
MSY-10 |
Massilia agri
|
K-3-1 |
98.07 |
+++ |
- |
- |
- |
NMC6-B431 |
|
107 |
MSY-14-1 |
Micrococcus luteus
|
NCTC 2665 |
98.99 |
+++ |
- |
- |
- |
NMC6-B432 |
|
108 |
TY-SF 3A |
Bacillus kokeshiiformis
|
MO-04 |
100 |
- |
+++ |
+ |
- |
NMC6-B433 |
|
109 |
TY-SF 4A |
Geobacillus thermodenitrificans subsp. Calidus
|
F84b |
99.86 |
++ |
+ |
- |
- |
NMC6-B434 |
|
110 |
TY-SF 2B |
Geobacillus thermodenitrificans subsp. Calidus
|
F84b |
99.86 |
++ |
+ |
- |
- |
NMC6-B435 |
|
111 |
TY-SF 4B |
Bacillus kokeshiiformis
|
MO-04 |
100 |
- |
++ |
++ |
- |
NMC6-B440 |
|
112 |
TY-SF 1C |
Bacillus kokeshiiformis
|
MO-04 |
100 |
- |
++ |
++ |
- |
NMC6-B450 |
|
113 |
TY-SF 2C |
Aeribacillus pallidus
|
KCTC3564 |
99.8 |
+++ |
- |
- |
- |
NMC5-B316 |
|
114 |
TY-SF 3C |
Bacillus kokeshiiformis
|
MO-04 |
100 |
+++ |
- |
- |
- |
NMC5-B317 |
|
115 |
TY-SF 1D |
Bacillus kokeshiiformis
|
MO-04 |
100 |
- |
- |
- |
- |
NMC5-B318 |
|
116 |
TY-SF 2D |
Bacillus kokeshiiformis
|
MO-04 |
100 |
- |
- |
- |
- |
NMC5-B319 |
Download Excel Table
총 28균주가 분리된 Proteobacteria 문에서는 Gammaproteobacteria 강에서 Halomonadaceae 17.9%, Lysobacteraceae 와 Pseudomonadaceae가 각각 10.7%, 그리고 Erwiniaceae 가 7.1%로 구성되었으며, Alphaproteobacteria 강에서는 Rhizobiaceae 17.9%, Sphingomonadaceae 7.1%, 그리고 Rhodobacteraceae가 3.6%로 구성되었으며, Betaproteobacteria 강에서는 Alcaligenaceae 17.9%와 Oxalobacteraceae가 7.1% 로 구성되어 있는 것을 확인하였으며 총 9과로 9속 15종 이 분리되었다. Bacteroidetes 문에서 Galbibacter 속 1종이 분리되었다. 또한 NWFY-22 (isolate name)로 나타낸 분리 균주는 근연 균주와의 16S rDNA 염기서열 상동성이 95.47%로 Galbibacter marinus로 동정되었으며 이는 신종 균주 가능성을 나타내는 것으로 녹원 목장 토양에서 분리 (과제기탁번호: NMC5-B356) 되었으며 이는 국내 생물 자 원의 다양성 확보 차원에서 의미가 있을 것으로 생각된다 (Table 3).
분해 효소 및 옥신 생산능 분석
토양 미생물들은 토양으로 유입되는 낙엽, 볏짚 등 유기 물들을 세포외 분해효소를 분비하여 영양물질 등으로 분해 하기 때문에 호르몬(hormone), 비타민(vitamin), 식물생장 촉진물질 등을 생산하여 식물의 생장을 돕는다고 알려져 있기에 분리된 호염성 미생물 균주들의 식품 산업에 적용 가능한 신규 분해 효소 탐색 자원 및 미생물비료로써의 산 업적 응용가능성을 확인하기 위하여 세포외 분해 효소 생 산능 및 옥신 생산능 분석을 실시하였다.
Table 3에 나타낸 것처럼 분리된 116 균주에서 24 균주 를 제외한 모든 균주에서 한 가지 이상의 분해 효소 활성 이 존재하는 것을 확인하였으며, 두 가지 이상의 효소 활 성을 가지는 균주 중 특히 세 가지 효소에 대하여 활성을 모두 가지고 있는 4균주(과제기탁번호: NMC5-B326, NMC5-B328, NMC5-B372, NMC6-B428)도 확인되었다. 또한 옥신 생산능을 확인한 결과, 17균주에서 생산능이 확 인되었으며 제주 양떼 목장 토양시료에서 5균주, 녹원 목 장 토양시료에서 12균주가 분리되었으며 아침미소 목장 토 양시료에서는 옥신 생산능을 가진 균주는 분리되지 않았 다. 이는 국내 목장 주변의 토양이 식물생장을 촉진시킬 수 있는 미생물비료 연구에 활용할 수 있는 미생물 탐색 소재로써 중요한 원천소재로써의 활용가치가 높을 것으로 예상되었다. 본 연구에서 수행한 결과들은 국내 미생물 생 물 자원의 다양성 확보 차원에서 큰 의미를 찾을 수 있을 것이며 국내 목장 주변 토양시료에서 분해 효소 탐색을 위 한 새로운 균주 탐색 가능성과 식품 산업용 효소 및 미생 물비료 연구의 기본 생물소재로 활용이 가능할 것으로 예 상된다. 또한 본 연구를 통하여 분리한 모든 균주들은 한 국생명공학연구원 미생물가치제고사업단에 기탁하였다.
요 약
본 연구는 국내 목장 토양 시료로부터 분리한 호염성 미 생물들의 다양성 및 특성에 관하여 조사하였다. 호염성 미 생물의 순수 분리를 위하여 marine agar 배지를 사용하였 으며 37°C에서 호기적으로 배양하였다. 순수 분리 후, 116 균주를 분리하였으며 16S rRNA 염기서열 분석 결과를 바 탕으로 계통학적 분석을 실시한 결과, 4문, 23과, 30속, 51 종으로 구성되어 있는 것을 확인하였다. 그리고 분리한 균 주들이 amylase, lipase, protease 같은 산업적으로 유용한 효소를 생산하는지 확인하기 위하여 효소 활성 평가를 실 시하였으며, 92균주가 최소 한 종류 이상의 효소 활성을 가지고 있는 것을 확인하였으며 세 가지 효소에 대하여 모 든 활성을 가지고 있는 균주도 4균주가 확인되었으며 auxin 생산능을 가진 균주도 17균주가 확인되었다. 이는 본 연구를 통하여 분리한 미생물들의 산업적 활용 가능성 을 나타내었다. 그러므로 이번 연구는 국내 유전자원 확보 및 염전 시료의 호염성 미생물의 다양성과 특성에 관한 과 학적 지식 확장에 도움이 될 것으로 생각된다.
감사의 글
이 논문은 2019년도 한국연구재단 이공분야기초연구사업 (NRF-2019R1F1A1060737)과 2016년도 바이오·의료기술개 발사업(NRF-2013M3A9A5076603)의 지원을 받아 수행된 연구임.
References
Chae HJ, In MJ, Kim MH.1997. Production and characteristic of enzymetically hydrolyzed soy sauce by the treatment using protease. J. Korean Soc. Food Sci. Nutr. 26: 784-787.
Felsenstein J.1985. Confidence limits on phylogenies: An approach using the bootstrap. Evolution. 39: 783-791.


Jeong KH, Seo JH, Jeong YJ.2005. Characteristics of soybean hydrolysates prepared with various protease. Korean J. Food Preserv. 12: 460-464.
Jung HK, Kim JR, Woo SM, Kim SC.2006. An auxin producing plant growth promoting rhizobacterium Bacillus subtilis AH18 which has siderophore-producing biocontrol activity. Korean J. Microbiol. Biotechnol. 34: 94-100.
Jung HK, Kim JR, Woo SM, Kim SD.2007. Selection of the auxin, siderophore, and cellulase-producing PGPR, Bacillus licheniformis K11 and its plant growth promoting mechanisms. J. Korean Soc. Appl. Biol. Chem. 50: 23-28.
Kim BG, Choi SY, Suh HJ, Park HJ.2011. Bitterness reduction and enzymatic transformation of ginsenosides from Korean red ginseng (Panax ginseng) extract. J. Food Biochem. 35: 1267-1282.

Kim DC, Lee TJ, In MJ.2019. Potential of proteolytic enzyme treatment for production of Korean red ginseng extract. J. Appl. Biol. Chem. 62: 385-389.

Kim SK, Park PJ, Kim KH.2000. Preparation of sauce from enzymetic hydrolysates of cod frame protein. J. Korean Soc. Food Sci. Nutr. 29: 635-641.
Mayuri S, Yogesh G, Shalini A, Vikas K, Anil P, Ashwani K.2019. A review on microbial alkaline protease: An essential tool for various industrial approaches. Ind. Biotechnol. 15: 69-78.

Park JH, Kim YM, Kim DS, Kim SM.2005. Functionality of low molecular weight peptides of accelerately manufactured anchovy sauce with Bacillus subtilis JM3 protease. Korean J. Food Sci. Technol. 37: 827-832.
Saitou N, Nei M.1987. The neighbor-joining method: A new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 4: 406-425.
Wu JL, Lu BJ, Du MH, Liu GM, Hara KJ, Su WJ, Cao MJ.2008. Purification and characterization of gelatinase-like proteinases from the dark muscle of common carp. J. Agr. Food Chem. 56: 2216-2222.