Article
다양한 염전으로부터 호기적 조건에서 분리된 미생물의 다양성 및 효소 생산능 분석에 관한 연구
이용직, 신기선1, 이상재2,*
A Study of the Diversity and Profile for Enzyme Production of Aerobically Cultured Halophilic Microorganisms from the Various Solar Salterns
Yong-Jik Lee, Kee-Sun Shin1, Sang-Jae Lee2,*
Department of Bio-Cosmetics, Seowon University
1Industrial Bio-materials Research Center, Korea Research Institute of Bioscience and Biotechnology (KRIBB)
2Major in Food Biotechnology and Research Center for Extremophiles & Marine Microbiology, Silla University
*Corresponding author: Sang-Jae Lee, Major in Food Biotechnology and Research Center for Extremophiles & Marine Microbiology, Silla University, 140 Baegyang-daero, 700beon-gil, Sasang-Gu, Busan 46958, Korea Tel : +82-51-999-5447; Fax : +82-51-999-5458 E-mail:
sans76@silla.ac.kr
© Korean Society for Food Engineering. All rights reserved.
Received: Jan 30, 2020; Revised: Feb 17, 2020; Accepted: Feb 18, 2020
Abstract
This research confirmed the diversity and characterization of halophilic microorganisms isolated from the various solar salterns, collected on the inside and outside of the country. To isolate strains, the marine agar medium was basically used and cultivated at 37°C for several days aerobically. After single colony isolation, a totally of 230 pure colonies were isolated and phylogenetic analysis was carried out based on the result of 16S rDNA sequencing, indicating that isolated strains were divided into 4 phyla, 12 families, 27 genera and 64 species. Firmicutes phylum, the main phyletic group, comprised 89.6% with 3 families, 17 genera and 52 species of Bacillaceae, Staphylococcaceae and Carnobacteriaceae. To confirm whether isolated strain can produce industrially useful enzyme or not, amylase, lipase, and protease enzyme assays were performed individually, showing that 177 strains possessed at least one enzyme activity. Especially 17 strains showed all enzyme activity tested. This result indicated that isolated strains have shown the possibility of the industrial application. Therefore, this study has contributed to securing domestic genetic resources and the expansion of scientific knowledge of the halophilic microorganisms community in solar salterns.
Keywords: Salt; Halophilic; Diversity; Extracellular enzyme; Bacteria
서 론
산업용 효소 시장에서 protease는 60% 이상의 가장 큰 비율로 사용되고 있으며 식품, 세제, 섬유·펄프 및 피혁공 업, 의약품 등에 이용되어 왔으며 근래에는 미생물로부터 다량 생산하여 이용하고 있다(Singh et al., 2016). Protease 는 효소 활성의 최적 pH에 따라 알카리성, 중성, 산성 protease로 구별되며, 알카리성 protease는 주로 세제산업에 활용되는데 Bacillus sp.와 같은 세균이나, Aspergillus sp. 와 같은 곰팡이가 생산한다. 지금까지 탐색 개발된 것은 주로 알카리성 또는 고온성 균주로서 호염성 protease는 아 직 개발된 예가 많지 않다. 최근 발표된 알카리성 protease 생산 균주인 Bacillus amyloliquefaciens CMB01 균주는 부 패된 쌀죽에서 분리한 균주로서 amylase 및 lipase 활성과 함께 세제용 효소 생산 균주로서 이용가능성이 높으나 염 류에 대한 영향은 그리 크지 않아서 적용범위가 한정적이 다(Ban et al., 2003). 따라서, 고농도의 염분을 함유한 염 전과 같은 환경에서 분리한 균주가 가지고 있는 호염성의 알카리 protease는 세제용으로뿐만 아니라 식품용으로도 그 적용범위를 넓혀 이용할 수가 있다(Mayuri et al., 2019).
호염성 미생물은 소금호수, 염전, 해양 등 염분 함량이 높은 자연환경에서 다양하게 존재하고 있으며 계통학적 다 양성뿐만 아니라 염내성 amylase, nuclease, protease의 개 발과 더불어 호염성 세균이 생산하는 β-carotene은 항산화 제와 식품 색소제등 다양한 바이오 산업 분야에서 응용되 면서 최근에는 염전에서 생육하는 호염미생물의 군집분 석(Baati et al., 2010; Beak, 1987; Birbir et al., 2007; Gibtan et al., 2017), 생화학(Oren, 2008) 및 유전학적 특성 구명(Pesenti et al., 2008; VijayAnand et al., 2010) 등 천 일염 유래 호염미생물의 산업화를 위한 호염성 세균 다양 성 확보를 위해 많은 연구자들의 관심 속에서 이와 관련하 여 활발하게 연구가 수행되고 있다. 따라서 본 연구에서는 국내 염전뿐만 아니라 에티오피아의 염전으로부터 호염성 미생물을 분리하고 전분가공용, 식품 산업용 및 세제용 산 업용 효소시장 등에 응용 가능한 효소 3종(amylase, lipase, protease)의 활성을 중심으로 분리된 미생물의 다양한 분해 효소 생산 특성 탐색과 함께 작물의 생육을 촉진하는 기능 을 함으로써 화학비료를 대체하는 미생물비료로써의 활용 을 위한 auxin 생산능 분석을 실시하였다. 이는 호염성 미 생물 자원의 가치를 제고하고 산업용 효소 관련 생물공학 및 미생물비료 연구의 기본 생물소재로 활용이 가능할 것 으로 예상된다.
재료 및 방법
호염성 세균 분리 및 배양
국내외 다양한 염전으로부터 호염성 미생물 분리를 위해 각각의 샘플을 멸균된 0.85% 생리식염수에 첨가하여 voltexing으로 현탁하였다. 현탁한 샘플 1 mL을 사용하여 10−1-10−4배로 단계희석 한 후, 일반 증식배지로 해양미생물 전용배지인 marine agar (BD, NJ, USA) 배지를 제작하여 희석액을 도말하여 37°C에서 호기적으로 호염미생물을 배 양하였다. 배양 후 선택적으로 배지상에 나타나는 균의 크 기, 모양, 색깔 등 형태학적 모습을 관찰한 후 동일한 고 체배지를 사용하여 추가적으로 single colony isolation을 수행하였다. 순수분리된 균주의 혼합배지에서의 생육 가능 성을 확인하기 위하여, nutrient agar (BD, NJ, USA), R2A agar (BD, NJ, USA), 및 tryptic soy agar (BD, NJ, USA) 에 평판 도말법을 이용하여 37°C에서 7일간 정치 배양을 하였다.
16S rDNA 염기서열의 계통학적 분석
국내외 다양한 염전으로부터 호기적 배양 조건에서 분리 된 균주들의 분자생물학적 동정을 위해 marine agar (BD, NJ, USA) 배지에 각각 분리된 균주의 colony가 배양된 상 태의 고체 배지를 ㈜바이오팩트에 보내어 16S rDNA 염기 서열의 분석을 의뢰하였다. 분석된 16S rDNA 염기서열로 부터 가장 유사한 근연 균주의 확인을 위하여 NCBI wensite의 BLAST search program (https://blast.ncbi.nlm. nih.gov/Blast.cgi)과 ㈜천랩의 Ezbiocloud (https://www. ezbiocloud.net/)를 사용하였다. 계통학적 분석은 Clustal W 및 Mega 6.0 프로그램을 이용하여 확인하였다.
분해 효소 생산능 분석
분리된 호염성 미생물의 세포외 분해 효소 amylase, lipase, protease 생산능 확인을 위하여 각각의 효소와 특이 적으로 반응할 기질 성분이 포함된 고체평판 선별배지를 사용하였다. 먼저 amylase 생산능은 0.2% soluble starch (BD, NJ, USA)를, lipase 생산능은 1% Tween 80 (Sigma, MO, USA)을, protease 생산능은 2% skim milk (BD, NJ, USA)를 기질로 선택하여 marine agar (BD, NJ, USA) 배 지에 각각 첨가하여 제조하였으며 분리된 균주를 직접 접 종하여 37°C에서 7일 배양한 후 투명환(Clear zone)의 직 경으로 조사하였다. 분리된 균주의 효소활성 분해능 평가 는 배양 후 나타나는 접종균 주위의 투명환의 크기(+++ : > 7 mm, ++ : 4~6 mm, + : 1~3 mm)로 나타내었다.
옥신(Auxin) 생산능 분석
분리된 균주의 auxin 생산능은 0.1% L-tryptophan이 첨가된 marin broth (BD, NJ, USA) 배지에 순수분리된 colony를 tooth-picking 한 후 37°C에서 5일 배양하여 Salkowski 시약(35 % HClO4 50 mL + 0.5 M FeCl3 1 mL) 800 uL을 배양 상등액 400 uL에 섞어준 후 어두운 곳에서 30 min 반응시켜 육안으로 확인하여 붉은 색깔 변화 정도 에 따라 옥신 생성능 정도를 표시하였다(짙은 붉은색: +++; 일반 붉은색: ++; 옅은 붉은색: +; 주황색: w; 무색: -).
결과 및 고찰
호염성 세균 분리
국내외 염전에서 산업적 활용이 용이할 것으로 생각되는 호기적으로 생육 가능한 호염성 미생물을 분리하고자 marine agar 배지에 시료를 희석, 도말하여 배양한 후, 배 양된 colony의 모양, 색깔 등 형태학적 특징을 육안으로 구분이 가능한 균주들을 대상으로 동일한 고체배지를 사용 하여 2차로 단일 균주 분리를 수행하였다. 그 결과 Table 1에서 나타낸 것처럼 국내 신안 토판염과 장판염에서는 100균주와 26균주가 각각 분리되었으며 국내 곰소 타일염 에서는 36균주가 분리되었다. 그리고 국외 염전인 에티오 피아 rock salt와 lake salt에서는 각각 54균주와 14균주가 분리되었으며 본 실험을 통하여 호염성 미생물 총 230 균 주를 순수 분리하였다. 국내 염전에서 분리된 균주의 경우 상대적으로 토판염에서 많은 호염성 미생물이 분리되었는 데 이는 염전에 사용한 바닥소재에 따라 천일염은 토판염, 장판염, 타일염으로 구분되는데 장판염이나 타일염은 가소 성소재나 타일을 이용한 바닥에서 생산된 천일염이며 토판 염은 갯벌을 단단하게 다진 결정지 판 위에서 생산되는 천 일염이기에 갯벌에서 생육하는 다양한 호염성 미생물 균주 가 함께 분리된 것으로 예상된다(Park et al., 2000; Lee et al., 2007). 또한 주요성분이 무기염으로 이루어진 marine agar 배지는 해양미생물 배양에 유리한 배지이기에 산업적 활용가능성을 확인하기 위하여 대량 배양 등에 많이 활용 되는 혼합 배지(nutrient agar, R2A agar, tryptic soy agar) 에서의 분리 균주들의 생육 가능성을 확인한 결과 230균 주 중 62균주(26.9%)가 최소 1종류 이상의 혼합 배지에서 생육이 가능(weak growth 포함)한 것을 확인하였다. 이는 호염성 미생물세균의 탐색에 있어서 분리 배지의 구성성분 이 어느 정도 영향을 미치는 것으로 생각되며, 본 연구 결 과를 바탕으로 호염성 미생물 분리를 위한 최적 배지는 marine agar 배지로 나타났다. 또한 최적의 생육 pH 조건 을 확인하기 위하여 pH를 5, 7, 9로 각각 조절한 marine agar 배지에 분리 균주들의 생육을 확인해본 결과 분리된 모든 균주는 pH 7에서 생육이 가능하였으며 이중 19균주 는 pH 5에서도 생육이 가능(weak growth 포함)하였으며 pH 9에서 생육 가능(weak growth 포함)한 균주는 209균주 였다(Table 1).
Table 1.
Isolation and identification of aerobically cultured halophilic microorganisms isolated from the commercial salts
|
No. |
Source |
Isolate number |
NA# |
R2A |
TSA |
MA* |
|
pH5 |
pH7 |
pH9 |
|
1 |
|
KTB062 |
-c |
- |
- |
- |
+a |
+ |
|
2 |
|
KTB091 |
- |
- |
- |
- |
+ |
+ |
|
3 |
|
KTB151 |
- |
+ |
- |
- |
+ |
+ |
|
4 |
|
KTB274 |
- |
- |
- |
- |
+ |
+ |
|
5 |
|
KTB094 |
- |
- |
- |
- |
+ |
+ |
|
6 |
|
KTB064 |
- |
- |
- |
- |
+ |
+ |
|
7 |
|
KTB093 |
- |
- |
- |
- |
+ |
+ |
|
8 |
|
KTB012 |
- |
- |
- |
- |
+ |
+ |
|
9 |
|
KTB052 |
- |
- |
- |
- |
+ |
+ |
|
10 |
|
KTB131 |
- |
- |
- |
- |
+ |
+ |
|
11 |
|
KTB254 |
- |
- |
- |
- |
+ |
+ |
|
12 |
|
KTB021 |
- |
- |
- |
- |
+ |
+ |
|
13 |
|
KTB102 |
- |
- |
- |
- |
+ |
+ |
|
14 |
|
KTB041 |
- |
- |
- |
- |
+ |
+ |
|
15 |
|
KTB241 |
- |
- |
- |
- |
+ |
+ |
|
16 |
|
KTB101 |
- |
- |
- |
- |
+ |
+ |
|
17 |
|
KTB202 |
- |
+ |
- |
- |
+ |
+ |
|
18 |
|
KTB051 |
- |
+ |
- |
- |
+ |
+ |
|
19 |
|
KTB092 |
+ |
+ |
- |
- |
+ |
+ |
|
20 |
|
KTB061 |
- |
- |
+ |
- |
+ |
+ |
|
21 |
|
KTB201 |
- |
- |
- |
- |
+ |
+ |
|
22 |
|
12ST1-1 |
- |
- |
- |
- |
+ |
+ |
|
23 |
|
12ST1-2 |
- |
- |
- |
- |
+ |
+ |
|
24 |
|
12ST1-3 |
+ |
+ |
+ |
- |
+ |
+ |
|
25 |
신안 토판염 |
12ST1-4 |
- |
- |
- |
- |
+ |
+ |
|
26 |
|
12ST1-5 |
- |
- |
- |
- |
+ |
+ |
|
27 |
|
12ST1-6 |
- |
- |
- |
- |
+ |
+ |
|
28 |
|
12ST1-7 |
- |
- |
- |
- |
+ |
+ |
|
29 |
|
12ST1-8 |
- |
- |
- |
- |
+ |
+ |
|
30 |
|
12ST1-9 |
+ |
+ |
+ |
- |
+ |
+ |
|
31 |
|
12ST1-10 |
+ |
+ |
+ |
- |
+ |
+ |
|
32 |
|
12ST1-11 |
- |
- |
- |
- |
+ |
+ |
|
33 |
|
12ST1-12 |
- |
- |
- |
- |
+ |
+ |
|
34 |
|
12ST1-13 |
- |
- |
- |
- |
+ |
+ |
|
35 |
|
12ST1-14 |
- |
- |
- |
- |
+ |
+ |
|
36 |
|
12ST1-15 |
- |
- |
- |
- |
+ |
+ |
|
37 |
|
12ST1-16 |
- |
- |
- |
- |
+ |
+ |
|
38 |
|
12ST1-17 |
- |
- |
- |
- |
+ |
+ |
|
39 |
|
12ST1-18 |
+ |
+ |
+ |
- |
+ |
+ |
|
40 |
|
12ST1-19 |
- |
- |
- |
- |
+ |
+ |
|
41 |
|
12ST1-20 |
- |
- |
- |
- |
+ |
+ |
|
42 |
|
12ST2-1 |
- |
- |
- |
- |
+ |
+ |
|
43 |
|
12ST2-2 |
- |
- |
- |
- |
+ |
+ |
|
44 |
|
12ST2-3 |
- |
- |
- |
- |
+ |
+ |
|
45 |
|
12ST2-4 |
- |
- |
- |
- |
+ |
+ |
|
46 |
|
12ST2-5 |
- |
wb |
- |
- |
+ |
+ |
|
47 |
|
12ST2-6 |
- |
- |
- |
- |
+ |
+ |
|
48 |
|
12ST2-7 |
- |
- |
- |
- |
+ |
+ |
|
49 |
|
12ST2-8 |
+ |
+ |
+ |
- |
+ |
+ |
|
50 |
|
12ST2-9 |
- |
- |
- |
- |
+ |
+ |
|
51 |
|
12ST2-10 |
- |
- |
- |
- |
+ |
+ |
|
52 |
|
12ST2-13 |
- |
- |
- |
- |
+ |
+ |
|
52 |
|
12ST2-13 |
- |
- |
- |
- |
+ |
+ |
|
53 |
|
12ST2-14 |
- |
- |
- |
- |
+ |
+ |
|
54 |
|
12ST2-15 |
- |
- |
- |
- |
+ |
+ |
|
55 |
|
12ST2-16 |
- |
- |
- |
- |
+ |
+ |
|
56 |
|
12ST2-18 |
- |
- |
- |
- |
+ |
+ |
|
57 |
|
12ST2-19 |
- |
- |
- |
- |
+ |
+ |
|
58 |
|
12ST2-20 |
- |
- |
- |
- |
+ |
+ |
|
59 |
|
13ST1-1 |
- |
- |
- |
- |
+ |
+ |
|
60 |
|
13ST1-2 |
- |
- |
- |
w |
+ |
+ |
|
61 |
|
13ST1-3-1 |
- |
- |
- |
- |
+ |
+ |
|
62 |
|
13ST1-4 |
- |
- |
- |
w |
+ |
+ |
|
63 |
|
13ST1-5 |
+ |
+ |
+ |
- |
+ |
+ |
|
64 |
|
13ST1-7 |
- |
- |
- |
- |
+ |
+ |
|
65 |
|
13ST1-8 |
- |
- |
w |
w |
+ |
+ |
|
66 |
|
13ST1-9 |
- |
- |
w |
w |
+ |
+ |
|
67 |
|
13ST1-10 |
+ |
+ |
+ |
- |
+ |
+ |
|
68 |
|
13ST1-11 |
- |
- |
- |
- |
+ |
+ |
|
69 |
|
13ST1-12 |
- |
- |
- |
- |
+ |
+ |
|
70 |
|
13ST1-13 |
- |
- |
- |
- |
+ |
+ |
|
71 |
|
13ST1-14 |
- |
- |
- |
- |
+ |
+ |
|
72 |
|
13ST1-15 |
- |
- |
- |
- |
+ |
+ |
|
73 |
|
13ST1-16 |
- |
- |
- |
- |
+ |
+ |
|
74 |
신안 토판염 |
13ST1-17 |
- |
- |
- |
- |
+ |
+ |
|
75 |
13ST1-18 |
+ |
- |
+ |
- |
+ |
+ |
|
76 |
|
13ST1-19 |
- |
w |
- |
- |
+ |
+ |
|
77 |
|
13ST1-20 |
- |
- |
- |
- |
+ |
+ |
|
78 |
|
13ST2-1 |
- |
- |
- |
- |
+ |
+ |
|
79 |
|
13ST2-2 |
- |
- |
- |
- |
+ |
+ |
|
80 |
|
13ST2-3 |
- |
- |
- |
- |
+ |
+ |
|
81 |
|
13ST2-4 |
- |
+ |
- |
- |
+ |
+ |
|
82 |
|
13ST2-5 |
+ |
+ |
+ |
- |
+ |
+ |
|
83 |
|
13ST2-6 |
+ |
- |
- |
- |
+ |
+ |
|
84 |
|
13ST2-7 |
- |
- |
- |
- |
+ |
+ |
|
85 |
|
13ST2-8 |
+ |
- |
+ |
- |
+ |
+ |
|
86 |
|
13ST2-9 |
- |
- |
- |
- |
+ |
+ |
|
87 |
|
13ST2-11 |
- |
- |
- |
- |
+ |
+ |
|
88 |
|
13ST2-12-1 |
- |
- |
- |
- |
+ |
+ |
|
89 |
|
13ST2-13 |
- |
+ |
- |
- |
+ |
+ |
|
90 |
|
13ST2-14 |
- |
- |
- |
- |
+ |
+ |
|
91 |
|
13ST2-15 |
- |
- |
- |
- |
+ |
+ |
|
92 |
|
13ST2-16 |
- |
- |
- |
- |
+ |
+ |
|
93 |
|
13ST2-18 |
- |
- |
- |
- |
+ |
+ |
|
94 |
|
13ST2-19 |
- |
- |
- |
- |
+ |
+ |
|
95 |
|
13ST2-20 |
- |
- |
- |
- |
+ |
+ |
|
96 |
|
KTB011 |
- |
w |
w |
- |
+ |
+ |
|
97 |
|
KTB022 |
- |
- |
- |
- |
+ |
+ |
|
98 |
|
KTB023 |
- |
- |
w |
- |
+ |
+ |
|
99 |
|
KTB081 |
- |
- |
w |
- |
+ |
+ |
|
100 |
|
KTB272 |
- |
- |
w |
- |
+ |
+ |
|
101 |
|
ST3rd#203 |
- |
+ |
+ |
- |
+ |
+ |
|
102 |
|
ST2nd#107 |
- |
- |
- |
- |
+ |
+ |
|
103 |
|
ST2nd#108 |
- |
- |
+ |
- |
+ |
+ |
|
104 |
|
ST3rd#201 |
- |
- |
- |
- |
+ |
+ |
|
105 |
|
ST3rd#205 |
- |
- |
+ |
- |
+ |
+ |
|
106 |
|
ST3rd#239 |
- |
- |
- |
- |
+ |
+ |
|
107 |
|
ST2nd#109 |
- |
- |
- |
- |
+ |
+ |
|
108 |
|
ST2nd#110 |
- |
- |
- |
- |
+ |
+ |
|
109 |
|
ST3rd#210 |
- |
- |
- |
- |
+ |
+ |
|
110 |
|
ST3rd#212 |
- |
- |
- |
- |
+ |
+ |
|
111 |
|
ST3rd#222 |
+ |
+ |
+ |
- |
+ |
+ |
|
112 |
|
ST3rd#229 |
+ |
+ |
+ |
- |
+ |
+ |
|
113 |
신안 장판염 |
13SJ1-1 |
+ |
+ |
+ |
- |
+ |
+ |
|
114 |
13SJ2-1 |
- |
- |
- |
- |
+ |
+ |
|
115 |
|
14SJ1-1 |
- |
- |
- |
- |
+ |
+ |
|
116 |
|
14SJ1-2 |
- |
- |
- |
- |
+ |
+ |
|
117 |
|
ST2nd#101 |
- |
- |
- |
w |
+ |
+ |
|
118 |
|
ST3rd#204 |
- |
- |
w |
w |
+ |
+ |
|
119 |
|
ST3rd#225 |
w |
- |
- |
w |
+ |
+ |
|
120 |
|
ST3rd#228 |
w |
- |
w |
w |
+ |
+ |
|
121 |
|
ST3rd#231 |
+ |
+ |
+ |
+ |
+ |
+ |
|
122 |
|
ST3rd#238 |
+ |
+ |
- |
w |
+ |
+ |
|
123 |
|
ST3rd#241-1 |
- |
- |
- |
+ |
+ |
+ |
|
124 |
|
ST3rd#242 |
- |
- |
- |
w |
+ |
+ |
|
125 |
|
STCP#202 |
- |
w |
- |
w |
+ |
+ |
|
126 |
|
STCP#209 |
+ |
+ |
- |
+ |
+ |
+ |
|
127 |
|
GL1-1 |
- |
- |
- |
- |
+ |
+ |
|
128 |
|
GL1-2 |
- |
- |
- |
- |
+ |
+ |
|
129 |
|
GT1-1 |
- |
- |
- |
- |
+ |
+ |
|
130 |
|
GT1-2 |
- |
- |
- |
- |
+ |
+ |
|
131 |
|
GT1-3 |
- |
- |
- |
- |
+ |
+ |
|
132 |
|
GT1-4 |
- |
- |
- |
- |
+ |
+ |
|
133 |
|
GT1-5 |
- |
- |
- |
- |
+ |
+ |
|
134 |
|
GT1-6 |
- |
- |
- |
- |
+ |
+ |
|
135 |
|
GT1-7-1 |
- |
- |
- |
- |
+ |
+ |
|
136 |
|
GT1-8 |
- |
- |
- |
- |
+ |
+ |
|
137 |
|
GT1-9 |
- |
+ |
- |
- |
+ |
+ |
|
138 |
곰소 타일염 |
GT1-10 |
- |
- |
- |
- |
+ |
+ |
|
139 |
GT1-11 |
- |
- |
- |
- |
+ |
+ |
|
140 |
|
GT1-12 |
- |
- |
- |
- |
+ |
+ |
|
141 |
|
GT1-13 |
- |
- |
- |
- |
+ |
+ |
|
142 |
|
GT2-1 |
+ |
- |
+ |
- |
+ |
+ |
|
143 |
|
GT2-2 |
- |
- |
- |
- |
+ |
+ |
|
144 |
|
GT2-3 |
- |
- |
- |
- |
- |
w |
|
145 |
|
GT2-4 |
- |
- |
- |
- |
+ |
+ |
|
146 |
|
GT2-5 |
- |
+ |
- |
- |
+ |
+ |
|
147 |
|
GT2-6 |
- |
- |
- |
- |
+ |
- |
|
148 |
|
GT2-7 |
- |
+ |
- |
- |
+ |
+ |
|
149 |
|
GT2-9 |
- |
- |
- |
- |
+ |
+ |
|
150 |
|
GT2-10 |
- |
- |
- |
- |
+ |
+ |
|
151 |
|
GS-RS 1A |
+ |
+ |
+ |
- |
+ |
+ |
|
152 |
|
GS-RS 5A |
+ |
+ |
+ |
- |
+ |
w |
|
153 |
|
GS-RS 1D |
+ |
+ |
+ |
- |
+ |
- |
|
154 |
|
GS-RS 2D |
w |
w |
+ |
- |
+ |
- |
|
155 |
|
GS-RS 4D |
+ |
+ |
+ |
- |
+ |
- |
|
156 |
곰소 타일염 |
GS-RS 5D |
+ |
+ |
+ |
- |
+ |
- |
|
157 |
GS-OS 1A |
+ |
+ |
+ |
- |
+ |
- |
|
158 |
|
GS-OS 2A |
- |
+ |
- |
- |
+ |
- |
|
159 |
|
GS-OS 6A |
- |
- |
- |
- |
+ |
+ |
|
160 |
|
GS-OS 7A |
+ |
+ |
+ |
- |
+ |
- |
|
161 |
|
GS-OS 4B |
+ |
+ |
+ |
- |
+ |
+ |
|
162 |
|
GS-OS 6B |
+ |
+ |
+ |
- |
+ |
- |
|
163 |
|
ER1-1 |
- |
- |
+ |
- |
+ |
- |
|
164 |
|
ER1-2 |
- |
- |
+ |
- |
+ |
- |
|
165 |
|
ER1-3 |
- |
w |
w |
- |
+ |
+ |
|
166 |
|
ER1-4 |
- |
w |
w |
- |
+ |
+ |
|
167 |
|
ER1-5 |
- |
- |
- |
- |
+ |
+ |
|
168 |
|
ER1-6 |
- |
- |
- |
- |
+ |
+ |
|
169 |
|
ER1-7 |
- |
- |
- |
- |
+ |
+ |
|
170 |
|
ER1-8 |
- |
- |
- |
- |
+ |
+ |
|
171 |
|
ER1-9 |
- |
- |
- |
- |
+ |
+ |
|
172 |
|
ER1-10 |
- |
- |
- |
- |
+ |
+ |
|
173 |
|
ER1-11 |
- |
- |
- |
- |
+ |
+ |
|
174 |
|
ER2-1 |
- |
- |
- |
- |
w |
w |
|
175 |
|
ER2-3 |
- |
- |
w |
- |
+ |
+ |
|
176 |
|
ER2-4 |
- |
- |
w |
- |
+ |
+ |
|
177 |
|
ER2-5 |
- |
- |
+ |
- |
+ |
+ |
|
178 |
|
ER2-6 |
- |
w |
+ |
- |
+ |
+ |
|
179 |
|
ER2-7 |
- |
w |
+ |
- |
+ |
+ |
|
180 |
|
ER2-8 |
- |
- |
- |
- |
+ |
+ |
|
181 |
에티오피아 |
ER2-9 |
- |
w |
+ |
- |
+ |
- |
|
182 |
Rock salt |
ER2-10 |
- |
w |
+ |
- |
+ |
- |
|
183 |
|
R450201 |
+ |
+ |
+ |
- |
+ |
- |
|
184 |
|
R450202 |
- |
- |
- |
w |
+ |
- |
|
185 |
|
R450203 |
+ |
+ |
+ |
w |
+ |
- |
|
186 |
|
R450204 |
- |
- |
- |
w |
+ |
- |
|
187 |
|
R450205 |
+ |
+ |
+ |
- |
+ |
- |
|
188 |
|
R450206 |
+ |
+ |
+ |
- |
+ |
- |
|
189 |
|
R450301 |
+ |
+ |
+ |
- |
+ |
- |
|
190 |
|
R450302 |
- |
+ |
+ |
- |
+ |
- |
|
191 |
|
R450303 |
- |
- |
w |
- |
+ |
- |
|
192 |
|
R450304-1 |
- |
+ |
w |
- |
+ |
- |
|
193 |
|
R450305 |
+ |
+ |
+ |
- |
+ |
- |
|
194 |
|
R450306 |
+ |
+ |
- |
- |
+ |
- |
|
195 |
|
R450307 |
+ |
+ |
+ |
- |
+ |
- |
|
196 |
|
R450308 |
+ |
+ |
+ |
- |
+ |
w |
|
197 |
|
R450401 |
+ |
+ |
+ |
- |
+ |
w |
|
198 |
|
R450402 |
+ |
+ |
+ |
- |
+ |
- |
|
199 |
|
R450403 |
- |
+ |
+ |
- |
+ |
- |
|
200 |
|
R450405 |
+ |
+ |
+ |
- |
+ |
+ |
|
201 |
|
R450408 |
- |
- |
- |
- |
+ |
- |
|
202 |
|
R450409-1 |
- |
- |
- |
- |
+ |
w |
|
203 |
|
R450501 |
- |
- |
- |
- |
+ |
w |
|
204 |
|
R450502 |
- |
- |
- |
- |
+ |
w |
|
205 |
|
R450503 |
- |
- |
- |
- |
+ |
- |
|
206 |
|
R450504 |
- |
- |
- |
- |
+ |
w |
|
207 |
|
R450505 |
- |
- |
- |
- |
+ |
- |
|
208 |
에티오피아 |
R450506-1 |
- |
- |
- |
- |
+ |
- |
|
209 |
Rock salt |
R450507-1 |
- |
- |
- |
w |
+ |
w |
|
210 |
|
ERB012 |
- |
- |
- |
- |
+ |
+ |
|
211 |
|
ERB032 |
- |
- |
+ |
- |
+ |
+ |
|
212 |
|
ERB054 |
- |
- |
|
- |
+ |
+ |
|
213 |
|
ERB061 |
- |
- |
- |
- |
+ |
+ |
|
214 |
|
ERB081 |
- |
- |
+ |
- |
+ |
+ |
|
215 |
|
ERB082 |
- |
- |
- |
w |
+ |
+ |
|
216 |
|
ERB112 |
- |
- |
- |
- |
+ |
+ |
|
217 |
|
EL1-1 |
- |
- |
+ |
- |
+ |
+ |
|
218 |
|
EL1-2 |
- |
- |
w |
- |
+ |
+ |
|
219 |
|
EL1-4 |
- |
w |
w |
- |
+ |
+ |
|
220 |
|
EL1-5 |
- |
- |
+ |
- |
+ |
- |
|
221 |
|
EL1-6 |
- |
- |
w |
- |
+ |
+ |
|
222 |
|
EL1-7 |
- |
w |
w |
- |
+ |
+ |
|
223 |
에티오피아 |
EL1-8 |
- |
- |
+ |
- |
+ |
- |
|
224 |
Lake salt |
EL2-1 |
- |
- |
+ |
- |
+ |
+ |
|
225 |
|
EL2-2 |
- |
- |
+ |
- |
+ |
+ |
|
226 |
|
EL2-4 |
- |
- |
- |
- |
+ |
+ |
|
227 |
|
EL2-5 |
- |
- |
+ |
- |
+ |
+ |
|
228 |
|
EL2-7 |
- |
- |
+ |
- |
+ |
+ |
|
229 |
|
EL2-9 |
- |
- |
+ |
- |
+ |
+ |
|
230 |
|
EL2-10 |
- |
- |
+ |
- |
+ |
+ |
Download Excel Table
16S rDNA 염기서열의 계통학적 분석
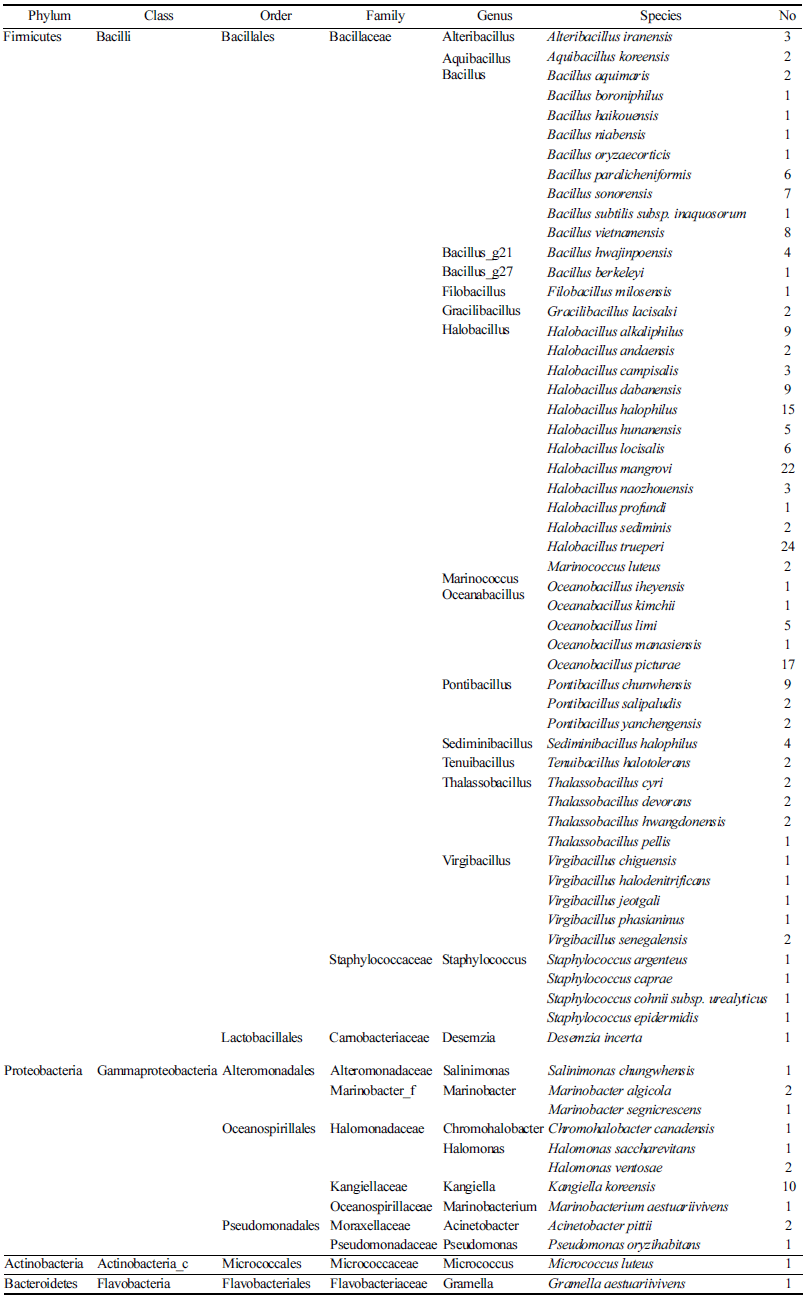
국내외 다양한 염전으로부터 호기적으로 분리된 230균주 의 16S rDNA 염기서열을 바탕으로 NBLAST program과 Ezbiocloud를 사용하여 미생물 동정을 실시한 결과 크게 4 문 7목 12과 27속 64종으로 나타났으며(Table 2), 분리 균 주와 근연 균주 및 상동성을 Table 3에 나타내었다. 또한 분리 동정된 균주들 간의 유연관계를 확인하기 위하여 계 통수를 작성하였다(결과 미제시). Table 2에서 보는 바와 같이 Firmicutes (Bacilli)가 89.6%로 가장 우점도가 높았고, Proteobacteria (Gammaproteobacteria)가 9.6%, Actinobaceria 와 Bacteroidetes는 각각 0.4%로 나타났다. Firmicutes 문은 Bacillaceae 97.6%, Staphylococcaceae 1.9%, Carnobacteriaceae 0.5%로 구성되었으며, 총 3과로 17속 52종이 분리되었 다. 그리고 약 9.56%가 분리된 Proteobacteria (Gammaproteobacteria) 문에서는 Kangiellaceae 45.5%, Halomonadaceae 18.2%, Marinobacter_f 13.6%, Moraxellaceae 9.1%, 그리고 Alteromonadaceae, Oceanospirillaceae와 Pseudomonadaceae 가 각각 4.5%로 구성되었으며, 총 7과로 8속 10종이 분리 되었다. Actinobateria 문과 Firmicutes 문에서 Micrococcus 속과 Gramella 속 1종씩이 분리되었다. 본 연구에서 가장 많이 분리된 Bacillaceae 과는 Halobacillus sp.이 염전에서 많이 분리된다는 것이 알려져 있기에 본 연구에서도 국내 외 다양한 염전 시료에서 43.9%로 가장 많이 분리된 것으 로 예상되었으며(Na et al., 2011) Photobacterium 속, Vibrio 속과 같은 어류병원체 관련 균주는 분리되지 않았 다. 그리고 국외 염전 시료에서만 Oceanobacillus sp. 25 균주가 분리되었는데 이는 분리원이 위치한 지역이 지구상 에서 가장 평균기온이 높은 곳이며 우수한 내염성을 가진 균주로 알려져 있기에 고온균이며 내염성이 우수한 균주의 분리가능성을 나타냈다(Morell, 2005; Lu et al., 2001). 또 한 분리 균주 중 근연 균주와의 16S rDNA 염기서열 상동 성이 낮은 10균주(96% 이하)는 신종 균주 가능성을 나타 내는 것으로 국외 염전 시료에서 6균주와 국내 염전 시료 에서 4균주(과제기탁번호: NMC5-B330, NMC6-B431, NMC6-B408, NMC4-B516)가 분리되었으며 국내에서 분리 된 균주들은 모두 국내 생물 자원의 다양성 확보 차원에서 큰 의미가 있을 것으로 생각된다.
Table 2.
Phylum analysis of aerobically cultured halophilic microorganisms isolated from the commercial salts
Download Excel Table
Table 3.
Representative sequences of an aerobically isolated halophilic strains from the commercial salts
|
No |
Isolate Name |
Closed species |
Closed strain number |
Similarity (%) |
Extracellular enzyme activity |
Auxin |
Deposited number |
|
Amylase |
Lipase |
Protease |
|
1 |
KTB062 |
Halobacillus trueperi
|
DSM 10404 |
99.46 |
-a |
- |
- |
w |
NMC6-B390 |
|
2 |
KTB091 |
Halobacillus dabanensis
|
D-8 |
97.46 |
- |
- |
- |
w |
NMC6-B391 |
|
3 |
KTB151 |
Halobacillus alkaliphilus
|
FP5 |
99.57 |
- |
- |
- |
w |
NMC6-B392 |
|
4 |
KTB274 |
Halobacillus trueperi
|
DSM 10404 |
99.31 |
- |
- |
- |
w |
NMC6-B393 |
|
5 |
KTB094 |
Halobacillus trueperi
|
DSM 10404 |
99.35 |
- |
++ |
- |
w |
NMC6-B394 |
|
6 |
KTB064 |
Halobacillus trueperi
|
DSM 10404 |
99.45 |
- |
- |
- |
w |
NMC6-B395 |
|
7 |
KTB093 |
Halobacillus trueperi
|
DSM 10404 |
99.68 |
- |
- |
- |
w |
NMC6-B396 |
|
8 |
KTB012 |
Halobacillus trueperi
|
DSM 10404 |
99.66 |
- |
- |
- |
w |
NMC6-B397 |
|
9 |
KTB052 |
Halobacillus trueperi
|
DSM 10404 |
99.79 |
- |
- |
- |
w |
NMC6-B398 |
|
10 |
KTB131 |
Halobacillus mangrovi
|
MS10 |
99.04 |
- |
- |
- |
w |
NMC6-B399 |
|
11 |
KTB254 |
Halobacillus mangrovi
|
MS10 |
99.30 |
- |
- |
- |
w |
NMC6-B400 |
|
12 |
KTB021 |
Halobacillus mangrovi
|
MS10 |
99.47 |
- |
- |
- |
w |
NMC6-B401 |
|
13 |
KTB102 |
Halobacillus alkaliphilus
|
FP5 |
99.28 |
- |
- |
- |
- |
NMC6-B402 |
|
14 |
KTB041 |
Halobacillus alkaliphilus
|
FP5 |
99.28 |
- |
- |
- |
- |
NMC6-B403 |
|
15 |
KTB241 |
Halobacillus trueperi
|
DSM 10404 |
99.78 |
- |
+ |
- |
w |
NMC6-B404 |
|
16 |
KTB101 |
Halobacillus alkaliphilus
|
FP5 |
99.45 |
- |
- |
- |
- |
NMC6-B405 |
|
17 |
KTB202 |
Halobacillus alkaliphilus
|
FP5 |
99.59 |
- |
- |
- |
- |
NMC6-B406 |
|
18 |
KTB051 |
Halobacillus alkaliphilus
|
FP5 |
97.09 |
- |
- |
- |
- |
NMC6-B407 |
|
19 |
KTB092 |
Halobacillus alkaliphilus
|
FP5 |
99.02 |
- |
- |
- |
- |
NMC6-B412 |
|
20 |
KTB061 |
Halobacillus trueperi
|
DSM 10404 |
99.43 |
- |
++ |
- |
w |
NMC6-B417 |
|
21 |
KTB201 |
Halobacillus alkaliphilus
|
FP5 |
97.69 |
- |
- |
- |
- |
NMC6-B418 |
|
22 |
12ST1-1 |
Halobacillus halophilus
|
DSM 2266 |
97.87 |
+++ |
- |
- |
- |
NMC5-B320 |
|
23 |
12ST1-2 |
Pontibacillus chungwhensis
|
BH030062 |
99.34 |
+++ |
- |
- |
- |
NMC5-B321 |
|
24 |
12ST1-3 |
Bacillus vietnamensis
|
15-1 |
98.32 |
+++ |
- |
- |
- |
NMC5-B322 |
|
25 |
12ST1-4 |
Halobacillus trueperi
|
DSM 10404 |
98.17 |
+++ |
- |
- |
- |
NMC5-B323 |
|
26 |
12ST1-5 |
Halobacillus mangrovi
|
MS10 |
99.06 |
+++ |
- |
- |
- |
NMC5-B324 |
|
27 |
12ST1-6 |
Halobacillus mangrovi
|
MS10 |
99.05 |
+++ |
- |
- |
- |
NMC5-B325 |
|
28 |
12ST1-7 |
Halobacillus mangrovi
|
MS10 |
98.58 |
+++ |
- |
- |
- |
NMC5-B326 |
|
29 |
12ST1-8 |
Halobacillus campisalis
|
ASL-17 |
98.71 |
+++ |
- |
- |
- |
NMC5-B327 |
|
30 |
12ST1-9 |
Bacillus vietnamensis
|
15-1 |
98.62 |
+++ |
- |
- |
- |
NMC5-B328 |
|
31 |
12ST1-10 |
Bacillus hwajinpoensis
|
SW-72 |
96.89 |
+ |
- |
- |
- |
NMC5-B329 |
|
32 |
12ST1-11 |
Bacillus oryzaecorticis
|
R1 |
92.17 |
+++ |
- |
- |
- |
NMC5-B330 |
|
33 |
12ST1-12 |
Halobacillus mangrovi
|
MS10 |
98.49 |
+++ |
- |
- |
- |
NMC5-B331 |
|
34 |
12ST1-13 |
Halobacillus trueperi
|
DSM 10404 |
98.90 |
+++ |
- |
- |
- |
NMC5-B332 |
|
35 |
12ST1-14 |
Thalassobacillus devorans
|
G-19.1 |
97.23 |
+++ |
+++ |
- |
- |
NMC5-B333 |
|
36 |
12ST1-15 |
Halobacillus trueperi
|
DSM 10404 |
98.00 |
+++ |
- |
+++ |
- |
NMC5-B334 |
|
37 |
12ST1-16 |
Halobacillus halophilus
|
DSM 2266 |
98.63 |
+++ |
- |
- |
- |
NMC5-B335 |
|
38 |
12ST1-17 |
Pontibacillus chungwhensis
|
BH030062 |
99.14 |
++ |
- |
+++ |
- |
NMC5-B336 |
|
39 |
12ST1-18 |
Bacillus vietnamensis
|
15-1 |
98.62 |
+++ |
- |
- |
- |
NMC5-B337 |
|
40 |
12ST1-19 |
Halobacillus halophilus
|
DSM 2266 |
98.81 |
+++ |
- |
- |
- |
NMC5-B338 |
|
41 |
12ST1-20 |
Halobacillus trueperi
|
DSM 10404 |
98.34 |
+++ |
- |
- |
- |
NMC5-B339 |
|
42 |
12ST2-1 |
Halobacillus halophilus
|
DSM 2266 |
98.42 |
+++ |
- |
+++ |
- |
NMC5-B340 |
|
43 |
12ST2-2 |
Thalassobacillus cyri
|
CCM7597 |
99.36 |
+++ |
- |
- |
- |
NMC5-B341 |
|
44 |
12ST2-3 |
Halobacillus mangrovi
|
MS10 |
98.03 |
+++ |
- |
- |
- |
NMC5-B342 |
|
45 |
12ST2-4 |
Halobacillus halophilus
|
DSM 2266 |
98.44 |
+++ |
- |
- |
- |
NMC5-B343 |
|
46 |
12ST2-5 |
Thalassobacillus pellis
|
18OM |
98.13 |
- |
- |
- |
- |
NMC5-B344 |
|
47 |
12ST2-6 |
Halobacillus mangrovi
|
MS10 |
98.31 |
+++ |
- |
- |
- |
NMC5-B345 |
|
48 |
12ST2-7 |
Halobacillus dabanensis
|
D-8 |
97.58 |
+++ |
- |
- |
- |
NMC5-B346 |
|
49 |
12ST2-8 |
Bacillus vietnamensis
|
15-1 |
97.92 |
+++ |
- |
- |
- |
NMC5-B347 |
|
50 |
12ST2-9 |
Halobacillus dabanensis
|
D-8 |
98.42 |
+++ |
- |
- |
- |
NMC5-B348 |
|
51 |
12ST2-10 |
Halobacillus trueperi
|
DSM 10404 |
98.25 |
+++ |
- |
- |
- |
NMC5-B349 |
|
52 |
12ST2-13 |
Bacillus hwajinpoensis
|
SW-72 |
99.36 |
+++ |
- |
- |
- |
NMC5-B350 |
|
53 |
12ST2-14 |
Pontibacillus chungwhensis
|
BH030062 |
98.96 |
+++ |
- |
- |
- |
NMC5-B351 |
|
54 |
12ST2-15 |
Halobacillus mangrovi
|
MS10 |
98.40 |
+++ |
- |
- |
- |
NMC5-B352 |
|
55 |
12ST2-16 |
Halobacillus dabanensis
|
D-8 |
97.38 |
+++ |
- |
- |
- |
NMC5-B353 |
|
56 |
12ST2-18 |
Halobacillus halophilus
|
DSM 2266 |
98.24 |
+++ |
- |
- |
- |
NMC5-B354 |
|
57 |
12ST2-19 |
Thalassobacillus devorans
|
G-19.1 |
97.30 |
+++ |
- |
- |
- |
NMC5-B355 |
|
58 |
12ST2-20 |
Halobacillus halophilus
|
DSM 2266 |
98.06 |
+++ |
- |
- |
- |
NMC5-B356 |
|
59 |
13ST1-1 |
Halobacillus mangrovi
|
MS10 |
98.58 |
+++ |
- |
- |
- |
NMC5-B357 |
|
60 |
13ST1-2 |
Halobacillus halophilus
|
DSM 2266 |
99.36 |
++ |
- |
++ |
- |
NMC5-B358 |
|
61 |
13ST1-3-1 |
Halobacillus andaensis
|
NEAU-ST10-40 |
97.55 |
++ |
- |
- |
- |
NMC5-B359 |
|
62 |
13ST1-4 |
Halobacillus dabanensis
|
D-8 |
97.94 |
+++ |
++ |
- |
- |
NMC5-B360 |
|
63 |
13ST1-5 |
Bacillus vietnamensis
|
15-1 |
98.72 |
+++ |
- |
+ |
- |
NMC5-B361 |
|
64 |
13ST1-7 |
Halobacillus mangrovi
|
MS10 |
98.20 |
+++ |
- |
++ |
- |
NMC5-B362 |
|
65 |
13ST1-8 |
Halobacillus halophilus
|
DSM 2266 |
98.45 |
+++ |
- |
- |
- |
NMC5-B363 |
|
66 |
13ST1-9 |
Halobacillus halophilus
|
DSM 2266 |
96.84 |
+++ |
- |
- |
- |
NMC5-B364 |
|
67 |
13ST1-10 |
Bacillus vietnamensis
|
15-1 |
98.21 |
+++ |
+ |
+ |
- |
NMC5-B365 |
|
68 |
13ST1-11 |
Pontibacillus chungwhensis
|
BH030062 |
98.86 |
+++ |
- |
++ |
- |
NMC5-B366 |
|
69 |
13ST1-12 |
Halobacillus naozhouensis
|
JSM 071068 |
98.53 |
+++ |
- |
++ |
- |
NMC5-B367 |
|
70 |
13ST1-13 |
Thalassobacillus cyri
|
CCM7597 |
99.54 |
+++ |
- |
- |
- |
NMC5-B368 |
|
71 |
13ST1-14 |
Halobacillus halophilus
|
DSM 2266 |
98.91 |
+++ |
- |
++ |
- |
NMC5-B369 |
|
72 |
13ST1-15 |
Halobacillus mangrovi
|
MS10 |
98.79 |
+++ |
- |
- |
- |
NMC5-B370 |
|
73 |
13ST1-16 |
Halobacillus trueperi
|
DSM 10404 |
97.87 |
+++ |
- |
- |
- |
NMC5-B371 |
|
74 |
13ST1-17 |
Halobacillus mangrovi
|
MS10 |
98.48 |
+++ |
- |
- |
- |
NMC5-B372 |
|
75 |
13ST1-18 |
Bacillus vietnamensis
|
15-1 |
99.02 |
+++ |
- |
- |
- |
NMC5-B373 |
|
76 |
13ST1-19 |
Halobacillus dabanensis
|
D-8 |
99.45 |
+++ |
- |
- |
- |
NMC5-B374 |
|
77 |
13ST1-20 |
Halobacillus mangrovi
|
MS10 |
98.68 |
+++ |
- |
- |
- |
NMC5-B375 |
|
78 |
13ST2-1 |
Halobacillus trueperi
|
DSM 10404 |
98.25 |
+++ |
- |
- |
- |
NMC5-B376 |
|
79 |
13ST2-2 |
Halobacillus halophilus
|
DSM 2266 |
99.36 |
+++ |
- |
- |
- |
NMC5-B377 |
|
80 |
13ST2-3 |
Halobacillus trueperi
|
MSS-402 |
98.15 |
+++ |
- |
- |
- |
NMC5-B378 |
|
81 |
13ST2-4 |
Halobacillus dabanensis
|
D-8 |
97.94 |
+++ |
- |
- |
- |
NMC5-B379 |
|
82 |
13ST2-5 |
Bacillus aquimaris
|
15-1 |
98.72 |
+++ |
- |
- |
- |
NMC5-B380 |
|
83 |
13ST2-6 |
Halobacillus mangrovi
|
MS10 |
97.65 |
+++ |
- |
- |
- |
NMC5-B381 |
|
84 |
13ST2-7 |
Pontibacillus salipaludis
|
9DM |
98.52 |
+++ |
- |
- |
- |
NMC5-B382 |
|
85 |
13ST2-8 |
Bacillus aquimaris
|
15-1 |
98.42 |
+++ |
++ |
+++ |
- |
NMC5-B383 |
|
86 |
13ST2-9 |
Halobacillus mangrovi
|
MS10 |
97.94 |
+++ |
- |
- |
- |
NMC5-B384 |
|
87 |
13ST2-11 |
Halobacillus trueperi
|
DSM 10404 |
98.43 |
+++ |
- |
- |
- |
NMC5-B385 |
|
88 |
13ST2-12-1 |
Pontibacillus salipaludis
|
BH030062 |
98.58 |
+++ |
+++ |
- |
- |
NMC5-B386 |
|
89 |
13ST2-13 |
Halobacillus campisalis
|
MS10 |
99.44 |
+++ |
- |
- |
- |
NMC5-B387 |
|
90 |
13ST2-14 |
Halobacillus mangrovi
|
MS10 |
97.52 |
+++ |
- |
- |
- |
NMC5-B388 |
|
91 |
13ST2-15 |
Halobacillus hunanensis
|
JSM 071077 |
98.99 |
++ |
- |
- |
- |
NMC5-B389 |
|
92 |
13ST2-16 |
Halobacillus trueperi
|
MS10 |
97.44 |
+++ |
- |
+ |
- |
NMC5-B390 |
|
93 |
13ST2-18 |
Halobacillus campisalis
|
MS10 |
98.50 |
+++ |
- |
++ |
- |
NMC5-B391 |
|
94 |
13ST2-19 |
Halobacillus trueperi
|
DSM 10404 |
98.52 |
+++ |
- |
- |
- |
NMC5-B392 |
|
95 |
13ST2-20 |
Halobacillus halophilus
|
DSM 2266 |
99.36 |
+++ |
- |
- |
- |
NMC5-B393 |
|
96 |
KTB011 |
Halobacillus trueperi
|
DSM 10404 |
100.00 |
+++ |
++ |
+++ |
- |
NMC4-B482 |
|
97 |
KTB022 |
Halobacillus alkaliphilus
|
FP5 |
100.00 |
+++ |
- |
++ |
- |
NMC4-B483 |
|
98 |
KTB023 |
Halobacillus halophilus
|
DSM 2266 |
99.87 |
+++ |
- |
+++ |
- |
NMC4-B484 |
|
99 |
KTB081 |
Bacillus hwajinpoensis
|
SW-72 |
100.00 |
++ |
++ |
+++ |
- |
NMC4-B485 |
|
100 |
KTB272 |
Pontibacillus chungwhensis
|
BH030062 |
100.00 |
+++ |
+++ |
+ |
- |
NMC4-B486 |
|
101 |
ST3rd#203 |
Bacillus berkeleyi
|
KMM 6244 |
95.89 |
- |
- |
- |
- |
NMC6-B408 |
|
102 |
ST2nd#107 |
Kangiella koreensis
|
DSM 16069 |
96.62 |
- |
+++ |
+++ |
+ |
NMC6-B427 |
|
103 |
ST2nd#108 |
Kangiella koreensis
|
DSM 16069 |
96.43 |
- |
+++ |
+++ |
+ |
NMC6-B428 |
|
104 |
ST3rd#201 |
Kangiella koreensis
|
DSM 16069 |
96.88 |
- |
+++ |
+ |
+ |
NMC6-B429 |
|
105 |
ST3rd#205 |
Kangiella koreensis
|
DSM 16069 |
95.98 |
- |
+++ |
+ |
w |
NMC6-B430 |
|
106 |
ST3rd#239 |
Kangiella koreensis
|
DSM 16069 |
95.47 |
- |
+++ |
+ |
+ |
NMC6-B431 |
|
107 |
ST2nd#109 |
Kangiella koreensis
|
DSM 16069 |
97.87 |
- |
+++ |
+++ |
+ |
NMC6-B432 |
|
108 |
ST2nd#110 |
Kangiella koreensis
|
DSM 16069 |
96.85 |
- |
+++ |
+++ |
++ |
NMC6-B433 |
|
109 |
ST3rd#210 |
Kangiella koreensis
|
DSM 16069 |
96.42 |
- |
+++ |
+ |
+ |
NMC6-B434 |
|
110 |
ST3rd#212 |
Kangiella koreensis
|
DSM 16069 |
96.11 |
- |
+++ |
+ |
++ |
NMC6-B435 |
|
111 |
ST3rd#222 |
Marinobacter algicola
|
DG893 |
96.20 |
+++ |
- |
- |
- |
NMC6-B440 |
|
112 |
ST3rd#229 |
Halomonas ventosae
|
Al12 |
97.97 |
+++ |
- |
- |
++ |
NMC6-B450 |
|
113 |
13SJ1-1 |
Bacillus vietnamensis
|
15-1 |
99.11 |
+++ |
- |
- |
- |
NMC5-B316 |
|
114 |
13SJ2-1 |
Halobacillus mangrovi
|
MS10 |
97.73 |
+++ |
- |
- |
- |
NMC5-B317 |
|
115 |
14SJ1-1 |
Halobacillus trueperi
|
DSM 10404 |
98.43 |
+++ |
- |
- |
- |
NMC5-B318 |
|
116 |
14SJ1-2 |
Pontibacillus chungwhensis
|
BH030062 |
98.66 |
+++ |
- |
- |
- |
NMC5-B319 |
|
117 |
ST2nd#101 |
Kangiella koreensis
|
DSM 16069 |
100.00 |
- |
+++ |
+ |
- |
NMC4-B514 |
|
118 |
ST3rd#204 |
Gramella aestuariivivens
|
BG-MY13 |
96.05 |
- |
+++ |
+ |
- |
NMC4-B515 |
|
119 |
ST3rd#225 |
Marinobacter segnicrescens
|
SS011B1-4 |
95.99 |
- |
+++ |
- |
- |
NMC4-B516 |
|
120 |
ST3rd#228 |
Halomonas saccharevitans
|
AJ275 |
97.07 |
- |
- |
- |
- |
NMC4-B517 |
|
121 |
ST3rd#231 |
Marinobacterium aestuariivivens
|
DB-1 |
100.00 |
- |
+++ |
+ |
- |
NMC4-B518 |
|
122 |
ST3rd#238 |
Halobacillus mangrovi
|
MS10 |
100.00 |
+++ |
+ |
+++ |
- |
NMC4-B519 |
|
123 |
ST3rd#241-1 Salinimonas chungwhensis |
YelD216 |
96.86 |
+ |
+++ |
- |
- |
NMC4-B520 |
|
124 |
ST3rd#242 |
Halomonas ventosae
|
Al12 |
98.41 |
- |
- |
- |
- |
NMC4-B521 |
|
125 |
STCP#202 |
Marinobacter algicola
|
DG893 |
96.11 |
- |
- |
- |
- |
NMC4-B522 |
|
126 |
STCP#209 |
Bacillus subtilis subsp. Inaquosorum
|
KCTC 13429 |
99.93 |
+++ |
- |
+++ |
- |
NMC4-B523 |
|
127 |
GL1-1 |
Halobacillus mangrovi
|
MS10 |
98.87 |
+++ |
- |
- |
- |
NMC5-B394 |
|
128 |
GL1-2 |
Pontibacillus chungwhensis
|
DSM 10404 |
98.43 |
+++ |
+ |
- |
- |
NMC5-B395 |
|
129 |
GT1-1 |
Halobacillus trueperi
|
DSM 10404 |
98.53 |
+++ |
- |
++ |
- |
NMC5-B396 |
|
130 |
GT1-2 |
Halobacillus locisalis
|
MSS-155 |
98.43 |
+++ |
- |
- |
- |
NMC5-B397 |
|
131 |
GT1-3 |
Pontibacillus chungwhensis
|
BH030062 |
98.20 |
- |
++ |
- |
- |
NMC5-B398 |
|
132 |
GT1-4 |
Halobacillus locisalis
|
MSS-155 |
99.27 |
- |
- |
- |
- |
NMC5-B399 |
|
133 |
GT1-5 |
Pontibacillus yanchengensis
|
Y32 |
99.54 |
- |
- |
- |
- |
NMC5-B400 |
|
134 |
GT1-6 |
Halobacillus trueperi
|
DSM 10404 |
98.63 |
+++ |
- |
- |
- |
NMC5-B401 |
|
135 |
GT1-7-1 |
Aquibacillus koreensis
|
BH30097 |
97.81 |
- |
- |
- |
- |
NMC5-B402 |
|
136 |
GT1-8 |
Halobacillus locisalis
|
MSS-155 |
98.81 |
- |
- |
- |
- |
NMC5-B403 |
|
137 |
GT1-9 |
Halobacillus mangrovi
|
MS10 |
99.07 |
+++ |
- |
- |
- |
NMC5-B404 |
|
138 |
GT1-10 |
Thalassobacillus hwangdonensis
|
AD-1 |
98.64 |
- |
- |
- |
- |
NMC5-B405 |
|
139 |
GT1-11 |
Thalassobacillus hwangdonensis
|
AD-1 |
98.64 |
- |
- |
- |
- |
NMC5-B406 |
|
140 |
GT1-12 |
Pontibacillus yanchengensis
|
Y32 |
99.54 |
- |
- |
- |
- |
NMC5-B407 |
|
141 |
GT1-13 |
Aquibacillus koreensis
|
BH30097 |
97.81 |
- |
- |
- |
- |
NMC5-B408 |
|
142 |
GT2-1 |
Virgibacillus chiguensis
|
NTU-101 |
97.83 |
- |
- |
- |
- |
NMC5-B409 |
|
143 |
GT2-2 |
Halobacillus dabanensis
|
D-8 |
98.13 |
+++ |
++ |
- |
- |
NMC5-B410 |
|
144 |
GT2-3 |
Pontibacillus chungwhensis
|
BH030062 |
96.60 |
- |
- |
- |
- |
NMC5-B411 |
|
145 |
GT2-4 |
Halobacillus profundi
|
IS-Hb4 |
97.69 |
+++ |
- |
- |
- |
NMC5-B412 |
|
146 |
GT2-5 |
Bacillus hwajinpoensis
|
SW-72 |
98.63 |
+++ |
- |
- |
- |
NMC5-B413 |
|
147 |
GT2-6 |
Filobacillus milosensis
|
DSM 13259 |
96.98 |
- |
- |
- |
- |
NMC5-B414 |
|
148 |
GT2-7 |
Halobacillus halophilus
|
DSM 2266 |
96.93 |
- |
- |
- |
- |
NMC5-B415 |
|
149 |
GT2-9 |
Virgibacillus halodenitrificans
|
SK37 |
96.60 |
- |
- |
+ |
- |
NMC5-B416 |
|
150 |
GT2-10 |
Halobacillus locisalis
|
MSS-155 |
97.43 |
+++ |
- |
- |
- |
NMC5-B417 |
|
151 |
GS-RS 1A |
Micrococcus luteus
|
NCTC 2665 |
100.00 |
- |
++ |
- |
- |
NMC4-B86 |
|
152 |
GS-RS 5A |
Bacillus niabensi
|
4T19 |
100.00 |
+++ |
- |
- |
- |
NMC4-B87 |
|
153 |
GS-RS 1D |
Pseudomonas oryzihabitans
|
NBRC 102199 |
100.00 |
- |
++ |
+ |
- |
NMC4-B88 |
|
154 |
GS-RS 2D |
Desemzia incerta
|
DSM 2058 |
99.86 |
- |
- |
- |
- |
NMC4-B89 |
|
155 |
GS-RS 4D |
Acinetobacter pittii
|
CIP 70.29 |
100.00 |
- |
++ |
- |
- |
NMC4-B90 |
|
156 |
GS-RS 5D |
Acinetobacter pittii
|
CIP 70.29 |
100.00 |
- |
++ |
- |
- |
NMC4-B91 |
|
157 |
GS-OS 1A |
Bacillus haikouensis
|
C-89 |
100.00 |
+++ |
- |
- |
- |
NMC4-B92 |
|
158 |
GS-OS 2A |
Bacillus boroniphilus
|
JCM 21738 |
100.00 |
+++ |
- |
- |
- |
NMC4-B93 |
|
159 |
GS-OS 6A |
Staphylococcus caprae
|
ATCC 35538 |
99.80 |
+++ |
+++ |
- |
- |
NMC4-B94 |
|
160 |
GS-OS 7A |
Staphylococcus cohnii subsp. urealyticus
|
ATCC 49330 |
100.00 |
- |
++ |
- |
- |
NMC4-B95 |
|
161 |
GS-OS 4B |
Staphylococcus argenteus
|
MSHR1132 |
99.86 |
- |
+++ |
++ |
- |
NMC4-B96 |
|
162 |
GS-OS 6B |
Staphylococcus epidermidis
|
ATCC 14990 |
99.93 |
- |
- |
- |
- |
NMC4-B97 |
|
163 |
ER1-1 |
Virgibacillus jeotgali
|
NS3012 |
96.64 |
- |
+ |
++ |
- |
NMC5-B432 |
|
164 |
ER1-2 |
Virgibacillus phasianinus
|
LM2416 |
97.62 |
- |
+ |
++ |
- |
NMC5-B433 |
|
165 |
ER1-3 |
Oceanobacillus picturae
|
LMG 19492 |
97.24 |
+++ |
- |
+ |
- |
NMC5-B434 |
|
166 |
ER1-4 |
Oceanobacillus picturae
|
LMG 19492 |
98.90 |
- |
- |
+++ |
- |
NMC5-B435 |
|
167 |
ER1-5 |
Halobacillus naozhouensis
|
JSM 071068 |
98.27 |
- |
- |
+ |
- |
NMC5-B436 |
|
168 |
ER1-6 |
Halobacillus hunanensis
|
JSM 071077 |
98.54 |
- |
- |
- |
- |
NMC5-B437 |
|
169 |
ER1-7 |
Halobacillus hunanensis
|
JSM 071077 |
97.67 |
- |
- |
- |
- |
NMC5-B438 |
|
170 |
ER1-8 |
Halobacillus andaensis
|
NEAU-ST10-40 |
95.49 |
- |
- |
- |
- |
NMC5-B439 |
|
171 |
ER1-9 |
Halobacillus hunanensis
|
JSM 071077 |
98.91 |
+++ |
- |
- |
- |
NMC5-B440 |
|
172 |
ER1-10 |
Halobacillus hunanensis
|
JSM 071077 |
99.45 |
+++ |
- |
- |
- |
NMC5-B441 |
|
173 |
ER1-11 |
Halobacillus naozhouensis
|
JSM 071068 |
98.27 |
- |
- |
+ |
- |
NMC5-B442 |
|
174 |
ER2-1 |
Halobacillus locisalis
|
MSS-155 |
96.98 |
- |
- |
- |
- |
NMC5-B443 |
|
175 |
ER2-3 |
Oceanobacillus picturae
|
LMG 19492 |
97.13 |
+++ |
++ |
+ |
- |
NMC5-B444 |
|
176 |
ER2-4 |
Oceanobacillus iheyensis
|
HTE831 |
95.39 |
- |
- |
- |
- |
NMC5-B445 |
|
177 |
ER2-5 |
Oceanobacillus picturae
|
LMG 19492 |
98.27 |
- |
- |
- |
- |
NMC5-B446 |
|
178 |
ER2-6 |
Oceanobacillus picturae
|
LMG 19492 |
97.30 |
+++ |
++ |
- |
- |
NMC5-B447 |
|
179 |
ER2-7 |
Oceanobacillus picturae
|
LMG 19492 |
97.78 |
- |
+ |
++ |
- |
NMC5-B448 |
|
180 |
ER2-8 |
Halobacillus locisalis
|
MSS-155 |
99.20 |
- |
++ |
+ |
- |
NMC5-B449 |
|
181 |
ER2-9 |
Oceanobacillus picturae
|
LMG 19492 |
97.30 |
- |
- |
- |
- |
NMC5-B450 |
|
182 |
ER2-10 |
Oceanobacillus picturae
|
LMG 19492 |
97.30 |
- |
+ |
- |
- |
NMC5-B451 |
|
183 |
R450201 |
Bacillus paralicheniformis
|
KJ-16 |
97.92 |
++ |
+ |
+++ |
- |
NMC4-B383 |
|
184 |
R450202 |
Oceanobacillus limi
|
H9B |
97.94 |
- |
- |
+++ |
- |
NMC4-B384 |
|
185 |
R450203 |
Bacillus paralicheniformis
|
KJ-16 |
97.66 |
++ |
+ |
+++ |
- |
NMC4-B385 |
|
186 |
R450204 |
Oceanobacillus limi
|
H9B |
99.22 |
- |
+ |
+++ |
- |
NMC4-B386 |
|
187 |
R450205 |
Bacillus sonorensis
|
NSP9.1 |
97.34 |
++ |
+ |
+++ |
- |
NMC4-B387 |
|
188 |
R450206 |
Bacillus paralicheniformis
|
KJ-16 |
97.72 |
++ |
+ |
+++ |
- |
NMC4-B388 |
|
189 |
R450301 |
Sediminibacillus halophilus
|
EN8d |
97.55 |
+++ |
+ |
+++ |
- |
NMC4-B389 |
|
190 |
R450302 |
Sediminibacillus halophilus
|
EN8d |
98.06 |
- |
- |
+++ |
- |
NMC4-B390 |
|
191 |
R450303 |
Sediminibacillus halophilus
|
EN8d |
98.87 |
- |
- |
+++ |
- |
NMC4-B391 |
|
192 |
R450304-1 |
Sediminibacillus halophilus
|
EN8d |
96.61 |
++ |
- |
- |
- |
NMC4-B392 |
|
193 |
R450305 |
Bacillus sonorensis
|
NSP9.1 |
97.42 |
++ |
- |
- |
- |
NMC4-B393 |
|
194 |
R450306 |
Bacillus sonorensis
|
NSP9.1 |
97.95 |
++ |
- |
- |
- |
NMC4-B394 |
|
195 |
R450307 |
Bacillus sonorensis
|
NSP9.1 |
98.00 |
++ |
- |
- |
- |
NMC4-B395 |
|
196 |
R450308 |
Bacillus sonorensis
|
NSP9.1 |
97.16 |
+++ |
- |
+++ |
- |
NMC4-B396 |
|
197 |
R450401 |
Bacillus paralicheniformis
|
KJ-16 |
97.59 |
+++ |
++ |
+++ |
- |
NMC4-B397 |
|
198 |
R450402 |
Bacillus paralicheniformis
|
KJ-16 |
97.37 |
+++ |
++ |
+++ |
- |
NMC4-B398 |
|
199 |
R450403 |
Bacillus sonorensis
|
NSP9.1 |
96.38 |
+++ |
- |
+++ |
- |
NMC4-B399 |
|
200 |
R450405 |
Bacillus paralicheniformis
|
KJ-16 |
99.02 |
++ |
- |
+++ |
- |
NMC4-B400 |
|
201 |
R450408 |
Bacillus sonorensis
|
NSP9.1 |
97.88 |
- |
- |
- |
- |
NMC4-B401 |
|
202 |
R450409-1 |
Gracilibacillus lacisalsi
|
BH312 |
95.07 |
++ |
- |
- |
- |
NMC4-B402 |
|
203 |
R450501 |
Gracilibacillus lacisalsi
|
BH312 |
98.01 |
++ |
- |
- |
- |
NMC4-B403 |
|
204 |
R450502 |
Alteribacillus iranensis
|
DSM 23995 |
99.06 |
- |
- |
- |
- |
NMC4-B404 |
|
205 |
R450503 |
Oceanobacillus limi
|
H9B |
98.03 |
- |
- |
+++ |
- |
NMC4-B405 |
|
206 |
R450504 |
Alteribacillus iranensis
|
DSM 23995 |
99.08 |
- |
- |
- |
- |
NMC4-B406 |
|
207 |
R450505 |
Oceanobacillus limi
|
H9B |
97.82 |
- |
- |
+++ |
- |
NMC4-B407 |
|
208 |
R450506-1 |
Oceanobacillus limi
|
H9B |
97.33 |
- |
- |
+++ |
- |
NMC4-B408 |
|
209 |
R450507-1 |
Alteribacillus iranensis
|
DSM 23995 |
96.74 |
- |
- |
- |
- |
NMC4-B409 |
|
210 |
ERB012 |
Halobacillus sediminis
|
NGS-2 |
100.00 |
- |
- |
- |
- |
NMC4-B456 |
|
211 |
ERB032 |
Oceanobacillus kimchii
|
X50 |
100.00 |
- |
- |
+ |
- |
NMC4-B457 |
|
212 |
ERB054 |
Marinococcus luteus
|
DSM 23126 |
99.80 |
- |
- |
- |
- |
NMC4-B458 |
|
213 |
ERB061 |
Marinococcus luteus
|
DSM 23126 |
99.80 |
- |
- |
- |
- |
NMC4-B459 |
|
214 |
ERB081 |
Chromohalobacter canadensis
|
ATCC 43984 |
99.93 |
+++ |
+ |
++ |
- |
NMC4-B460 |
|
215 |
ERB082 |
Halobacillus sediminis
|
NGS-2 |
100.00 |
+ |
- |
+++ |
- |
NMC4-B461 |
|
216 |
ERB112 |
Halobacillus dabanensis
|
D-8 |
100.00 |
+++ |
- |
+++ |
- |
NMC4-B462 |
|
|
|
|
|
|
|
|
|
|
|
|
217 |
EL1-1 |
Oceanobacillus picturae
|
R-5321 |
95.57 |
+++ |
- |
+ |
- |
NMC5-B418 |
|
218 |
EL1-2 |
Oceanobacillus picturae
|
R-5321 |
96.83 |
- |
- |
+ |
- |
NMC5-B419 |
|
219 |
EL1-4 |
Oceanobacillus picturae
|
R-5321 |
96.79 |
+++ |
+++ |
+ |
- |
NMC5-B420 |
|
220 |
EL1-5 |
Virgibacillus senegalensis
|
Marseille-P3518 |
96.47 |
- |
+++ |
- |
- |
NMC5-B421 |
|
221 |
EL1-6 |
Oceanobacillus picturae
|
LMG 19492 |
97.60 |
+++ |
+++ |
+ |
- |
NMC5-B422 |
|
222 |
EL1-7 |
Oceanobacillus picturae
|
LMG 19492 |
96.02 |
+++ |
+++ |
- |
- |
NMC5-B423 |
|
223 |
EL1-8 |
Virgibacillus senegalensis
|
Marseille-P3518 |
96.47 |
- |
+++ |
- |
- |
NMC5-B424 |
|
224 |
EL2-1 |
Oceanobacillus picturae
|
LMG 19492 |
97.03 |
+++ |
- |
+ |
- |
NMC5-B425 |
|
225 |
EL2-2 |
Oceanobacillus manasiensis
|
YD3-56 |
95.80 |
- |
- |
- |
- |
NMC5-B426 |
|
226 |
EL2-4 |
Tenuibacillus halotolerans
|
YIM 94025 |
98.71 |
- |
- |
- |
- |
NMC5-B427 |
|
227 |
EL2-5 |
Tenuibacillus halotolerans
|
YIM 94025 |
95.88 |
+++ |
- |
- |
- |
NMC5-B428 |
|
228 |
EL2-7 |
Oceanobacillus picturae
|
LMG 19492 |
98.81 |
+++ |
- |
- |
- |
NMC5-B429 |
|
229 |
EL2-9 |
Oceanobacillus picturae
|
LMG 19492 |
97.40 |
+++ |
- |
- |
- |
NMC5-B430 |
|
230 |
EL2-10 |
Oceanobacillus picturae
|
LMG 19492 |
96.93 |
+++ |
- |
- |
- |
NMC5-B431 |
Download Excel Table
분해 효소 및 옥신 생산능 분석
해양 미생물들은 유기물이 부족한 주변 환경에서 생육하 기 위하여 영양분을 분해하여 에너지로 활용하기 위한 다 양한 분해 효소 분비등과 같은 상호작용이 필요할 것으로 예상되어 분리된 호염성 미생물 균주들의 신규 분해 효소 탐색 자원 및 미생물비료로써의 산업적 응용가능성을 확인 하기 위하여 세포외 분해 효소 생산능 및 옥신 생산능 분 석을 실시하였다.
Table 3에 나타낸 것처럼 분리된 230 균주에서 53 균주 를 제외한 모든 균주에서 한 가지 이상의 분해 효소 활성 이 존재하는 것을 확인하였으며, 두 가지 이상의 효소 활 성을 가지는 균주 중 특히 세 가지 효소에 대하여 활성을 모두 가지고 있는 것으로 확인된 17균주(과제기탁번호: NMC5-B365, NMC5-B383, NMC4-B482, NMC4-B485, NMC4-B486, NMC4-B519, NMC5-B444, NMC4-B383, NMC4-B385, NMC4-B387, NMC4-B388, NMC4-B389, NMC4-B397, NMC4-B398, NMC4-B460, NMC5-B420, NMC5-B422)는 Bacillaceae 과 16균주, Halomonadaceae 과 1균주인 것으로 확인하였다. 이 중 11균주는 국외 염전 시료에서 분리된 균주였으며 나머지 6균주가 국내 염전 시 료에서 분리한 균주였다. 또한 옥신 생산능을 확인한 결과, 24 균주에서 생산능이 확인되었으며 신안 토판염에서 분리 된 균주 중 14균주는 모두 Halobacillus sp. 균주였으며 나 머지 10균주는 모두 신안 장판염에서 Kangiella sp. 9균주 와 Halomonas sp. 1균주가 분리되었으며 국외 염전 시료 에서는 옥신 생산능을 가진 균주는 분리되지 않았다. 특히 신안 장판염에서 분리한 균주들의 생산능이 토판염에서 분 리한 균주들의 생성능 보다 높은 것을 알 수 있었으며 이 는 식물생장을 촉진시킬 수 있는 미생물비료 연구에 활용 할 수 있는 미생물 탐색 소재로써 국내 염전 중 장판염으 로 생산하는 천일염이 중요한 원천소재로써의 활용가치가 높을 것으로 예상되었다. 본 연구에서 수행한 결과들은 국 내 생물 자원의 다양성 확보 차원에서 큰 의미를 찾을 수 있을 것이며 국내외 염전 시료에서 분해 효소 탐색을 위한 신균주의 활용 가능성과 산업용 효소 관련 생물공학 및 미 생물비료 연구의 기본 생물소재로 활용이 가능할 것으로 예상된다. 또한 본 연구를 통하여 분리한 모든 균주들은 한국생명공학연구원 미생물가치제고사업단에 기탁하였다.
요 약
본 연구는 국내외 염전 시료로부터 분리한 호염성 미생 물들의 다양성 및 특성에 관하여 조사하였다. 호염성 미생 물의 순수 분리를 위하여 marine agar 배지를 사용하였으 며 37°C에서 호기적으로 배양하였다. 순수 분리 후, 230 균주를 분리하였으며 16S rRNA 염기서열 분석 결과를 바 탕으로 계통학적 분석을 실시한 결과, 4문, 12과, 27속, 64 종으로 구성되어 있는 것을 확인하였다. 특히, Firmicutes 문은 89.6%의 분포를 나타내었으며 3과, 17속, 52종으로, Bacillaceae, Staphylococcaceae와 Carnobacteriaceae로 분포 하는 것을 확인하였다. 그리고 분리한 균주들이 amylase, lipase, protease와 같은 산업적으로 유용한 효소를 생산하 는지 확인하기 위하여 효소 활성 평가를 실시하였으며, 177균주가 최소 한 종류 이상의 효소 활성을 가지고 있는 것을 확인하였으며 세 가지 효소에 대하여 모든 활성을 가 지고 있는 균주도 17균주가 확인되었으며 이는 본 연구를 통하여 분리한 미생물들의 산업적 활용 가능성을 나타내었 다. 그러므로 이번 연구는 국내 유전자원 확보 및 염전 시 료의 호염성 미생물의 다양성과 특성에 관한 과학적 지식 확장에 도움이 될 것으로 생각된다.
감사의 글
이 논문은 2019년도 한국연구재단 이공분야기초연구사업 (NRF-2019R1F1A1060737)과 바이오·의료기술개발사업(NRF- 2013M3A9A5076603)의 지원을 받아 수행된 연구임.
References
Baati H, Guermazi S, Gharsallah N, Sghir A, Ammar E.2010. Microbial community of salt crystals processed from Mediterranean seawater based on 16S rRNA analysis. Can. J. Microbiol. 56: 44-51.


Ban OH, Han SS, Lee YN.2003. Identification of a potent protease-producing bacterial isolate, Bacillus amyloliquefaciens CMB01. Ann. Microbiol. 53: 95-103.
Beak HY.1987. A study on nutrition of salt. Kor. J. Soc. Food Cookery Sci. 3: 92-106.
Birbir M, Calli B, Mertoglu B, Bardavid RE, Oren A, Ogmen MN, Ogan A.2007. Extremely halophilic Archaea from Tuz Lake, Turkey, and the adjacent Kaldirim and Kayacik salterns. World J. Microbiol. Biotech. 23: 309-316.

Lee KD, Park JW, Choi CR, Song HW, Yun SK, Yang HC, Ham KS.2007. Salinity and heavy metal contents of solar salts produced in Jeollanamdo province of Korea. Korean J. Food Sci. Nutr. 36: 753-758.

Lu J, Nogi Y, Takami H.2001. Oceanobacillus iheyensis gen. nov., sp. nov., a deep-sea extremely halotolerant and alkaliphilic species isolated from a depth of 1050 m on the Iheya Ridge. FEMS Microbiol Lett. 205: 291-297.


Mayuri S, Yogesh G, Shalini A, Vikas K, Anil P, Ashwani K.2019. A review on microbial alkaline protease: An essential tool for various industrial approaches. Industrial Biotechnology. 15: 69-78.

Morel V.2005. Cruelest Place on Earth, The Africa's Danakil Desert. National Geographic, 208(4): 32–53.
Na JM, Kang MS, Kim JH, Jin YX, Je OH, Kim JB, Cho YS, Kim JH, Kim SY.2011. Distribution and identification of halophilic bacteria in solar salts produced during entire manufacturing process. Korean J. Microbiol. Biotechnol. 39: 133-139.
Park JW, Kim SJ, Kim SH, Kim BH, Kang SG, Nam SH, Jung ST.2000. Determination of mineral and heavy metal contents of various salts. Korean J. Food Sci. Technol. 32: 1442-1445.
Pesenti PT, Sikaroodi M, Gillevet PM, Sanchez-Porro C, Ventosa A, Litchfield CD.2008. Halorubrum californiense sp. nov., an extreme archaeal halophile isolated from a crystallizer pond at a solar salt plant in California, USA. Int. J. Syst. Evol. Microbiol. 58: 2710-2715.


VijayAnand S, Hemapriya J, Selvin J, Kiran S, Hemapriya J.2010. Production and optimization of haloalkaliphilic protease by an extremophile Halobacterium sp. JS1, isolated from thalassohaline environment. Global J. Biotech. Biochem. 5: 44-49.