서 론
가짜식품(economically motivated adulteration, EMA)이란 경제적 이득을 취하기 위하여 원재료를 속이고, 의도적으 로 저렴한 원료를 사용하는 등 부당한 방법으로 제조된 식 품을 의미한다(Kim et al., 2014). 가짜 식품의 한 예로 쌀 과자에 밀 등 기타재료를 섞어놓고도 쌀과자로 속여 부당 이익을 얻거나 하는 등의 부정식품 판매 업체 58곳을 적 발하였다는 언론보도사례 등이 발생 되고 있다(Http://news. jtbc.joins.com/article/article.aspx?news_id=NB11162064). 외국 의 경우 미국 식품의약품청, 영국 식품기준청 그리고 캐나 다 국경관리청과 같은 정부기관들에서 가짜 식품을 비롯한 불량식품의 관리를 강화하고 있고, 우리나라도 2010년부터 식품의약품안전처에서 다소비식품을 대상으로 유전자 분석 을 활용한 식품원료의 진위 판별법을 마련하고 있다(Hong et al., 2015). 혼합식품이나 가짜식품 등을 가공하거나 처 리하였을 경우 외관으로 원재료의 판별이 거의 불가능하기 때문에 원재료의 정확한 표시와 원재료를 정확하게 검증할 수 있는 과학적인 기술개발이 중요하다. 원재료 검증 방법 에는 원재료의 특정 성분 물질의 유무를 확인할 수 있는 이화학적 분석법(Schellenberg et al., 2010), 면역효소측정 법과 등전점 전기영동법으로 대표되는 단백질 분석법 그 리고 DNA분자수준에서의 염기서열 차이에 기초한 PCR 기법 등과 같은 유전자 분석법(Folmer et al., 1994;Fabric et al., 2005)이 있다. 이화학적 분석법은 방법이 복잡하고 고가의 분석기기를 필요로 하기 때문에 적용이 쉽지 않고, 단백질 분석법은 동종간 판별이 어려울 뿐만 아니라 열처 리 과정을 거친 가공품인 경우 항원의 변성으로 감수성이 떨어진다. 그러므로 최근에는 열에 비교적 안정한 유전자를 대상으로 유전자 분석법이 사용되고 있다. 그 중 PCR을 이용한 유전자 검출 방법은 특이성, 민감성이 매우 높고 식 품가공이나 제조 과정 중에서도 단백질에 비해 비교적 안 정한 물질이므로 외부적 환경 요인에 큰 영향을 받지 않기 때문에 분석결과의 오차를 줄일 수 있는 장점을 가지고 있 다(Hebert et al., 2003;Monaghan et al., 2003).
PCR을 이용한 식품원료의 유전자분석법에는 일반 프라 이머(universal primer)를 이용하여 증폭 후 염기서열을 결 정하고 미국 국립보건원에서 운영하는 유전자은행에 등록 되어있는 염기서열과 비교하여 종을 판별하는 방법과 종 특이프라이머(species-specific primer)를 이용하여 유전자 증폭 유무로 판별하는 방법(Kocher et al., 1989)이 있다. 특히, 가공식품은 제조 가공과정 중 고압살균, 화학작용 등 에 의해 DNA가 손상될 가능성이 있기 때문에 일반프라이 머를 이용한 염기서열 분석은 PCR증폭 산물의 크기가 700 bp 이상으로 가공식품에 적용하기가 어려워 PCR증폭 산물의 크기를 200 bp 내외가 되도록 설계하고 이를 이용 하여 증폭 여부로 종을 확인하는 방법(Park et al., 2012a;Park et al., 2012b;Park et al., 2012c;Park et al., 2012d;Park et al., 2012e;Park et al., 2013)이 많이 활용된다. 특 히, real time PCR분석법의 경우 기존의 PCR방법보다 빠 른 시간 내에 높은 감도로 증폭할수 있고, 프로브를 사용 하여 기존PCR의 위양성을 효과적으로 줄일 수 있어 신뢰 성을 높일수 있다는 평가를 받고 있다(Chung et al., 2018). 사용원료의 부정확한 표시나 제조 과정 중 원재료 외 다른 원료들의 비의도적 혼입을 통제하기 위해서는 제품에 함유 된 원료 물질을 모니터링 할 수 있는 효율적이고 정확한 분석방법의 개발이 필요하다. 따라서 본 연구에서는 쌀 등 의 가공식품에서 밀의 혼입 여부를 판별하기 위해서 다른 영역에 비해 빠른 진화 속도를 보여 종간, 종내 및 집단 내에서 변이조사 등에 광범위하게 이용되고 있는 internal transcribed spacer (ITS)영역(Kress et al., 2005;Kress et al., 2007;Kress et al., 2008;Lahaye et al., 2008)의 밀 구분마커로서 유용성과 real-time PCR 분석법의 활용가능 성을 알아보고자 하였다.
재료 및 방법
실험에 사용한 쌀과 밀 그리고 그 가공식품들은 2018년 6월부터 11월까지 경기도의 중소형유통매장 및 재래시장에 서 구입하였다. 곡류는 막자사발을 이용하여 균질하게 분 쇄하였고, 과자류는 분쇄 후 증류수로 3회 세척하였으며, 떡류는 고물이 있는 경우는 고물을 제거한 후 떡 부분만을 채취하여 분쇄한 것을 검체로 사용하였다. Real time PCR 은 Applied Biosystems 7500 Fast Real-Time PCR system (Life Technologies, Carlsbad, CA, USA)을 사용하였다.
DNA추출은DNeasy Plant mini kit (Qiagen GmbH, Hilden, Germany)를 사용하였고, 추출 방법은 쌀과밀 원재 료는 제조사에서 제공하는 방법에 따라 추출하였으며, 과 자와 떡 등의 가공식품은 추가로 α-amylase를 proteinase K와 동량, 동일시간 처리 하였다.
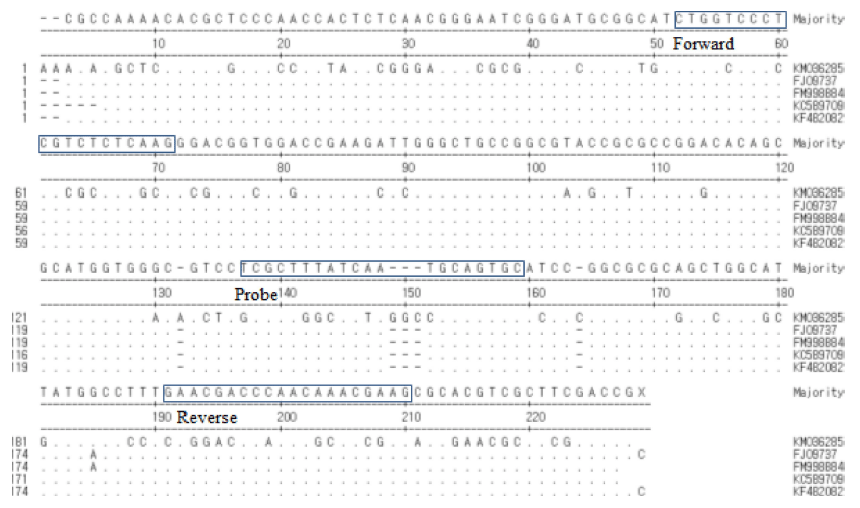
밀(Triticum aestivum, access number: FJ609737, FM998884, KC589709, KF482082)과 쌀(Oryza sative, access number: KM036285)의 ITS2 (Internal transcribed spacer) 염기서열은 National Center Biotechnology Information (NCBI, http:// www.ncbi.nlm.nih.gov)에 등록된 염기서열로부터 확보하였다. 확보된 쌀과 밀의 ITS2 염기서열은 Lasergene (Copyright 1997 by DNASTAR, Inc., Madison, WI, USA)의 MegAlign Programme을 통해서 비교 분석하였다(Fig. 1). 프라이머 및 프로브는 Primer3 plus (http://www.bioinformatics.nl/cbibin/ primer3plus/primer3plus.cgi)를 사용하여 설계하고 바이 오니아(Bioneer, Korea)에 의뢰하여 합성하였다.
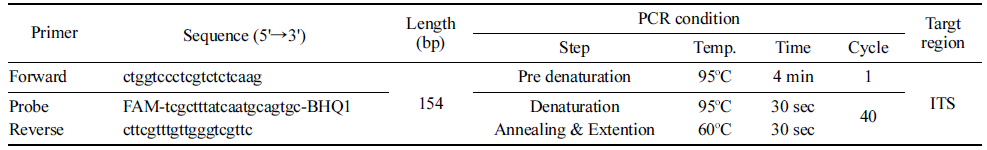
유전자 증폭을 위해 AccuPower PlusDualStar qPCR 2X Master mix (Bioneer, Korea)를 사용하였고 주형DNA 100 ng/μL, 10 pM의 정방향 및 역방향 프라이머 각각 1 μL, 프 로브 0.5 μL, 50XROX dye 0.4 μL, 2X Maxter mix 10 μL 그리고 최종 용량이 20 μL가 되도록 증류수를 첨가하였다. 본 연구에서 개발된 프라이머 및 프로브의 염기서열 및 검 출을 위한 최적의 반응 조건은 Table 1에 나타내었다.
|
밀 유전자 증폭산물 154 bp에 해당하는 염기서열의 합성 및 2,002 bp의 pHBA vector에 삽입한 총 2,156 bp의 유전 자 표준 DNA는 바이오니아(Bioneer, Korea)에 의뢰하여 합성하였다(Fig. 2).
결과 및 고찰
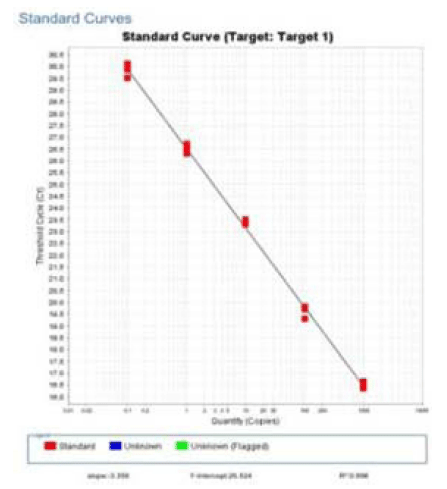
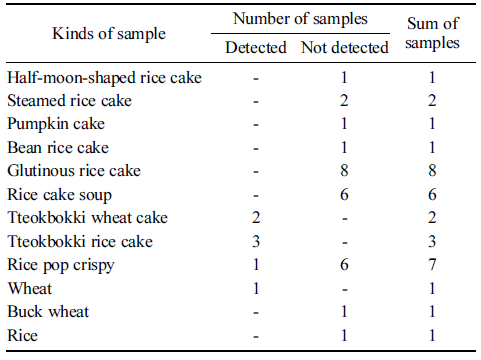
밀(Triticum aestivum, access number: FJ609737, FM998884, KC589709, KF482082)과 쌀(Oryza sative, access number: KM036285)의 ITS2 (internal transcribed spacer)부 위 염기서열을 확보하여 상이한 부위를 이용, 밀 특이 프 라이머 및 프로브를 제작하고 증폭 산물 154 bp의 염기서 열은 미국 국립보건원 유전자은행에 등록되어있는 염기서 열과 비교하여(NCBI nblast)밀 특이 프라이머로서의 가능 성을 확인하였다. 밀 유전자 표준품을 10 ng부터 10배씩 1 pg 농도까지 단계희석하고 본 연구에서 제작한 프라이머를 사용하여 real-time PCR 3회 반복한 결과에서 standard curve 기울기(slope)는 -3.356, 상관 계수(R2)는 0.998, 증폭 효율은 98.589%를 나타내었다(Fig. 3). 쌀 가공식품에서의 밀 검출 효율을 알아보기 위해 쌀가루에 통밀가루를 50%, 10%, 1%, 0.1%, 0.01% 그리고 0.001%가 되도록 각각 혼 합한 시료를 만들어 실험한 결과 밀이 0.01%까지 혼합된 시료까지 검출이 가능함을 알 수 있었다. 밀과 쌀 가공식 품 31개와 원재료 3개, 모두 34개 종류의 시료에서 밀 특 이 프라이머 및 프로브를 이용한 real-time PCR 분석결과 는 Table 2와 같다. 밀, 메밀 그리고 쌀 각각 1개의 원재 료 시료 중 밀 시료에서만 밀특이 유전자가 검출되어 밀에 대한 특이성이 확인되었다. 떡볶이떡을 제외한 쌀떡이라고 표기된 떡 19개의 시료(모시떡 1, 시루떡 2, 호박떡 1, 콩 떡 1, 찹쌀떡 2, 인절미 2, 모찌떡 2, 증편 2, 떡국떡 6)에 서는 모두 밀 특이 유전자가 검출되지 않았다. 쌀과자는 7 개의 시료(뻥튀기 2, 누룽지 2, 쌀과자 3) 중 밀 40%가 포 함되어있다고 성분 표시된 1개의 시료(뻥튀기)에서만 밀 특이 유전자가 검출 되었다. 떡볶이 떡은 5개의 시료 모두 에서 밀 특이 유전자가 검출되었다. 제품명이 밀 떡볶이라 고 표기된 2개의 제품과 쌀 떡볶이로 표기된 3개의 제품 이었다. 제품명이 쌀 떡볶이로 표기된 3개의 제품은 함량 은 표기되어있지 않았지만 성분표에서 소맥분(밀)이 포함 되어있다는 것을 확인할 수 있었다. 재래시장의 방앗간 등 에서 판매되는 떡과 같은 가공식품은 제품에 성분표시가 되어있지 않기 때문에 원재료의 성분에 대한 정보를 전적 으로 판매자의 설명에 의존하여 구매할 수밖에 없는 실정 이다. 또한 쌀 떡볶이의 경우 밀에 대한 성분표시가 있더 라도 쌀 떡볶이라는 제품명으로 판매되고 있는 제품들이 있어 밀에 대한 알레르기를 가진 소비자가 쌀 떡볶이로 오 인하고 섭취할 가능성도 배제할 수 없다. 밀은 세계적으로 전체 단백질 소비량의 약 70% 정도를 차지하는 식품으로 서 국내에서도 2014년 1인당 밀 소비량이 33.6 kg으로 1975년 이후 꾸준한 섭취량을 보이고 있으며 우유, 계란, 대두, 땅콩 등과 함께 알레르기를 일으키는 주요 원인 중 하나로 알레르기 유발물질 표시대상 21개 품목에 속해있 다. 비의도적이라도 밀이 혼입된 가공식품은 특히 어린 영 유아에게 급작스럽게 발생하는 심한 알레르기 과민반응으 로 정의되는 아나필락시스를 일으킬 수도 있기 때문에 (Yoon et al., 2016) 가짜 식품이나 비의도적인 원재료의 혼 입 여부를 확인 할 수 있는 방법 개발이 중요하다. 따라서 본 연구에서 개발된 쌀과 밀의 ITS2 (internal transcribed spacer)부위 염기서열의 종간 다양성을 이용한 real-time PCR 방법은 가짜 식품이나 비의도적인 원재료 혼입 등을 판별해내고 불량식품 근절과 소비자에게 정확한 정보를 제 공하는데 기여할 수 있을 것으로 생각 된다.
요 약
밀(Triticum aestivum)의 ITS2 (Internal transcribed spacer) 부위 염기서열을 이용하여 밀 특이프라이머 및 프로브를 제작하고 real-time PCR 조건을 정립하였다. 검량선은 기 울기 -3.356, 상관계수 0.998, 증폭효율 98.589%를 나타내 었다. 쌀가루에 통밀가루를 50%, 10%, 1%, 0.1%, 0.01% 그리고 0.001%가 되도록 각각 혼합한 시료를 만들어 실험 한 결과 밀이 0.01%까지 혼합된 시료까지 검출이 가능했 다. 쌀과 밀 그리고 그 가공식품 34건을 대상으로 밀 검출 가능성을 확인한 결과 밀이 포함된 7건의 가공식품 모두 검출이 가능했다. 본연구에서 개발한 밀특이 프라이머 및 프로브를 이용한 real-time PCR 방법은 사용원료의 부정확 한 표시나 제조 과정 중 밀의 비의도적인 혼입 여부 등 식품원료의 진위 판별에 활용이 가능할 것이다.