서 론
전 세계적으로 무슬림 인구는 2010년에 약 16억명, 2030년에 22억명으로 증가 할 것으로 예상되며, 전체 할랄 (Halal) 시장의 규모는 2조 달러에서 3조 1000억 달러까지 예측되고 있다(Mukherjee, 2014). 매년 꾸준한 성장률을 보 이는 할랄시장은 이미 포화상태에 접어든 현 시장에 등장 한 새로운 개척지로 식품, 의약품, 화장품 그리고 관광업 등의 다양한 분야에서 활발한 진출이 이루어지고 있다 (Kong, 2012). 할랄의 의미는 ‘이슬람율법에서 허용된 것’ 이고, 하람(Haram)은 할랄과 반대되는 개념으로 ‘금지된’이 라는 의미를 가지고 있다. 일반적으로 돼지, 인체 성분, 개, 동물의 피, 알코올 등은 haram으로 분류되며 식물은 대부 분 halal으로 인정되나 GMO 식물은 halal로 인정되지 않 는다(Kim, 2015). 또한 효소나 소재를 이용할 때에도 유래 를 따져야 하는데 미생물 유래일 때에는 사용된 배지에 하 람 성분이 혼입된 것은 사용하면 안 된다.
할랄 시장에 진입하기 위해서는 제품에 대한 할랄인증을 받아야 하는데, 세계적으로 150-200여 개의 할랄인증 기구 가 존재하며 각 기관의 승인을 받은 제품에 한해 인증서를 발행한다(Han & Lee, 2016). 말레이시아의 JAKIM, 인도 네시아의 MUI 인증이 대표적이며 할랄제품으로 인증 받기 위해서는 제품제조에 사용되는 모든 재료, 제조시설, 공정, 작업자 교육 등에 대한 서류심사와 현지실사 등과 같은 엄 격한 심사과정을 거쳐야 한다(Seo, 2014).
할랄 인증을 위해서 제품의 종류에 따라 물질 특성을 반 영한 적절한 분석법이 필요하다. 현재 이용되고 있는 분석 법은 DNA나 단백질 분석을 기반으로 한 것들로, 식품의 경우 이화학적 분석법이 주로 이용된다. 단백질에 기초한 방법에는 전자코를 이용한 돼지 휘발성화합물 검출(Che Man et al., 2005a;Nurjuliana et al., 2011;Park et al., 2017), fourier transform infrared (FTIR) spectroscopy를 이 용한 돼지기름 위화물검출(Che Man et al., 2005b), high performance liquid chromatogrphy (HPLC)를 이용한 가공 식품에서의 돼지 성분검출(Bargen et al., 2014;Rashood et al., 1995), nuclear magnetic resonance (NMR) spectroscopy 를 이용한 식품에서의 돼지유래 지방산 정량 (Siciliano et al., 2013) 등의 방법이 이용되고 있다. 하지만 제품 내 다 양한 성분이 혼합되어 있고 열처리, 물리적, 화학적 처리와 같은 다양한 가공공정을 거치기 때문에 그에 따라 검출한 계가 달라 질 수 있다.
한편 DNA는 보다 안정적인 물질이며 어느 조직에서나 추출될 수 있기 때문에, 열처리된 가공 제품의 분석에서 더 낮은 검출한계를 보였다(Nakyinsige et al., 2012). 따라 서 Species-specific DNA를 검출하는 Polymerase Chain Reaction (PCR)을 이용한 분석법은 제품 중 미량의 DNA 를 증폭하여 해당 종에서 유래한 성분들의 존재를 확인할 수 있는 유용한 대안이 될 수 있다(Lockley & Bardsley, 2000). GMO 작물의 경우는 개발 과정에 외래 유전자 단 편이 삽입되므로 이 과정에 흔히 쓰이는 35S promoter와 nopaline synthase (NOS) terminator의 존재 여부로 GMO 를 판단할 수 있다(Barbau-Piednoir et al, 2010; Waiblinger et al., 2008). 식품공전에 GMO 콩과 옥수수의 정성시험법 으로 35S promoter와 NOS terminator를 이용한 PCR법이 사용되고 있다.
최근 식품에서는 PCR을 이용한 할랄분석이 많이 보고되 고 있으나(Che Man et al., 2007;Alaraidh, 2008), 화장품 에서는 할랄분석이 거의 알려져 있지 않다. 본 연구에서는 화장품으로 널리 사용되는 액상 마스크팩과 크림에 기능성 향상을 위한 식물 추출물(당근, 느타리버섯, 솔잎)을 첨가 하고 이들을 대상으로 중간 단계인 추출물과 추출물이 첨 가된 최종 제품이 할랄화장품으로 적합한지 확인하기 위하 여, 주요 하람 성분(돼지, 개, 사람, GMO)들을 신속 검출 할 수 있는 PCR 시스템을 구성하였다.
재료 및 방법
본 실험에 사용한 liquid-type mask pack 화장품, cream 제형의 화장품 그리고 식물추출물은 ㈜Chemland에서 제조 하였다. 추출물은 화장품에 기능성을 부가할 수 있는 유용 amino acid와 peptide의 함량을 증가시키기 위해 파파야의 단백질 분해효소 papain과 파인애플의 단백질 분해효소 bromaline을 사용하였고 추출물은 마지막 단계에서 효소불 활성화를 위해 열처리되었다.
각 식물의 추출물 샘플은 당근, 느타리버섯, 솔잎 각각의 효소 미처리군, papain 처리군 및 bromelain 처리군으로 총 9종을 사용하였다. 각 추출물은 당근, 느타리버섯, 솔잎 원 물을 각각 1% (w/v) 함유하며, 화장품에는 50%의 추출물 이 사용되었다. 화장품은 액상 마스크팩 제형과 크림 제형 각각에 대해 추출물 무첨가군 1종, 효소 무처리 추출물 첨 가군 3종, papain 처리 추출물 첨가군 3종 및 bromelain 처리 추출물 첨가군 3종으로 총 20종을 사용하였다.
돼지고기와 개고기는 시중 마트에서 구입하였고, GMO 대두(Roundup Ready Soya Bean Powder)는 Sigma 제품 을 사용하였으며, 인체 구강상피세포는 실험자의 샘플을 직접 채취하여 사용하였다. 구강상피세포 채취는 먼저 생 리식염수 20 mL을 입안에 넣고 약 2분간 헹군 후 13,000 rpm에서 10분간 원심분리하여 그 pellet을 샘플로 사용하 였다.
각 샘플로부터 chromosomal DNA를 추출하기 위해 Power PrepTM DNA extraction kit (Kogenbiotech, Seoul, Korea)를 사용하였다. DNA 추출은 제조사의 방법을 따랐 다. 또한 3종의 식물추출물, 2종의 마스크팩과 크림 그리 고 추출물이 첨가된 화장품에서 DNA를 추출하는데 역시 Power PrepTM DNA extraction kit (Kogenbiotech.)를 사용 하였다.
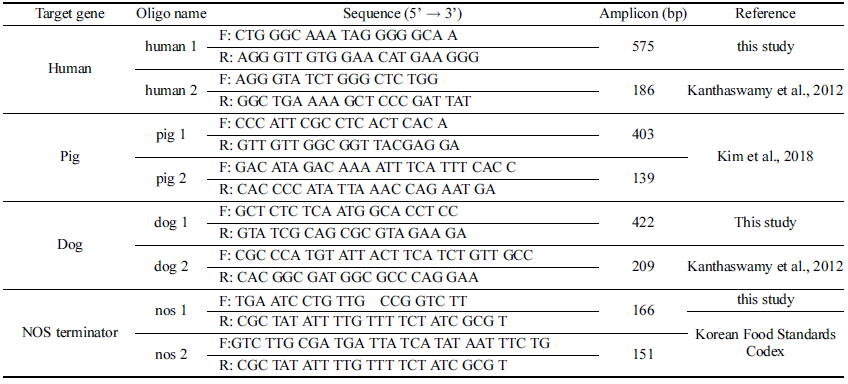
돼지, 인체, 개 및 GMO DNA를 증폭시키기 위한 primer 들의 염기서열을 Table 1에 제시하였다. PCR에 사용된 primers는 ㈜코스모진텍(Seoul, Korea)에서 합성하여 사용 하였다. 분석하려는 target DNA (인체, 186 bp (base pair); 돼지, 139 bp; 개, 209 bp; NOS terminator, 151 bp)의 검출 한계를 copy number로 측정하기 위해 chromosomal DNA 로부터 target DNA 영역을 포함하는 template DNA(인체, 575 bp; 돼지, 403 bp; 개, 422 bp; NOS terminator, 166 bp) 를 PCR로 각각 증폭하여 사용하였다.
Thermocycler로 StepOnePlus real-time PCR System (Applied Biosystems, Foster City, CA, USA)을 이용해 PCR을 진행하였다. 각 샘플의 chromosomal DNA로부터 template DNA 증폭과 template DNA로부터 최종 target DNA의 증폭에 사용한 PCR 조건은 다음과 같다. PCR 반 응액의 조성은 chromosomal DNA 또는 template DNA 1 μL (microliter)와 10 pmol (picomole)/μL의 primer set 1 μL, 10x Taq reaction buffer 2.5 μL, dNTP 0.5 μL (containing 10 mM of each dNTP), DNA polymerase (Taq DNA polymerase, SolGent Co., Daejeon, Korea) 0.125 μL 를 첨가하고 멸균 증류수로 총량이 25 μL가 되도록 하였 다. Thermal cycle 조건은 95°C에서 5분간 가열 후 95°C 30초, 60°C 30초, 72°C 30초를 1 cycle로 하여 총 40 cycle 을 수행 후 72°C에서 3분간 더 반응시켰다. PCR 산물은 1.5% agarose gel에서 전기영동하여 확인하였다.
PCR 검출방법의 구성을 위해 사용한 primer set (Table 1)들에 의해 증폭되는 DNA 단편의 크기를 확인하기 위해 4 종의 chromosomal DNA로부터 각 primer set (human 1, pig 1, dog 1 and nos 1)을 이용하여 template DNA를 증 폭하였다. 증폭된 인체, 돼지, 개 및 GMO 대두의 template DNA 크기는 각각 575, 403, 422 및 166 bp 이었다 (lane 1, 4, 7 and 10 in Fig. 1). 또한 각 template DNA를 정제하여 내측의 target DNA를 증폭시키는 primer set (human 2, pig 2, dog 2 and nos 2)로 증폭 시켜, 인체, 돼지, 개 및 GMO 대두의 target DNA 크기가 각각 186, 139, 209 및 151 bp임을 확인하였다(lane 2, 5, 8 and 11 in Fig. 1).

PCR로 증폭한 template DNA를 agarose gel에서 회수하 기 위해 PCR clean-up gel extraction kit (Macherey-Nagel, Durenm, Germany)를 사용하였다. Template DNA 농도 측 정을 위해 Spectrophotometer (Model 6705 UV, Jenway Co, Staffordshire, UK)를 사용하여 DNA copy number를 계산하였다(Whelan et al., 2003).
결과 및 고찰
PCR로 특정 생물 종의 DNA를 증폭시킬 때 template DNA로 chromosomal DNA 또는 target region이 포함된 작은 사이즈의 DNA 단편을 사용한다(Lampel et al., 2000;Whyte et al., 2002). 할랄제품 생산 시 유입될 수 있는 대 표적인 하람 성분으로 사람, 돼지, 개, NOS terminator를 이용한 GMO를 설정하였다.
각각의 PCR 반응에서 정확한 크기의 DNA 단편 이외에 다른 DNA 단편이 증폭되지 않아 비특이적 반응은 일어나 지 않았음을 알 수 있었다.
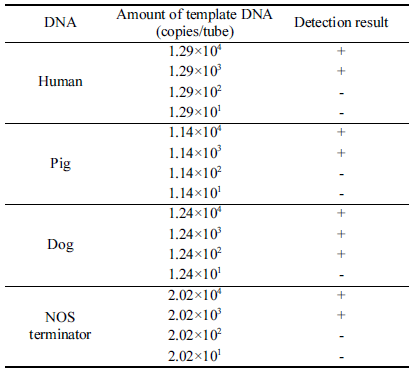
본 실험에서는 정확한 검출한계 측정을 위해 target DNA region이 포함된 template DNA 단편을 PCR로 증폭한 다 음(Fig. 1) copy number를 계산하여 사용하였다. PCR로 증폭한 각 template DNA를 agarose gel에서 정제한 후 spectrophotometer를 이용하여 각각의 농도를 결정하였다. 측 정한 각 DNA 농도는 사람 8.1 ng/μL, 돼지 50.5 ng/μL, 개 57.5 ng/μL, GMO 콩 36.9 ng/μL이었다. 각 template DNA의 농도와 amplicon size (bp)를 이용하여 template DNA의 copy number를 계산한 결과, 사람 DNA는 1.29×1010, 돼지 DNA는 1.14×1011, 개 DNA는 1.24×1011, GMO 콩 DNA는 2.02×1011 (copies/μL)이었다.
본 실험에서 구성한 PCR 방법의 검출한계를 조사하기 위해 4종의 template DNA를 여러 농도(약 101-104 copy number/tube)로 희석하여 PCR을 수행하였다. PCR 방법의 검출한계는 사람 DNA는 1.29×103, 돼지 DNA는 1.14×103, 개 DNA는 1.24×102, GMO 콩 DNA는 2.02×103 (copies/ μL)이었다(Table 2).
PCR을 이용한 DNA 검출 반응에서 흔히 문제가 되는 것이 측정하고자 하는 샘플 내에 존재할 수 있는 여러 PCR 저해제에 의한 위음성 반응이다(Demeke & Jenkins, 2010). 식품의 경우 polysaccharides, tannic acid, collagen, myoglobin, ethanol, 금속이온 등이 PCR을 저해하는 것으 로 알려져 있다(Rossen et al., 1992;Lübeck et al., 2003;Schrader et al., 2012). 화장품의 경우는 조성이 식품에 비 해 비교적 단순하나(Ku et al., 2013) PCR을 저해하는 성 분에 대해서는 잘 알려져 있지 않다. 또한 식물 성분 중에 pectin, polyphenols, polysaccharides 등이 DNA를 손상시키 거나, primer 또는 polymerase와의 결합을 방해하여 PCR 반응을 저해할 수 있다고 알려져 있다(Schrader et al., 2012).
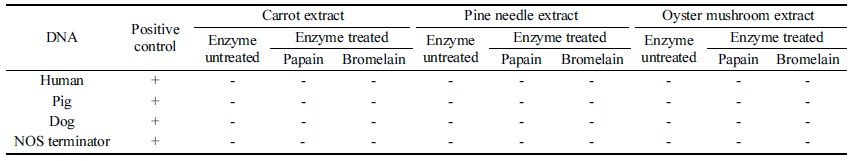
식물 추출물이나 화장품 성분이 PCR을 저해하는지 확인 하기 위해 당근, 솔잎 및 느타리버섯 추출물과 액상 마스 크팩 및 크림에 인위적으로 돼지 DNA를 여러 농도로 첨 가한 후 각 DNA를 추출하여 PCR을 수행하였다. 당근, 솔 잎 및 느타리버섯 추출물 6종 그리고 화장품 2종에서 돼 지 DNA의 검출한계는 1.14×103 (copies/μL) 이었다(Table 3). 이 결과는 PCR 방법의 돼지 DNA 검출한계를 측정한 Table 2의 결과와 동일하여 본 연구에 사용한 식물 추출물 및 화장품 성분들은 PCR을 저해하지 않는 것으로 판단하 였다.

|
세계적으로 할랄시장의 규모가 증가하고 글로벌화함에 따라 유통되는 할랄 제품에 대한 관리가 점점 어려워지고 있다. 할랄인증 과정은 주로 제품제조에 사용되는 모든 재 료의 출처에 대한 서류심사와 현지 제조시설에 대한 출장 감사 등으로 이루어진다(An, 2015). 그러나 서류심사나 출 장감사로는 제품의 제조과정 중 의도적 또는 비의도적으로 혼입된 하람 성분을 확인하기에는 한계가 있다. 따라서 제 조회사가 원부재료, 중간제품 및 최종 제품들을 대상으로 하람 성분들에 대한 지속적인 모니터링을 통해 할랄 제품 에 대한 신뢰성을 제고할 필요가 있다.
본 연구에서 분석한 대상이 모두 할랄에 속하는 식물추 출물이고 사용한 효소도 식물에서 유래하였으나, 추출물과 마스크 팩과 크림 성분들을 혼합하고 제조하는 과정 및 시 설에서 하람 성분이 오염될 가능성을 배제할 수 없다. 따 라서 당근, 느타리버섯, 솔잎 추출물 3종과 이 추출물에 papain과 bromelain을 각각 처리한 6종의 추출물을 포함한 총 9종의 추출물 샘플 그리고 상기 추출물이 포함 또는 포함되지 않은 마스크 팩과 크림 화장품 총 20종을 대상으 로 4종의 하람성분을 검출할 수 있는 PCR을 수행하였다.
먼저 추출물에 대한 PCR 결과, 양성 대조군을 제외한 9종의 추출물은 4가지 하람 성분에 대해 모두 음성반응을 나타내었다(Table 4). 효소를 처리하지 않은 당근, 느타리버 섯 및 솔잎 추출물뿐만 아니라 이들에 papain과 bromelain 을 각각 처리한 효소처리군도 모두 음성이어서 추출물 및 두 효소의 사용은 문제가 없는 것으로 보인다.
|
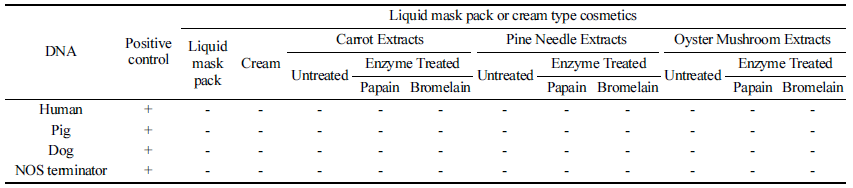
또한 추출물과 효소 처리한 추출물을 마스크팩과 크림에 각각 혼합한 제품들에 대해 PCR을 수행한 결과, 양성 대 조군을 제외한 20종의 화장품은 4가지 하람성분에 대해 모두 음성반응을 나타내었다(Table 5). 마스크팩과 크림에 첨가한 추출물 및 효소 처리한 추출물에서 모두 음성 반응 을 나타내 화장품 원료, 중간제품 및 최종제품에 이르기까 지 모두 하람 성분은 없는 것으로 추정이 가능하다.
|
비록 PCR 방법이 극미량의 DNA도 검출할 수 있는 매 우 민감한 방법이기는 하나 PCR 검출한계 이하로 존재하 는 성분들이 있을 가능성을 완전히 배제할 수는 없다. 또한 GMO의 경우, 외래유전자 도입에 35S promoter와 NOS terminator를 쓰지 않은 시스템은 본 연구에서 제안한 PCR 방법으로는 검출할 수 없다. 이러한 문제들은 모든 분석법 들이 지닌 공통적인 문제이며 향후 더욱 다양하고 민감한 방법의 개발이 필요하다. 본 실험에서 개발한 PCR 방법은 할랄 제품의 제조과정 중 원부재료, 중간산물 및 최종제품 에 이르기까지 하람 성분의 신속한 모니터링에 활용이 가 능할 것으로 보인다.
요 약
최근 국내의 식품 및 화장품 분야에서 해외 할랄시장 진 출을 위해 할랄 인증에 대한 관심이 높아지고 있다. 할랄 인증을 받으려면 해당 제품에 돼지, 개, 사람, GMO 등과 같은 haram 성분들이 포함되어서는 안 된다. 본 연구에서 는 papain과 bromelain을 처리한 식물추출물(당근, 느타리 버섯, 솔잎)이 첨가된 화장품(cream과 mask pack)이 할랄 인증에 적합한지를 확인하기 위해 이들 추출물과 최종제품 에서 haram 성분의 존재 여부를 PCR을 이용하여 분석하 였다. 사람, 돼지, 개 및 GMO 대두의 template DNA를 특 이적으로 검출하는 PCR 방법의 검출한계는 각각 1.29×103, 1.14×103, 1.24×102 및 2.02×103 copies/tube 이었다. PCR은 식물 추출물이나 화장품 성분에 의해 저해 받지 않았다. 식물추출물과 이들이 첨가된 cream과 mask pack에 대해 PCR을 수행한 결과 모두 음성이었다. 본 PCR 방법은 식 품이나 화장품의 제조과정 중 원재료나 최종제품에서 haram 성분의 존재를 신속하게 확인하는 데 활용이 가능 할 것으로 보인다.